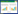中国科学院微生物研究所、中国微生物学会主办
文章信息
- 叶相相, 毕永江, 冉琼, 张晓辉, 王邦俊
- YE Xiangxiang, BI Yongjiang, RAN Qiong, ZHANG Xiaohui, WANG Bangjun
- 植物WRKY转录因子在应对盐胁迫中的作用研究进展
- The role of plant WRKY transcription factors against salt stress: a review
- 生物工程学报, 2023, 39(7): 2600-2611
- Chinese Journal of Biotechnology, 2023, 39(7): 2600-2611
- 10.13345/j.cjb.220652
-
文章历史
- Received: August 17, 2022
- Accepted: December 14, 2022
- Published: December 16, 2022
2. 西南大学生命科学学院 重庆市资源植物保护与种质创新重点实验室, 重庆 400715;
3. 重庆理工大学管理学院, 重庆 400054
2. Chongqing Key Laboratory of Plant Resource Conservation and Germplasm Innovation, College of Life Sciences, Southwest University, Chongqing 400715, China;
3. School of Management, Chongqing University of Technology, Chongqing 400054, China
适宜的土壤是作物养分有效吸收和生长的基础。然而,随着自然环境多变、海平面上升以及人类不合理生产活动的增多,导致地下水中可溶性盐大量积累,盐胁迫对作物生长发育和产量的影响日益加重。在此环境压力下,植物进化出了不同的胁迫反应和耐性调节途径,例如激素信号途径[1]、盐胁迫(salt overly sensitive, SOS)途径[2]、染色质修饰[3]和转录调节[4]。转录因子是调节基因转录的蛋白质,是环境胁迫下信号转导和基因表达调控过程中的重要调节因子。转录因子可以与顺式作用元件结合,通过抑制或增强靶基因的转录来调节其转录效率[5]。
WRKY家族是高等植物和藻类的一个独特的转录因子超家族,在许多生命过程中发挥着重要作用,特别是在应对生物和非生物胁迫方面[6],目前已有广泛的研究证明WRKY转录因子在胁迫诱导的多条途径中发挥作用[7]。20世纪90年代,研究人员从甘薯中克隆出第1个WRKY蛋白SPF1[8],后来越来越多的WRKY蛋白被从各种植物中鉴定出来。到目前为止,已在不同的物种中发现了WRKY转录因子家族,其中,拟南芥(Arabidopsis thaliana)中有74个成员[9],水稻(Oryza sativa)中有100多个成员[10],大豆(Glycine max)中有197个成员[11],大麦(Hordeum vulgare)中有45个成员[12],黄瓜(Cucumis sativus)中有55个成员[13],番茄(Solanum lycopersicum)中有81个成员[14]。在遭受环境胁迫(盐、干旱等)的植物中,WRKY家族成员在不同的胁迫反应中起着至关重要作用。也有越来越多的研究表明,WRKY转录因子还参与了植物多种其他特异性反应,如衰老[15]、机械损伤[16]、热休克[17],以及其他许多反应过程[6]。因此,WRKY转录因子受到了广泛的关注。本文综述了WRKY转录因子家族的结构特征和调控机制,以及其在盐胁迫中的最新研究进展。
1 WRKY转录因子的结构WRKY蛋白由两部分构成,N端约60个氨基酸的DNA结合域和C端锌指结构[18-19]。所有已知的WRKY蛋白都有1个或2个WRKY结合域。根据WRKY结合域的数量和类锌指基序的特征,它们可以分为3组:第Ⅰ组包含两个WRKY结合域,并含有C2-H2基序(C-X4-5-C-X22-23-H-X1-H);第Ⅱ组包含一个WRKY结合域,并含有C2-H2基序;第Ⅲ组包含一个WRKY结合域,以及一个不同的类锌指基序C2-H-C (C-X7-C-X23-H-X1-C) (图 1)[20]。虽然它们的DNA结合域高度保守,但WRKY蛋白的整体结构却高度不同。其中,第Ⅱ组可通常分为5个亚组(Ⅱ a、Ⅱ b、Ⅱ c、Ⅱ d和Ⅱ e)[6]。基于系统发育数据的分析表明,高等植物中的WRKY家族被更准确地划分为组Ⅰ、Ⅱ a+Ⅱ b、Ⅱ c、Ⅱ d+Ⅱ e和Ⅲ[21-22],而组Ⅱ的WRKY并不单独分为一类。除WRKY结构域之外,一些WRKY蛋白还含有其他结构域,如谷氨酸富集区、脯氨酸富集区和亮氨酸拉链结构等[23]。
转录因子的表达模式一般分为组织型表达和诱导型表达两种,WRKY转录因子是在各种胁迫下可诱导表达的转录因子。目前已知WRKY的表达受多种环境(如病原体、盐碱、温度、干旱和机械胁迫)的影响,其表达具有快速、即时和组织特异性等特点[24]。
WRKY转录因子家族成员有不同的调控机制,主要包括转录的激活、抑制和去抑制。WRKY蛋白可以与W-box元件结合,激活或抑制下游靶基因的转录,同时,也会受到上游更高级调节蛋白质(如磷酸化酶)的调控。此外,它们还可以与其他作用元件结合形成蛋白质复合物,从而增强转录活性[19]。WRKY转录因子可以通过结合下游基因启动子区域保守的W-box顺式作用元件,激活下游基因的表达[25]。由于大多数WRKY转录因子的自身启动子中都含有丰富的W-box元件,使得这些WRKY转录因子可以与它们自身启动子结合或与其他WRKY转录因子结合,来实现自我调节或交叉调节[26]。此外,有些WRKY转录因子兼具激活和抑制转录的功能[15]。除保守的W-box以外,WRKY蛋白还可以选择与不一样的顺式作用元件结合,参与到更复杂的下游靶基因调控中。总之,WRKY转录因子与目标基因之间存在着多种结合方式。不同的结合方式和结合位点使WRKY转录因子在植物转录调控中具有多种功能。
丝裂原活化蛋白激酶(mitogen-activated protein kinase, MAPK)调节许多植物信号反应,如植物免疫、环境胁迫响应和正常生长发育等,其介导的信号通路的激活是植物反应的一个重要过程[27]。一些研究表明,MAPK参与控制某些WRKY转录因子的活性[28]。例如,在烟草(Nicotiana tabacum)中,创伤诱导蛋白激酶(wound induced protein kinase, WIPK)和水杨酸诱导蛋白激酶(salicylic acid induced protein kinase, SIPK)都位于NtWRKY1和NtWRKY3的上游[29]。
3 WRKY转录因子参与盐胁迫响应盐胁迫是环境中存在较为广泛的非生物胁迫,严重影响作物产量。WRKY转录因子在调节植物对盐胁迫的反应中起着至关重要的作用。
植物细胞内一般通过活性氧(reactive oxygen species, ROS)分子和Ca2+离子进行信号转导,当植物遭受到盐胁迫时,植物会处在一种应激反应状态,此时相应的信号被激活并传送到细胞内部,随后蛋白激酶如MPK被激活以调节相关转录因子的活性[30-31]。
在植物中,脱落酸(abscisic acid, ABA)是生物和非生物胁迫响应的关键调节因子,在胁迫下ABA的生物合成和积累也会增强。非生物胁迫信号通过ABA依赖和非ABA依赖两种途径起作用,不同的WRKY转录因子在ABA信号通路中协同或独立发挥作用[32]。已经有大量研究报道了不同的WRKY基因在盐胁迫下导致了不同植物的各种表型(表 1)。
| No. | Gene | Species | Function | References |
| 1 | AtWRKY28 | Arabidopsis thaliana | Tolerant to salt | [33] |
| 2 | AtWRKY33 | Arabidopsis thaliana | Tolerant to salt | [34] |
| 3 | AtWRKY46 | Arabidopsis thaliana | Tolerant to salt | [35] |
| 4 | AhWRKY75 | Arachis hypogaea Linn. | Tolerant to salt | [36] |
| 5 | BcWRKY1 | Boea crassifolia | Sensitive to salt | [37] |
| 6 | ClWRKY20 | Citrullus lanatus | Tolerant to salt and cold | [38] |
| 7 | CmWRKY17 | Chrysanthemum morifolium | Sensitive to salt | [39] |
| 8 | GhWRKY34 | Gossypium hirsutum | Tolerant to salt | [40] |
| 9 | GmWRKY13 | Glycine max | Sensitive to salt | [41] |
| 10 | GmWRKY54 | Glycine max | Tolerant to salt and drought | [41] |
| 11 | IbWRKY47 | Ipomoea batatas | Tolerant to salt | [42] |
| 12 | MdWRKY30 | Malus×domestica Borkh. | Tolerant to salt | [43] |
| 13 | MdWRKY55 | Malus×domestica Borkh. | Tolerant to salt | [44] |
| 14 | NbWRKY79 | Nicotiana benthamiana | Tolerant to salt | [45] |
| 15 | OsWRKY50 | Oryza sativa | Tolerant to salt | [46] |
| 16 | PbWRKY40 | Pyrus betulaefolia | Tolerant to salt and organic acid accumulation | [47] |
| 17 | PcWRKY11 | Polygonum cuspidatum | Tolerant to salt | [48] |
| 18 | PeWRKY31 | Populus x euramericana | Tolerant to salt | [49] |
| 19 | SbWRKY50 | Sorghum bicolor | Sensitive to salt | [50] |
| 20 | ZmWRKY17 | Zea mays | Sensitive to salt | [51] |
| 21 | ZmWRKY86 | Zea mays | Sensitive to salt | [52] |
| 22 | ZmWRKY104 | Zea mays | Tolerant to salt | [53] |
WRKY转录因子与其他基因之间协作形成调控网络来响应盐胁迫,植物在遭受盐胁迫时体内的活性氧平衡、离子平衡以及渗透平衡会被破坏,使得植物的内稳态紊乱,从而影响植物的生长发育。WRKY转录因子的调控网络可以对植物内稳态进行调节,使植物适应环境。WRKY转录因子家族对植物响应盐胁迫过程中ABA信号通路的调控机制较为复杂,其不仅可以直接调控ABA信号通路相关基因的表达,还可以依赖于茉莉酸信号通路与ABA信号通路之间的协同作用调控ABA信号通路[32]。
3.1 WRKY转录因子提高植物的耐盐性已有研究表明,在拟南芥中AtWRKY28和AtbHLH17共表达的转基因株系表现出对氯化钠、甘露醇和氧化胁迫的耐受性增强[33]。AtWRKY33介导了AtCYP94B1的调节增强了根部内皮细胞的亚皮素沉积,从而赋予拟南芥植株耐盐性;此外,atwrky33突变体表现出亚皮素减少和盐敏感的表型,在此背景下表达35S: : AtCYP94B1,这些表型得到了恢复[34]。对拟南芥ABA相关突变体的分析表明,AtWRKY46的表达被ABA信号下调,而被渗透/盐胁迫诱导的与ABA无关的信号上调。研究结果表明,AtWRKY46通过调节ABA信号和生长素平衡,对渗透/盐胁迫依赖的侧根抑制起到了前馈抑制作用[35]。水稻的OsWRKY50在细胞核中作为转录抑制因子发挥作用。OsWRKY50的转录在盐胁迫条件下被抑制,但在脱落酸ABA处理后被激活。与野生型相比,OsWRKY50的过表达植物显示了对盐胁迫的耐受性。在盐胁迫下,过表达OsWRKY50植株中OsLEA3、OsRAB21、OsHKT1;5和OsP5CS1的表达显著高于野生型和对照植物。OsWRKY50通过ABA非依赖性途径介导ABA依赖的种子萌发和幼苗生长,并增强盐胁迫耐受性[46]。ZmWRKY104定位于细胞核并显示出转录激活活性。ZmWRKY104在玉米(Zea mays)中的过表达通过正向调控ZmSOD4的表达,提高了玉米对盐胁迫的耐受性,缓解了盐引起的活性氧的积累、丙二醛(malondialdehyde, MDA)含量和电解质渗漏百分比的增加,从而提高了植株的耐盐性[53]。花生(Arachis hypogaea Linn.)的AhWRKY75通过提高ROS清除系统的效率和胁迫处理下的光合作用来赋予转基因花生株系耐盐性[36]。过表达杨树(Populus x euramericana)的PeWRKY31提高了转基因烟草的耐盐性,转录组测序和KEGG富集分析显示,与谷胱甘肽代谢、植物激素信号转导和MAPK信号通路相关的基因表达升高[49]。大豆的GmWRKY54通过调控DREB2A和STZ/Zat10而赋予植株耐盐性[41]。在甘薯(Ipomoea batatas)中,IbWRKY47基因正向调控非生物胁迫相关基因,从而提高甘薯的耐盐性[42]。有研究表明,陆地棉(Gossypium hirsutum)的GhWRKY34通过提高植株选择性吸收Na+和K+的能力以及在叶片和根部维持低Na+/K+的能力来增强植株对盐胁迫的耐受性。在NaCl处理下,与野生型相比,GhWRKY34转基因株系的几个指标(如种子发芽率、绿色子叶、根长和叶绿素含量)都明显高于野生型,并且转基因植物在盐胁迫下的胁迫相关基因的表达明显上调[40]。MdWRKY30在拟南芥中的过表达增强了苗期以及种子发芽和子叶变绿阶段的耐盐性和渗透性。MdWRKY30的过表达通过对胁迫相关基因的转录调控增强了转基因苹果(Malus×domestica Borkh.)胚胎对盐和渗透压的耐受性[43]。MdWRKY55在苹果幼苗中的过表达显著提高了耐盐性,MdWRKY55与MdNHX1启动子结合,从而增强其转录。MdNAC17-L通过形成蛋白复合物明显促进了MdWRKY55对下游MdNHX1表达的影响[44]。V型H+-ATP酶基因(VHA-B1)在植物胁迫反应中起着重要作用,研究以杜梨(Pyrus betulaefolia) PbVHA-B1的启动子为诱饵,获得了一个Ⅱ组WRKY基因PbWRKY40。PbWRKY40可以在启动子区域结合PbVHA-B1,从而提高植株的耐盐性和有机酸的积累。与野生型相比,转基因品系的发芽率、根系生长、叶绿素含量和总有机酸含量均高于野生型,而电解质渗漏、Na+/K+比率和MDA含量低于野生型。此外,敲除PbWRKY40基因在杜梨中也降低了PbVHA-B1的表达[47]。在本氏烟草(Nicotiana benthamiana)中,NbWRKY79的过表达明显增强了烟草植物对盐分的耐受性。NbWRKY79过表达株系对ABA介导的种子发芽和幼苗根系生长的敏感性提高,对氧化应激的耐受性增强。此外,还发现在盐胁迫条件下,ABA含量和ABA诱导基因的转录水平,包括NbAREB、NbDREB和NbNCED,都明显增加[45]。在虎杖(Polygonum cuspidatum)中,PcWRKY11与拟南芥AtWRKY11表现出高度的同源性,PcWRKY11的表达可以被NaCl、渗透压和紫外线诱导。在盐胁迫下,转基因植物比野生型显示出较低的MDA含量,较高的过氧化物酶(peroxidase, POD)、超氧化物歧化酶(super oxide dismutase, SOD)和过氧化氢酶(catalase, CAT)的酶活性。这些结果表明,PcWRKY11可能通过降低ROS水平和增加渗透物质的合成,在植物适应盐胁迫方面具有积极作用[48]。西瓜(Citrullus lanatus)的ClWRKY20在干旱、盐和外源植物激素(水杨酸、乙烯和脱落酸)的作用下上调。ClWRKY20的过表达增强了转基因拟南芥对盐和低温的耐受性。与野生型植物相比,转基因拟南芥中参与ABA信号通路、乙烯生物合成和乙烯信号通路、茉莉酸生物合成和茉莉酸信号通路的几个基因的转录水平明显增加。ClWRKY20正调控植物对盐和低温胁迫的耐受性,不仅通过ABA信号调节盐和低温反应,而且可能在几个信号通路中起整合作用[38]。
3.2 WRKY转录因子提高植物的盐敏感性ZmWRKY86是玉米中的一种核蛋白,在酵母中没有转录激活活性。ZmWRKY86能特异性地与W-box结合,与对照组相比,zmwrky86突变体对盐胁迫的耐受性增强,存活率、过氧化氢酶活性和K+含量较高,但在盐胁迫条件下MDA积累、相对电解质渗透水平和Na+含量较低。ZmWRKY86直接与两个盐胁迫相关基因Zm00001d020840和Zm00001d046813的启动子相互作用来参与盐胁迫调控[52]。玉米ZmWRKY17作为一个负调控因子,在拟南芥中过表达导致对盐胁迫的敏感性增加,并通过调节一些ABA和胁迫反应基因的表达而降低ABA敏感性。ZmWRKY17定位在细胞核中,在酵母中没有转录激活活性。通过对子叶绿化率、根系生长、相对电解质渗透率和MDA含量的生理学分析,发现明显降低了植物对盐胁迫的耐受性[51]。在高粱(Sorghum bicolor)中,与野生型相比,盐胁迫条件下,SbWRKY50过表达植株的发芽率、根长、生物量和钾离子含量明显下降,而过氧化氢、超氧阴离子和钠离子含量增加,且过表达植株中SOS1、HKT1以及与渗透压和氧化胁迫有关的基因的表达水平下降,降低了植物的耐盐性。SbWRKY50可以直接与SOS1和HKT1的上游启动子结合,通过控制离子稳态参与植物的盐胁迫反应[50]。菊花(Chrysanthemum morifolium)中的CmWRKY17可以被盐分诱导,作为转基因植物的转录抑制因子对盐胁迫进行负调控,提高植物对盐的敏感性。CmWRKY17在菊花和拟南芥中的过表达增加了对盐度胁迫的敏感性。在盐度胁迫下,转基因菊花的超氧化物歧化酶和过氧化物酶的活性以及叶片中脯氨酸的含量都明显低于野生型。与野生型相比,CmWRKY17转基因拟南芥中与胁迫有关的基因AtRD29、AtDREB2B、AtSOS1、AtSOS2、AtSOS3和AtNHX1的表达量减少[39]。在大豆中,GmWRKY13下游的靶基因是ABA途径中的负调控因子ABI1。与野生型植物相比,过表达GmWRKY13的转基因植物对盐和甘露醇胁迫的敏感性增加,但对脱落酸的敏感性降低。此外,GmWRKY13转基因植物表现出了侧根的增加。遗传转化实验表明,GmWRKY13的过表达显著增加了ABI1的表达,使植株对盐胁迫的耐受性降低[41]。厚叶旋蒴苣苔(Boea crassifolia)中的BcWRKY1受ABA的诱导表达,过表达BcWRKY1的转基因拟南芥表现出更强的盐敏感性和更高的H2O2含量。BcWRKY1能与BcZAT12的启动子结合,BcsAPX的表达被BcZAT12激活,从而抑制ROS的清除来使植株具有盐敏感性[37]。
4 结论与展望WRKY转录因子作为植物体内最大的转录因子家族之一,已被证实其不仅参与植物的生长发育过程,而且在植物受到盐胁迫时起着关键的作用。大量的WRKY转录因子已在模式植物中进行了功能鉴定,为其他植物提供了丰富的参考。
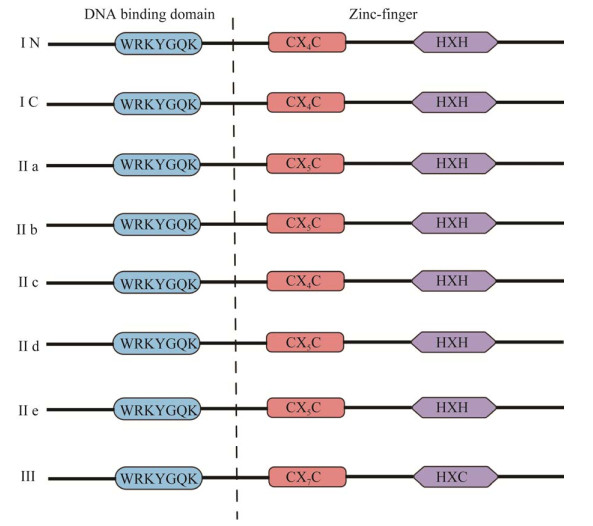
WRKY转录因子在植物对盐胁迫的响应过程中的精确调控和精细调节有助于建立复杂的信号网络[22]。WRKY转录因子可以直接调控转录,还可以由WRKY转录因子家族的其他成员间接调控[54],或者通过阻止其他WRKY转录因子的活动来进行调控[55]。然而,WRKY转录因子的转录调控过程是一个非常复杂的系统,许多来自不同家族的其他转录因子都可以与它们的启动子结合进行相互作用[56](图 2)。简单地说,为了进一步了解WRKY转录因子在盐胁迫下的作用,找出与它们相互作用的基因非常有必要,这些基因有助于协助WRKY转录因子控制下游靶基因的转录,对于阐明信号通路中的概念也很重要。酵母杂交、凝胶电泳迁移(electrophoretic mobility shift assay, EMSA)、染色质免疫沉淀(chromatin immunoprecipitation, ChIP)等技术可以帮助直接测定特定WRKY-DNA结合位点,这使我们能够识别WRKY调控基因[58]。
大豆是重要的粮食和经济作物。盐害作为主要非生物胁迫之一,对大豆的生长、性状、种子品质和种子数量造成负面影响,最终导致大豆产量的降低。大豆WRKY家族第Ⅰ、Ⅱ、Ш类基因均参与逆境调控,以第Ⅱ类研究最为广泛。其中GmWRKY21可以增强植物对低温的耐受性,GmWRKY13能够降低植物对盐胁迫的抗性,GmWRKY54、GmWRKY111和GmWRKY49等基因可以提高植物的耐盐性[59]。在我们目前的研究中,通过构建大豆cDNA文库并进行筛选,成功筛选出了盐敏感的GmWRKY56过表达拟南芥株系。该株系在150–200 mol/L的盐胁迫下,发芽率、根长等指标明显下降,初步说明过表达GmWRKY56会增加植物对盐的敏感性。目前70%的大豆WRKY转录因子家族基因尚未被发掘,一方面,可以通过对盐胁迫下相关大豆WRKY转录因子的研究,加快大豆耐盐分子机理的研究;另一方面,在实际大田条件下,大豆芽期耐盐性是直接决定盐碱地上大豆能否良好生长的关键,因此大豆芽期耐盐可能成为大豆耐盐育种研究的一个重要方向[60]。另外,借助基因工程等分子育种手段,将耐盐WRKY转录因子导入到具有应用价值的品种中,或通过基因编辑敲除大豆耐盐相关的负调控WRKY转录因子,育成综合性状优良的新品种,这对于大豆生产具有重要意义。在高通量技术发达的时代,转录组是研究WRKY基因在不同胁迫条件下在不同组织中的表达的重要手段,有助于研究胁迫响应基因及其底层网络。通过这些信息可以进一步用于利用转基因方法开发具有更高产量和抗盐性的新品种[61]。
随着越来越多的植物基因组被测序,特别是在重要的经济作物中,WRKY基因的全基因组鉴定将有助于筛选植物中与盐胁迫相关的功能基因。另外,鉴定受WRKY转录因子调控的下游基因对于阐明盐胁迫反应的调控网络有至关重要的作用。在之前的研究中,对WRKY转录因子功能的研究在很大程度上依赖于转录组学和基因的功能预测。目前,先进的分子生物学技术的发展,如微阵列、免疫沉淀分析和测序技术的结合,可以加速WRKY转录因子家族新功能的研究进展。此外,WRKY转录因子参与调控的非编码RNA和表观遗传修饰等相关内容应在未来的研究中进行探索。最终,在环境恶化持续加剧的背景下,利用WRKY转录因子筛选抗逆性植物品种,提高农作物的抗盐性,更好地适应盐碱地生长,将有利于提高农作物的产量和品质,为夯实国家粮食安全的根基提供重要保障。
| [1] |
HANIN M, EBEL C, NGOM M, LAPLAZE L, MASMOUDI K. New insights on plant salt tolerance mechanisms and their potential use for breeding. Frontiers in Plant Science, 2016, 7: 1787.
|
| [2] |
KIM JM, SASAKI T, UEDA M, SAKO K, SEKI M. Chromatin changes in response to drought, salinity, heat, and cold stresses in plants. Frontiers in Plant Science, 2015, 6: 114.
|
| [3] |
YANG YQ, GUO Y. Unraveling salt stress signaling in plants. Journal of Integrative Plant Biology, 2018, 60(9): 796-804. DOI:10.1111/jipb.12689
|
| [4] |
ZHU JK. Abiotic stress signaling and responses in plants. Cell, 2016, 167(2): 313-324. DOI:10.1016/j.cell.2016.08.029
|
| [5] |
LIU Y, YANG TY, LIN ZK, GU BJ, XING CH, ZHAO LY, DONG HZ, GAO JZ, XIE ZH, ZHANG SL, HUANG XS. A WRKY transcription factor PbrWRKY53 from Pyrus betulaefolia is involved in drought tolerance and AsA accumulation. Plant Biotechnology Journal, 2019, 17(9): 1770-1787.
|
| [6] |
PANDEY SP, SOMSSICH IE. The role of WRKY transcription factors in plant immunity. Plant Physiology, 2009, 150(4): 1648-1655. DOI:10.1104/pp.109.138990
|
| [7] |
RUSHTON PJ, SOMSSICH IE, RINGLER P, SHEN QJ. WRKY transcription factors. Trends in Plant Science, 2010, 15(5): 247-258. DOI:10.1016/j.tplants.2010.02.006
|
| [8] |
ISHIGURO S, NAKAMURA K. Characterization of a cDNA encoding a novel DNA-binding protein, SPF1, that recognizes SP8 sequences in the 5′ upstream regions of genes coding for sporamin and β-amylase from sweet potato. Molecular and General Genetics MGG, 1994, 244(6): 563-571. DOI:10.1007/BF00282746
|
| [9] |
ULKER B, SOMSSICH IE. WRKY transcription factors: from DNA binding towards biological function. Current Opinion in Plant Biology, 2004, 7(5): 491-498. DOI:10.1016/j.pbi.2004.07.012
|
| [10] |
WU KL, GUO ZJ, WANG HH, LI J. The WRKY family of transcription factors in rice and Arabidopsis and their origins. DNA Research, 2005, 12(1): 9-26. DOI:10.1093/dnares/12.1.9
|
| [11] |
SCHMUTZ J, CANNON SB, SCHLUETER J, MA JX, MITROS T, NELSON W, HYTEN DL, SONG QJ, THELEN JJ, CHENG JL, XU D, HELLSTEN U, MAY GD, YU Y, SAKURAI T, UMEZAWA T, BHATTACHARYYA MK, SANDHU D, VALLIYODAN B, LINDQUIST E, et al. Genome sequence of the palaeopolyploid soybean. Nature, 2010, 463(7278): 178-183. DOI:10.1038/nature08670
|
| [12] |
MANGELSEN E, KILIAN J, BERENDZEN KW, KOLUKISAOGLU UH, HARTER K, JANSSON C, WANKE D. Phylogenetic and comparative gene expression analysis of barley (Hordeum vulgare) WRKY transcription factor family reveals putatively retained functions between monocots and dicots. BMC Genomics, 2008, 9: 194. DOI:10.1186/1471-2164-9-194
|
| [13] |
LING J, JIANG WJ, ZHANG Y, YU HJ, MAO ZC, GU XF, HUANG SW, XIE BY. Genome-wide analysis of WRKY gene family in Cucumis sativus. BMC Genomics, 2011, 12: 471. DOI:10.1186/1471-2164-12-471
|
| [14] |
HUANG SX, GAO YF, LIU JK, PENG XL, NIU XL, FEI ZJ, CAO SQ, LIU YS. Genome-wide analysis of WRKY transcription factors in Solanum lycopersicum. Molecular Genetics and Genomics, 2012, 287(6): 495-513. DOI:10.1007/s00438-012-0696-6
|
| [15] |
MIAO Y, SMYKOWSKI A, ZENTGRAF U. A novel upstream regulator of WRKY53 transcription during leaf senescence in Arabidopsis thaliana. Plant Biology, 2008, 10: 110-120. DOI:10.1111/j.1438-8677.2008.00083.x
|
| [16] |
HARA K, YAGI M, KUSANO T, SANO H. Rapid systemic accumulation of transcripts encoding a tobacco WRKY transcription factor upon wounding. Molecular and General Genetics MGG, 2000, 263(1): 30-37. DOI:10.1007/PL00008673
|
| [17] |
LI SJ, ZHOU X, CHEN LG, HUANG WD, YU DQ. Functional characterization of Arabidopsis thaliana WRKY39 in heat stress. Molecules and Cells, 2010, 29(5): 475-483. DOI:10.1007/s10059-010-0059-2
|
| [18] |
RUSHTON PJ, TORRES JT, PARNISKE M, WERNERT P, HAHLBROCK K, SOMSSICH IE. Interaction of elicitor-induced DNA-binding proteins with elicitor response elements in the promoters of parsley PR1 genes. The EMBO Journal, 1996, 15(20): 5690-5700. DOI:10.1002/j.1460-2075.1996.tb00953.x
|
| [19] |
PHUKAN UJ, JEENA GS, SHUKLA RK. WRKY transcription factors: molecular regulation and stress responses in plants. Frontiers in Plant Science, 2016, 7: 760.
|
| [20] |
EULGEM T, RUSHTON PJ, ROBATZEK S, SOMSSICH IE. The WRKY superfamily of plant transcription factors. Trends in Plant Science, 2000, 5(5): 199-206. DOI:10.1016/S1360-1385(00)01600-9
|
| [21] |
RUSHTON PJ, BOKOWIEC MT, HAN SC, ZHANG HB, BRANNOCK JF, CHEN XF, LAUDEMAN TW, TIMKO MP. Tobacco transcription factors: novel insights into transcriptional regulation in the Solanaceae. Plant Physiology, 2008, 147(1): 280-295. DOI:10.1104/pp.107.114041
|
| [22] |
LI WX, PANG SY, LU ZG, JIN B. Function and mechanism of WRKY transcription factors in abiotic stress responses of plants. Plants (Basel, Switzerland), 2020, 9(11): 1515.
|
| [23] |
CHEN LG, SONG Y, LI SJ, ZHANG LP, ZOU CS, YU DQ. The role of WRKY transcription factors in plant abiotic stresses. Biochimica et Biophysica Acta, 2012, 1819(2): 120-128.
|
| [24] |
CHENG ZY, LUAN YT, MENG JS, SUN J, TAO J, ZHAO DQ. WRKY transcription factor response to high-temperature stress. Plants, 2021, 10(10): 2211. DOI:10.3390/plants10102211
|
| [25] |
CIOLKOWSKI I, WANKE D, BIRKENBIHL RP, SOMSSICH IE. Studies on DNA-binding selectivity of WRKY transcription factors lend structural clues into WRKY-domain function. Plant Molecular Biology, 2008, 68(1): 81-92.
|
| [26] |
ZENTGRAF U, LAUN T, MIAO Y. The complex regulation of WRKY53 during leaf senescence of Arabidopsis thaliana. European Journal of Cell Biology, 2010, 89(2/3): 133-137.
|
| [27] |
SHEIKH AH, ESCHEN-LIPPOLD L, PECHER P, HOEHENWARTER W, SINHA AK, SCHEEL D, LEE J. Regulation of WRKY46 transcription factor function by mitogen-activated protein kinases in Arabidopsis thaliana. Frontiers in Plant Science, 2016, 7: 61.
|
| [28] |
FIIL BK. Gene regulation by MAP kinase cascades. Current Opinion in Plant Biology, 2009, 12(5): 615-621. DOI:10.1016/j.pbi.2009.07.017
|
| [29] |
KIM CY, ZHANG SQ. Activation of a mitogen-activated protein kinase cascade induces WRKY family of transcription factors and defense genes in tobacco. The Plant Journal: for Cell and Molecular Biology, 2004, 38(1): 142-151. DOI:10.1111/j.1365-313X.2004.02033.x
|
| [30] |
GRIERSON C, DU JS, de TORRES ZABALA M, BEGGS K, SMITH C, HOLDSWORTH M, BEVAN M. Separate cis sequences and trans factors direct metabolic and developmental regulation of a potato tuber storage protein gene. The Plant Journal, 1994, 5(6): 815-826. DOI:10.1046/j.1365-313X.1994.5060815.x
|
| [31] |
SUN CX, PALMQVIST S, OLSSON H, BORÉN M, AHLANDSBERG S, JANSSON C. A novel WRKY transcription factor, SUSIBA2, participates in sugar signaling in barley by binding to the sugar-responsive elements of the iso1 promoter. The Plant Cell, 2003, 15(9): 2076-2092. DOI:10.1105/tpc.014597
|
| [32] |
WANI SH, ANAND S, SINGH B, BOHRA A, JOSHI R. WRKY transcription factors and plant defense responses: latest discoveries and future prospects. Plant Cell Rep, 2021, 40: 1071-1085. DOI:10.1007/s00299-021-02691-8
|
| [33] |
BABITHA KC, RAMU SV, PRUTHVI V, MAHESH P, NATARAJA KN, UDAYAKUMAR M. Co-expression of AtbHLH17 and AtWRKY28 confers resistance to abiotic stress in Arabidopsis. Transgenic Research, 2013, 22(2): 327-341. DOI:10.1007/s11248-012-9645-8
|
| [34] |
KRISHNAMURTHY P, VISHAL B, HO WJ, LOK FCJ, LEE FSM, KUMAR PP. Regulation of a cytochrome P450 gene CYP94B1 by WRKY33 transcription factor controls apoplastic barrier formation in roots to confer salt tolerance. Plant Physiology, 2020, 184(4): 2199-2215. DOI:10.1104/pp.20.01054
|
| [35] |
DING ZJ, YAN JY, LI CX, LI GX, WU YR, ZHENG SJ. Transcription factor WRKY46 modulates the development of Arabidopsis lateral roots in osmotic/salt stress conditions via regulation of ABA signaling and auxin homeostasis. The Plant Journal, 2015, 84(1): 56-69. DOI:10.1111/tpj.12958
|
| [36] |
ZHU H, JIANG YN, GUO Y, HUANG JB, ZHOU MH, TANG YY, SUI JM, WANG JS, QIAO LX. A novel salt inducible WRKY transcription factor gene, AhWRKY75, confers salt tolerance in transgenic peanut. Plant Physiology and Biochemistry: PPB, 2021, 160: 175-183. DOI:10.1016/j.plaphy.2021.01.014
|
| [37] |
YUAN SL, HU D, WANG Y, SHAO C, LIU TK, ZHANG CW, CHENG F, HOU XL, LI Y. BcWRKY1 confers salt sensitivity via inhibiting reactive oxygen species scavenging. Plant Molecular Biology, 2022, 109(6): 741-759. DOI:10.1007/s11103-022-01272-x
|
| [38] |
ZHU L, LI SL, OUYANG MZ, YANG LM, SUN SR, WANG YJ, CAI XX, WU GX, LI YM. Overexpression of watermelon ClWRKY20 in transgenic Arabidopsis improves salt and low-temperature tolerance. Scientia Horticulturae, 2022, 295: 110848. DOI:10.1016/j.scienta.2021.110848
|
| [39] |
LI PL, SONG AP, GAO CY, WANG LX, WANG YJ, SUN J, JIANG JF, CHEN FD, CHEN SM. Chrysanthemum WRKY gene CmWRKY17 negatively regulates salt stress tolerance in transgenic Chrysanthemum and Arabidopsis plants. Plant Cell Reports, 2015, 34(8): 1365-1378. DOI:10.1007/s00299-015-1793-x
|
| [40] |
ZHOU L. Overexpression of a cotton (Gossypium hirsutum) WRKY gene, GhWRKY34, in Arabidopsis enhances salt-tolerance of the transgenic plants. Plant Physiology and Biochemistry, 2015, 96: 311-320. DOI:10.1016/j.plaphy.2015.08.016
|
| [41] |
ZHOU QY, TIAN AG, ZOU HF, XIE ZM, LEI G, HUANG J, WANG CM, WANG HW, ZHANG JS, CHEN SY. Soybean WRKY-type transcription factor genes, GmWRKY13, GmWRKY21, and GmWRKY54, confer differential tolerance to abiotic stresses in transgenic Arabidopsis plants. Plant Biotechnology Journal, 2008, 6(5): 486-503. DOI:10.1111/j.1467-7652.2008.00336.x
|
| [42] |
QIN Z, HOU FY, LI AX, DONG SX, WANG QM, ZHANG LM. Transcriptome-wide identification of WRKY transcription factor and their expression profiles under salt stress in sweetpotato (Ipomoea batatas L.). Plant Biotechnology Reports, 2020, 14(5): 599-611. DOI:10.1007/s11816-020-00635-4
|
| [43] |
DONG QL, ZHENG WQ, DUAN DY, HUANG D, WANG Q, LIU CH, LI C, GONG XQ, LI CY, MAO K, MA FW. MdWRKY30, a group Ⅱa WRKY gene from apple, confers tolerance to salinity and osmotic stresses in transgenic apple callus and Arabidopsis seedlings. Plant Science, 2020, 299: 110611. DOI:10.1016/j.plantsci.2020.110611
|
| [44] |
SU MY, WANG S, LIU WJ, YANG M, ZHANG ZY, WANG N, CHEN XS. Interaction between MdWRKY55 and MdNAC17-L enhances salt tolerance in apple by activating MdNHX1 expression. Plant Science, 2022, 320: 111282. DOI:10.1016/j.plantsci.2022.111282
|
| [45] |
NAM TN, THIA LH, MAI DS, TUAN NV. Overexpression of NbWRKY79 enhances salt stress tolerance in Nicotiana benthamiana. Acta Physiologiae Plantarum, 2017, 39(6): 121. DOI:10.1007/s11738-017-2423-8
|
| [46] |
HUANG SZ, HU LJ, ZHANG SH, ZHANG MX, JIANG WZ, WU T, DU XL. Rice OsWRKY50 mediates ABA-dependent seed germination and seedling growth, and ABA-independent salt stress tolerance. International Journal of Molecular Sciences, 2021, 22(16): 8625. DOI:10.3390/ijms22168625
|
| [47] |
LIN LK, YUAN KL, HUANG YD, DONG HZ, QIAO QH, XING CH, HUANG XS, ZHANG SL. A WRKY transcription factor PbWRKY40 from Pyrus betulaefolia functions positively in salt tolerance and modulating organic acid accumulation by regulating PbVHA-B1 expression. Environmental and Experimental Botany, 2022, 196: 104782. DOI:10.1016/j.envexpbot.2022.104782
|
| [48] |
WANG GW, WANG XW, MA HP, FAN HL, LIN F, CHEN JH, CHAI TY, WANG H. PcWRKY11, an Ⅱ-d WRKY transcription factor from Polygonum cuspidatum, enhances salt tolerance in transgenic Arabidopsis thaliana. International Journal of Molecular Sciences, 2022, 23(8): 4357. DOI:10.3390/ijms23084357
|
| [49] |
YU XY, PAN Y, DONG Y, LU B, ZHANG C, YANG MS, ZUO LH. Cloning and overexpression of PeWRKY31 from Populus×euramericana enhances salt and biological tolerance in transgenic Nicotiana. BMC Plant Biology, 2021, 21(1): 80. DOI:10.1186/s12870-021-02856-3
|
| [50] |
SONG YS, LI JL, SUI Y, HAN GL, ZHANG Y, GUO SJ, SUI N. The sweet sorghum SbWRKY50 is negatively involved in salt response by regulating ion homeostasis. Plant Molecular Biology, 2020, 102(6): 603-614. DOI:10.1007/s11103-020-00966-4
|
| [51] |
CAI RH, DAI W, ZHANG CS, WANG Y, WU M, ZHAO Y, MA Q, XIANG Y, CHENG BJ. The maize WRKY transcription factor ZmWRKY17 negatively regulates salt stress tolerance in transgenic Arabidopsis plants. Planta, 2017, 246(6): 1215-1231. DOI:10.1007/s00425-017-2766-9
|
| [52] |
FANG X, LI W, YUAN HT, CHEN HW, BO C, MA Q, CAI RH. Mutation of ZmWRKY86 confers enhanced salt stress tolerance in maize. Plant Physiology and Biochemistry, 2021, 167: 840-850. DOI:10.1016/j.plaphy.2021.09.010
|
| [53] |
YAN JW, LI J, ZHANG HP, LIU Y, ZHANG AY. ZmWRKY104 positively regulates salt tolerance by modulating ZmSOD4 expression in maize. The Crop Journal, 2022, 10(2): 555-564. DOI:10.1016/j.cj.2021.05.010
|
| [54] |
LLORCA CM, POTSCHIN M, ZENTGRAF U. bZIPs and WRKYs: two large transcription factor families executing two different functional strategies. Frontiers in Plant Science, 2014, 5: 169.
|
| [55] |
EULGEM T. Regulation of the Arabidopsis defense transcriptome. Trends in Plant Science, 2005, 10(2): 71-78. DOI:10.1016/j.tplants.2004.12.006
|
| [56] |
SHEN ZD, YAO J, SUN J, CHANG LW, WANG SJ, DING MQ, QIAN ZY, ZHANG HL, ZHAO N, SA G, HOU PC, LANG T, WANG FF, ZHAO R, SHEN X, CHEN SL. Populus euphratica HSF binds the promoter of WRKY1 to enhance salt tolerance. Plant Science, 2015, 235: 89-100. DOI:10.1016/j.plantsci.2015.03.006
|
| [57] |
FINATTO T, VIANA VE, WOYANN LG, BUSANELLO C, MAIA LCD, OLIVEIRA AC. Can WRKY transcription factors help plants to overcome environmental challenges?. Genetics and Molecular Biology, 2018, 41(3): 533-544. DOI:10.1590/1678-4685-gmb-2017-0232
|
| [58] |
MIRZA Z, HAQUE MM, GUPTA M. WRKY transcription factors: a promising way to deal with arsenic stress in rice. Mol Biol Rep, 2022, 49: 10895-10904. DOI:10.1007/s11033-022-07772-9
|
| [59] |
方义生, 曹东, 杨红丽, 刘小荣, 张恒斌, 陈李淼, 周新安. 大豆耐盐相关基因研究进展. 中国油料作物学报, 2020, 42(4): 512-526. FANG YS, CAO D, YANG HL, LIU XR, ZHANG HB, CHEN LM, ZHOU XA. Research progress of salt-tolerance genes in soybean. Chinese Journal of Oil Crop Sciences, 2020, 42(4): 512-526 (in Chinese). DOI:10.19802/j.issn.1007-9084.2020167 |
| [60] |
张威, 廖锡良, 喻德跃, 阚贵珍. 大豆耐盐性研究进展. 土壤与作物, 2018, 7(3): 284-292. ZHANG W, LIAO XL, YU DY, KAN GZ. A review of salt tolerance in soybean (Glycine max(L.) Merill). Soils and Crops, 2018, 7(3): 284-292 (in Chinese). |
| [61] |
GOYAL P, DEVI R, VERMA B, HUSSAIN S, ARORA P, TABASSUM R, GUPTA S. WRKY transcription factors: evolution, regulation, and functional diversity in plants. Protoplasma, 2022, 1-18.
|
 2023, Vol. 39
2023, Vol. 39