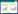中国科学院微生物研究所、中国微生物学会主办
文章信息
- 牛坤, 高利平, 葛丽蓉, 柳志强, 郑裕国
- NIU Kun, GAO Liping, GE Lirong, LIU Zhiqiang, ZHENG Yuguo
- 大肠杆菌代谢工程改造合成L-高丝氨酸及其衍生物研究进展
- Advances in the biosynthesis of L-homoserine and its derivatives by metabolic engineering of Escherichia coli
- 生物工程学报, 2022, 38(12): 4385-4402
- Chinese Journal of Biotechnology, 2022, 38(12): 4385-4402
- 10.13345/j.cjb.220377
-
文章历史
- Received: May 10, 2022
- Accepted: July 7, 2022
L-高丝氨酸(L-homoserine, L-HS) 又名2-氨基-4-羟基丁酸,广泛应用于γ-丁内酯、L-高丝氨酸内酯、1, 4-丁二醇等化合物的合成,并可以作为合成农药草铵膦的原料[1]。L-高丝氨酸作为一种非必需氨基酸,存在于细菌的肽聚糖中,也是微生物细胞内多种必需氨基酸,如L-苏氨酸、L-异亮氨酸和L-甲硫氨酸等生物合成的中间代谢产物[2]。传统的L-高丝氨酸生产方法主要是化学合成法,但该方法存在反应步骤长、反应条件苛刻和安全性低等问题,逐渐被取代。由于对气候变化和环境问题的日益关注,用于化学品绿色生产的生物合成技术越来越受到关注,特别是近年来合成生物学的发展,更为L-高丝氨酸及其衍生物的生物合成提供了有效的代谢工程改造策略[3-8]。通过代谢途径的理性设计和代谢改造,已实现了多种氨基酸的发酵法生产,如L-苏氨酸、L-赖氨酸和L-谷氨酸等。与化学合成法相比,微生物发酵合成氨基酸具有工艺环保、成本低、生产效率高等优点,逐渐成为氨基酸生产的主流方式[9]。大肠杆菌(Escherichia coli) 和谷氨酸棒杆菌(Corynebacterium glutamicum) 作为重要的工业微生物,已被用于生产L-天冬氨酸家族氨基酸,如L-苏氨酸和L-异亮氨酸,从而显示出生产L-高丝氨酸的巨大潜力[10]。相较于谷氨酸棒杆菌,大肠杆菌因具有清晰的遗传背景、繁殖迅速、高效的基因操作和相对简单的培养条件,而被大多科研工作者选作底盘菌株。因此,大肠杆菌L-高丝氨酸高产菌株的代谢改造具有重要的研究意义。
O-乙酰-L-高丝氨酸(O-acetyl-L-homoserine, OAH) 和O-琥珀酰-L-高丝氨酸(O-succinyl-L- homoserine, OSH) 是L-高丝氨酸下游代谢合成的重要衍生物,分别由高丝氨酸乙酰基转移酶(由metX编码) 和琥珀酰基转移酶(由metA编码) 催化生成[11]。前者的酰基供体为乙酰-CoA,后者的酰基供体为琥珀酰-CoA。在微生物代谢过程中,OAH和OSH可以合成多种C4化合物,在工业生产琥珀酸、1, 4-丁二醇、高丝氨酸内酯、γ-丁内酯、2, 4-二羟基丁酸及其衍生物等化合物过程中占据重要地位[12-13],同时也是合成含硫和含磷氨基酸的重要前体物质,如L-甲硫氨酸、L-草铵膦等。由于汽油价格的上涨、石油储量的枯竭和发酵成本的降低,OSH和OAH有望成为一系列C4化学品的生物合成平台。随着基因编辑技术的发展和代谢工程理论的完善,人们已深入研究利用大肠杆菌发酵合成L-高丝氨酸及其衍生物[14-17]。本文介绍了L-高丝氨酸及其衍生物的生物合成途径,并进一步综述了该类氨基酸生物合成的途径改造策略及相关研究进展[18-19]。
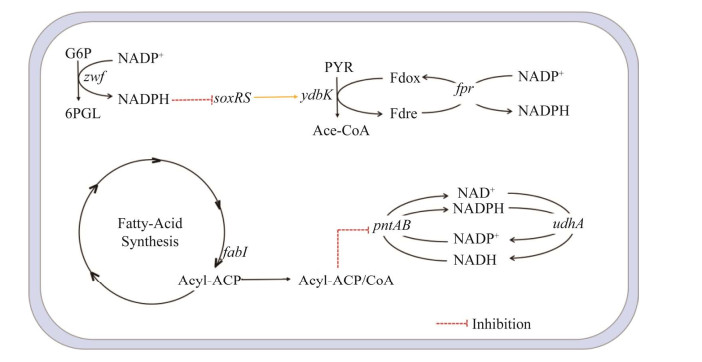
1 L-高丝氨酸及其衍生物的代谢合成途径生物法合成L-高丝氨酸最早是通过谷氨酸棒杆菌发酵生产L-苏氨酸和L-赖氨酸的过程中获得的,该菌株在含80 g/L玉米浆和150 g/L蔗糖的培养基中发酵72 h,最终得到14.5 g/L的L-高丝氨酸。在大肠杆菌中,以葡萄糖为底物合成L-高丝氨酸的代谢途径如图 1所示,菌株首先通过磷酸转移酶系统(phosphotransferase system, PTS) 吸收葡萄糖,后经糖酵解途径(glycolytic pathway, 又称Embden-Meyerhof pathway, EMP) 转化为磷酸烯醇式丙酮酸(phosphoenolpyruvate, PEP),一部分PEP转化为丙酮酸(pyruvic acid, PYR),并进一步代谢为乙酰-CoA (acetyl-CoA) 进入三羧酸循环(tricarboxylic acid cycle, TCA cycle),另一部分转化为天冬氨酸家族氨基酸的前体——草酰乙酸(oxalacetic acid, OAA)。OAA首先在天冬氨酸转氨酶(由aspC编码) 作用下转化为天冬氨酸,然后分别在天冬氨酸激酶(由thrA、metL和lysC等基因编码)、天冬氨酸半醛脱氢酶(由基因asd编码) 和高丝氨酸合成酶(由thrA和metL基因编码) 作用下催化为L-高丝氨酸[20]。L-高丝氨酸会进一步和乙酰-CoA在高丝氨酸乙酰转移酶(由metX编码) 的作用下生成OAH;琥珀酰-CoA在琥珀酰基转移酶(由metA编码) 的作用下生成OSH。
|
| 图 1 L-高丝氨酸及其衍生物合成途径 Fig. 1 Biosynthesis pathway of L-homoserine and its derivatives. Glu: glucose; G6P: glucose-6-phosphate; PEP: phosphoenolpyruvate; PYR: pyruvate; LAC: lactic acid; Ace-CoA: acetyl-CoA; ACE: acetic acid; OAA: oxaloacetic acid; CIT: citric acid; ICL: isocitric acid; α-KG: α-ketoglutaric acid; SuCoA: succinyl-CoA; SUC: succinic acid; FUM: fumaric acid; MAL: L-malate; GLU: glutamic acid; ASP: L-aspartic acid; ASP-P: aspartyl-phosphate; ASP-SA: L-aspartate-4-semialdehyde; HS: homoserine; OSH: O-succinyl-L-homoserine; OAH: O-acetyl-L-homoserine; LYS: lysine; THR: threonine; 6PGL: 6-phosphogluconic acid; MET: methionine; galP: encoding galactoside symporter; ptsG: encoding α-glucoside PTS system EⅡCB component; glk: encoding glucokinase; zwf: encoding glucose-6-phosphate dehydrogenase; pykA/pykF: encoding pyruvate kinase; ldhA: encoding lactate dehydrogenase; ppc: encoding phosphoenolpyruvate carboxylase; pck: encoding phosphoenolpyruvate carboxykinase; pyc: encoding pyruvate carboxylase; PDH: encoding pyruvate dehydrogenase; pta: encoding phosphate acetyltransferase; ackA: encoding acetate kinase; gltA: encoding citrate synthase; sucAB: encoding α-ketoglutaric acid dehydrogenase; sucC: encoding Succinate dehydrogenase; sucD: encoding succinyl-CoA synthetase; aceA: encoding isocitrate lyase; aceB: encoding malate synthase; iclR: encoding IclR family transcriptional regulator; lacI: encoding LacI family transcriptional regulator; rhtA: encoding inner membrane transporter; gltBD: encoding glutamate synthase; aspC: encoding aspartate aminotransferase; aspA: encoding aspartate oxidase; lysC: encoding aspartate kinase; thrA: encoding aspartokinase Ⅰ and homoserine dehydrogenase Ⅰ; metL: encoding bifunctional aspartokinase/homoserine dehydrogenase; asd: encoding aspartate-semialdehyde dehydrogenase; lysA: encoding diaminopimelate decarboxylase; thrB: encoding homoserine kinase; rhtB: encoding homoserine/homoserine lactone efflux protein; eamA: encoding O-acetylserine/cysteine efflux transporter; metA: encoding homoserine-O-succinyltransferase; metX: encoding homoserine-O- acetyltransferase; metB: encoding O-succinylhomoserine lyase; pntAB: encoding proton-translocating NADP+ transhydrogenase; sthA: encoding NADP transhydrogenase; fpr: encoding NADP+ reductase; fabI: encoding enoyl-[acyl-carrier protein] reductase. |
| |
基于L-高丝氨酸及其衍生物的重要用途,近年来国内外研究团队对其生物法合成开展了大量的研究工作,进展如表 1所示。目前虽然在底物利用、代谢途径中关键节点的改造、代谢途径中反馈抑制的解除、辅酶NADPH的循环供应、产物外运系统的增强等方面已开展了大量研究工作,但其发酵产量尚未达到工业化水平。
| Chassis cell | Genotype | Titer (g/L) | Yield (g/g) | Reference |
| E. coli W3110 | ΔJIB* ΔthrB ΔmetA ΔlysA ΔiclR ΔptsG ΔgalR ΔlacI: : Ptrc-rhtA Ptrc-rhtA Ptrc-eamA Ptrc-metL Ptrc-thrA Ptrc-glk Ptrc-gltB/pACYC-pyc-thrA-lysC | 37.57 (HS, 5 L fermentor) | 0.31 | [21] |
| E. coli BW25113 | ΔmetA ΔlysA ΔthrB ΔlacI ΔsthA: : Ptac-pntAB ΔldhA ΔpoxB ΔpflB ΔfliK: : Ptac-rhtB ΔyeeJ: : P119-rhtB ΔptsG: : P119-glk Δ galR: : P119-zglf ΔompT: : Ptac-ppc ΔiclR ΔyjiV: : Ptac-aspC-gdhA/pS95s-thrA* asd aspA |
84.10 (HS, 5 L fermentor) | 0.50 | [22] |
| E. coli W3110 | Ptrc-ppc Ptrc-aspA Ptrc-thrAfbr Ptrc-pntAB PfliC-thrB | 60.10 (HS, 5 L fermentor) | 0.42 | [23] |
| E. coli W3110 | ΔlysA ΔmetA ΔthrBC ΔiclR ΔgltA ΔpykA ΔpykF/pKK-miniPtac-thrA | 35.80 (HS, 7 L 3fermentor) | 0.35 | [24] |
| E. coli W3110 | ΔlacI ΔlysA ΔthrBC ΔmetA ΔtdcC/pBRmetL–pNrhtA | 39.54 (HS, 15 L fermentor) | 0.29 | [25] |
| E. coli W3110 | ΔmetI ΔmetJ ΔthrB ΔmetB ΔmetA ΔlysA Ptrc-metL Ptrc-ppc ΔiclR/ PACP-Ptrc-thrA*lysC*pyc* and PSCR-Ptrc-scrA scrB scrK | 11.10 (HS, shake flask) | 0.16 (sucrose) |
[20] |
| C. glutamicum ATCC 13032 | ΔmcbR ΔthrB ΔmetD Psod-lysCT311I Psod-asd Psod-hom Psod-pycP458S Δpck: : Psod-aspC Psod-brnFE/ pEC-thrAS345F | 8.80 (HS, shake flask) | 0.09 | [26] |
| E. coli W3110 | ΔmetJ ΔmetI ΔmetB ΔthrB Ptrc-metL/pTrc99A-metA-yjeH-thrAfbr | 102.50 (OSH, 5 L fermentor) | / | [27] |
| E. coli W3110 | ΔmetJ ΔmetI ΔmetB ΔthrB ΔsucD Ptrc-thrAfbr Ptrc-sucA Trc-metL/pTrc99A-metA11-yjeH | 24.10 (OSH, shake flask) | 0.61 | [28] |
| E. coli W3110 | ΔmetJ ΔmetI ΔmetB Ptrc-metL/pTrc99A-metAfbr-thrAfbr-yjeH | 9.31 (OSH, shake flask) | 0.47 | [29] |
| E. coli W3110 | ΔmetB ΔthrB ΔmetA ΔlysA Pppc: : PM1‑46/pTrc-M18TLA pACYC-ADAEFLm | 62.70 (OAH, 7.5 L fermentor) | 0.45 | [30] |
| E. coli W3110 | ΔmetJ ΔmetI ΔmetB ΔthrB ΔmetA ΔlysA ΔlacI: : Ptrc-rhtA ΔiclR Ptrc-metL Ptrc-thrA Ptrc-rhtA Ptrc-eamA J23100-glpD /pTrc99A-metXcg | 9.42 (OAH, shake flask) | 0.31 | [31] |
| C. glutamicum ATCC13032 | ∆mcbR ∆metD ∆thrB ∆NCgl2360: : Psod-thrAS345F ∆NCgl2688 ∆metY ∆pck: : Psod-aspC Psod-pycP458S Psod-lysCT311I Psod-asd Psod-homV59A Psod-brnFE-icdM1V-dapAM1V ∆NCgl2688: : PNCgl1676-metXr ∆Cas9 ∆recET/pEC-thrAS345F-PNCgl1676-metXr | 17.40 (OAH, 5 L fermentor) | / | [32] |
| “/”means no data. | ||||
葡萄糖是大肠杆菌最适宜的发酵底物,大肠杆菌通常利用自身的PTS系统摄取葡萄糖,但此过程在吸收1分子葡萄糖转化为葡萄糖-6-磷酸的同时,会消耗1分子PEP转化为PYR,从而消耗了合成L-高丝氨酸的前体,使其理论转化率降低。当大肠杆菌在以葡萄糖为唯一碳源培养基中生长时,PTS系统会消耗细胞内约50%的可用PEP,而其他酶,如PEP羧化酶、PYR激酶、UDP-N-乙基葡萄糖胺烯醇转移酶和3-脱氧-D-阿拉伯庚酮糖-7-磷酸合成酶(DAHP synthase) 所催化的反应分别消耗约16%、15%、16%和3%的剩余PEP[33-34]。因此,采用非PTS系统取代PTS葡萄糖转运系统可以有效利用葡萄糖,提高L-高丝氨酸前体PEP的供应,提升底物转化率。但实验发现PTS系统的破坏会影响菌株的生长,为解决该问题,本团队在敲除PTS系统的大肠杆菌中引入半乳糖渗透酶系统(galP-glk系统),发现过表达galP,并且利用原位启动子替换将glk启动子替换为Ptrc启动子后,可以增强菌体对葡萄糖的利用率,稳定菌体生长,提高L-高丝氨酸产量,摇瓶中L-高丝氨酸产量提高了19%,达到6.27 g/L[11]。
改变碳源也是增加目标产物的一种方法,除葡萄糖外,不少研究学者还尝试采用其他底物合成高丝氨酸及其衍生物。Hong等通过过表达scrKABR操纵子以利用蔗糖作为碳源发酵生产OSH,其糖酸转化率为0.66 g/g,接近氨基酸工业化生产水平[1]。本团队也开展了以蔗糖为底物发酵合成高丝氨酸的研究,在大肠杆菌中过表达蔗糖代谢基因scrA、scrB和scrK,发现菌株在摇瓶中的高丝氨酸产量达到了11.1 g/L,高于利用葡萄糖时的发酵产量[20]。虽然蔗糖代谢基因的导入对菌株本身造成了一定的负担,但实验发现蔗糖作为碳源进行发酵时更利于菌株的生长与产物的合成。除此之外,随着生物柴油产业的发展,粗甘油作为生物柴油的一种副产物,其产量越来越高,价格也日趋下降[35]。本团队考察了以甘油为底物代谢合成OAH的可行性,在前期构建的葡萄糖利用菌株基础上,过表达与甘油转运和降解相关的基因,并修饰甘油氧化途径,可以使大肠杆菌很好地利用甘油进行生长,且OAH在摇瓶中的发酵水平达到9.21 g/L[36]。因此,除葡萄糖外,其他廉价底物的有效利用也是L-高丝氨酸及其衍生物工业化开发过程中需要研究的重点方向。
2.2 代谢途径中关键节点的改造 2.2.1 PEP-PYR-OAA节点的代谢分配在大多数细菌中,EMP途径和TCA循环是碳源中心代谢的主要途径,是碳水化合物分解为PEP、PYR和乙酰-CoA的主要途径,为细胞的合成提供能量和前体物质。TCA循环在分解代谢和合成代谢中发挥双重作用,它催化乙酰-CoA完全氧化为CO2,并为生物合成提供前体代谢物、ATP和FADH2[37]。如图 1所示,上述途径由PEP、PYR和OAA三个节点连接,涉及系列反应,在EMP途径中,PEP和PYR最终产物通过生成乙酰-CoA (氧化丙酮酸脱羧获得) 和OAA (C3-羧化获得) 进入TCA循环。在糖异生条件下,TCA循环中间产物OAA或苹果酸(malic acid, MAL) 又可以通过脱羧作用(C4-脱羧) 转化为PYR和PEP。因此,PEP-PYR-OAA节点又为糖异生提供了直接前体,是与新陈代谢中心碳流分布高度相关的切换点。合理调控关键节点处的代谢分配是提高目的产物生物合成的关键。
在PEP-PYR-OAA节点处生成OAA的有限可用性可能是L-高丝氨酸合成的瓶颈,OAA是合成L-天冬氨酸家族氨基酸(包括L-高丝氨酸) 的关键前体,文献中已报道增强OAA含量可以促进L-高丝氨酸的合成[24, 38]。在大肠杆菌中,由PEP羧化酶(由ppc编码) 催化的补缺途径是OAA的重要来源[39]。Zhang等将Ptrc启动子介导的ppc基因整合到大肠杆菌基因组中过表达该基因,增加了从葡萄糖到OAA的碳通量,所得菌株生物量显著提高,且L-高丝氨酸的产量提高了61.1%[23]。本团队分别通过原位启动子替换和质粒导入的方法过表达了ppc基因和抗反馈基因pyccgP458S (编码PYR羧化酶),实验结果表明ppc基因的过表达使PEP到OAA的代谢通量增加;pyccgP458S基因的过表达能将部分PYR转化为OAA,使副产物乙酸的积累显著下降,当分别过表达ppc和pyccgP458S基因时,高丝氨酸摇瓶产量为5.5 g/L和4.4 g/L,将两个基因进行叠加时,高丝氨酸产量提高至5.9 g/L[20]。
乙醛酸循环途径作为TCA循环中的回补途径,可以将异柠檬酸(isocitric acid, ICL) 直接转化成延胡索酸(fumaric acid, FUM) 和苹果酸(MAL),加快中心碳代谢到目标产物的转化。IclR (异柠檬酸裂解酶抑制因子,由iclR编码) 是乙醛酸循环途径中的转录调控因子,该因子会抑制途径中相关基因的表达,敲除iclR基因可以解除乙醛酸循环途径关键基因的转录负调控作用[3, 40]。为了将更多的碳代谢通量引入L-天冬氨酸合成途径中,本团队对iclR基因进行了敲除。发酵结果显示敲除iclR基因的菌株生长并未受到影响,这表明敲除iclR基因没有对菌株的TCA循环造成负面影响,而解除乙醛酸循环途径关键基因的转录负调控作用后,该循环增强,TCA循环中的无效循环减弱,提高了关键前体OAA的供应,从而使菌株合成L-高丝氨酸的水平提高[11]。
2.2.2 天冬氨酸前体的供应L-天冬氨酸是大肠杆菌中L-高丝氨酸及其衍生物生物合成的关键前体。L-天冬氨酸可由3个酶催化的三步反应合成L-高丝氨酸,第一步反应由L-天冬氨酸转化生成L-天冬氨酸磷酸,该反应由3种天冬氨酸激酶同工酶催化,即天冬氨酸激酶Ⅰ、Ⅱ、Ⅲ (AKI、AKⅡ和AKⅢ),分别由基因thrA、metL和lysC编码,这3种酶的活性分别受到3种不同代谢支路产物的抑制,但在这3种同工酶中,AKI的胞内含量远高于其他两种酶[41-42]。第二步反应由L-天冬氨酸磷酸转化生成L-天冬氨酸-β-半醛,该步骤由天冬氨酸半醛脱氢酶(Asd,由asd基因编码) 催化完成,所需辅酶为还原型辅酶Ⅱ (NADPH)。第三步反应由L-天冬氨酸-β-半醛进一步转化生成L-高丝氨酸,该步骤由高丝氨酸脱氢酶(由thrA、metL编码) 催化完成,所需辅酶也是NADPH,同时,L-天冬氨酸-β-半醛还会被二氨基二庚酸脱羧酶(由lysA基因编码) 催化生成副产物L-赖氨酸。以上酶催化反应中,基因thrA和thrB、thrC属于同一个操纵子,即苏氨酸操纵子,由同一个启动子转录,它的转录受到先导序列thrL的调节。因此理论上,L-高丝氨酸的产率应随L-天冬氨酸碳通量的增加而增加。
在大肠杆菌中,L-天冬氨酸可以通过天冬氨酸氨基转移酶AspC (由aspC基因编码) 催化的转氨作用由OAA形成,或者通过天冬氨酸氧化酶AspA (由aspA基因编码) 催化的胺化作用由FUM转化合成[43-44]。AspC催化的反应通常用于生产L-天冬氨酸衍生的产物,该反应与α-酮戊二酸(α-KG) 的还原相结合产生L-谷氨酸[43, 45-46]。L-天冬氨酸通过固定1 mol CO2后由AspC途径获得,可以得到L-高丝氨酸的最高理论得率2 mol/mol葡萄糖,最高糖酸转化率为1.32 g/g葡萄糖,其化学反应方程如式(1) 所示[22]。而通过分析发现,以该路径获得最高理论得率2 mol L-高丝氨酸需要消耗6 mol NADPH,远高于1 mol葡萄糖代谢所产生的还原力(2 mol NADH),因此该路径存在严重的还原力不足问题,从而导致L-高丝氨酸无法达到最高理论得率。

|
(1) |
与上述途径相比,AspA催化的反应被认为是AspC催化的有效替代反应。葡萄糖经过代谢最终生成乙酰-CoA后进入TCA循环,产生的FUM通过AspA途径得到L-天冬氨酸,该途径L-高丝氨酸理论得率为1 mol/mol葡萄糖,理论糖酸转化率为0.66 g/g葡萄糖,其化学反应方程见式(2)[22]。

|
(2) |
通过分析发现在该途径中消耗2 mol NADPH,可生成5 mol NADH和1 mol FADH2,还原力大量剩余。因此,如果将上述两条途径结合,用AspA途径生成的还原力弥补AspC途径还原力的不足,则可以获得相对平衡的还原力,是理想的L-高丝氨酸合成途径,其化学反应方程如式(3) 所示[22]。

|
(3) |
在该反应中没有碳原子损失,L-高丝氨酸的理论得率是1.5 mol/mol葡萄糖,糖酸转化率为0.99 g/g葡萄糖。
目前文献中对前体L-天冬氨酸的合成途径已经做了大量研究[45, 47]。Zhang等为了增加L-天冬氨酸的供应,将Ptrc驱动的aspA基因整合至基因组中,菌株的L-高丝氨酸产量提高了58.6%[23]。Piao等通过TCA循环的还原分支设计了一株大肠杆菌来合成L-天冬氨酸和β-丙氨酸,在底盘菌中过表达ppc基因来强化PEP羧化酶,增加了前体OAA的供应,通过过表达aspC基因固定CO2,并通过氧化还原与谷氨酸脱氢酶(GdhA) 平衡,驱动α-KG和L-谷氨酸循环并再生NADPH,最终可得到L-高丝氨酸最高理论产量2 mol/mol葡萄糖[48]。研究表明,在分别过表达aspC和aspA基因时,L-高丝氨酸产率分别为0.42 g/(L·h) 和0.94 g/(L·h),在同时过表达aspC和aspA基因后,可平衡两条途径中的还原力,使L-高丝氨酸生产效率提高至1.69 g/(L·h),并产生81.2 g/L的L-高丝氨酸[22]。
2.2.3 代谢副产物调控L-高丝氨酸在野生型大肠杆菌中不易积累,需要通过阻断或弱化竞争及降解途径使L-高丝氨酸在菌株发酵过程中得到积累[49]。天冬氨酸-β-半醛是生物合成L-赖氨酸和L-高丝氨酸的中间体,因此,L-赖氨酸的生物合成会与L-高丝氨酸的合成竞争碳通量。同时,L-高丝氨酸作为代谢中间体,也是L-甲硫氨酸和L-苏氨酸的共同前体[50]。通过敲除或弱化编码高丝氨酸琥珀酰转移酶的metA基因以及编码高丝氨酸激酶和苏氨酸合成酶的thrBC基因可以减少L-高丝氨酸的进一步代谢,提高其合成效率。Li等将敲除lysA和thrBC基因的大肠杆菌W3110进行培养,发现当无外加必需氨基酸时,菌株无法生长,表明基因敲除成功,在培养基中添加L-赖氨酸、L-苏氨酸以及L-甲硫氨酸后,发现有少量L-高丝氨酸的积累,进一步对L-高丝氨酸的运输系统进行了改造,并通过质粒pBR322过表达代谢途径中的metL基因,在15 L发酵罐中发酵得到39.54 g/L的L-高丝氨酸,糖酸转化率达到0.29 g/g葡萄糖[25]。本团队在构建初始菌株时,成功敲除了lysA和thrB基因,构建获得了L-高丝氨酸合成菌株,摇瓶产量从1.85 g/L提高到2.01 g/L,细胞干重为0.33 g/g CDW[21]。
由上述研究可知,敲除副产物氨基酸合成途径虽然可以提高目标氨基酸的产量,但所构建菌株大都为营养缺陷型,需要在发酵期间额外添加必需氨基酸以保证菌体的正常生长,这无疑增加了发酵成本,也会对氨基酸的发酵调控过程造成一定的困难[51]。因此,除了敲除副产物氨基酸代谢途径外,采用启动子工程将原始启动子静态弱化或动态调控也是目前重要的研究方向。Zhang等在以野生型大肠杆菌W3110为底盘菌株构建L-高丝氨酸生产菌株时,为不破坏thrABC操纵子的天然结构并实现thrB和thrC的弱化,将该操纵子的启动子替换为不同强度的弱启动子,但发现启动子的替换同时会引起L-高丝氨酸合成关键基因thrA的弱化,故在出发菌株基因组中先整合1个拷贝强启动子Ptrc调控的thrAC1034T (解除L-苏氨酸反馈抑制)。发酵结果表明,菌株在不添加L-苏氨酸的培养基中能够生长,说明thrABC启动子替换为弱启动子未造成其L-苏氨酸缺陷,并且有效降低了thrBC的转录,进行摇瓶发酵积累了1.5 g/L L-高丝氨酸[52]。
动态调控的方式有人工诱导的动态调控和细胞自主诱导的动态调控。人工诱导的动态调控是指在人工控制的物理或化学信号(如光、温度和化学诱导剂等诱导因子) 存在下,利用响应该信号的启动子等元件调控代谢途径下游基因的表达[53]。目前在酶制剂、有机酸和氨基酸等的代谢合成过程中均有广泛地应用[54-55]。人工诱导的动态调控虽操作方便,可以实时控制,但也存在一定的缺陷,如在封闭或无氧系统中,由于大范围细胞受光不均匀,光诱导强度不均一和添加诱导剂为细胞非吸收物质时,会使诱导持续进行,导致反应不可逆或损害菌体。细胞自主诱导的动态调控有响应胞内代谢物的启动子、转录因子和核糖核酸开关以及群体感应系统。Long等设计了群体感应基因线路动态调控α-酮戊二酸脱氢酶(由sucAB基因编码) 活性,以PesaS启动子取代sucA基因的天然启动子。不存在高丝氨酸内酯(acylhomoserine lactone,AHL) 时,PesaS启动子可与转录调节子EsaRI70V结合,触发该启动子引导的目的基因转录,AHL含量随着细胞浓度的增加而增加,当AHL浓度达到一定阈值可与转录调节子EsaRI70V结合,阻碍PesaS与转录调节因子结合,抑制目的基因转录,进而用于调节α-酮戊二酸脱氢酶活性,改造后的菌株经补料分批发酵后的反式-4-羟基-L-脯氨酸产量达43.2 g/L,为对照菌的2.75倍[56]。
在大肠杆菌中,OSH由metA编码的高丝氨酸琥珀酰基转移酶专一性催化合成,包括高丝氨酸-γ-羟基的激活和琥珀酰-CoA的酰基化过程。在此过程中若无碳原子损失,则最高理论转化率为0.75 mol/mol葡萄糖,最高理论糖酸转化率为0.91 g/g葡萄糖。但在其合成过程中,存在多条副产物合成途径,从而降低其理论转化率。例如,L-苏氨酸的合成会与OSH竞争前体物质L-高丝氨酸,因此有必要阻断或降低L-苏氨酸的代谢通量。此外,由metB基因编码的胱硫醚-γ-合成酶可以将OSH分解生成半胱氨酸,通过敲除metB基因可以阻断OSH的降解途径。琥珀酰-CoA是合成OSH的重要底物,高丝氨酸琥珀酰基转移酶(MetA) 首先激活L-高丝氨酸上的羟基,再将琥珀酰基基团转移至被激活的L-高丝氨酸上,最终合成OSH。在TCA循环中,由sucAB基因编码的α-酮戊二酸脱氢酶可将α-KG转化成琥珀酰-CoA,进一步由sucCD基因编码的琥珀酰-CoA合酶将琥珀酰-CoA降解为琥珀酸,sucAB和sucCD是影响琥珀酰-CoA合成过程中重要的基因[57]。据报道,单独敲除sucD基因能够增加胞内琥珀酰-CoA的含量,用于提高己二酸的产量[58]。本团队以L-高丝氨酸生产菌株为底盘菌株,通过敲除thrB基因来阻断L-苏氨酸的生物合成,敲除metB阻断OSH的降解,构建OSH高产菌株,通过发酵优化使OSH产量达到102.5 g/L[27]。同时,本团队研究了前体琥珀酰-CoA对OSH生产的影响,发现敲除sucD基因和过表达sucA基因,虽然增加了前体琥珀酰-CoA的供应,但该操作阻断了菌体的中心代谢TCA循环,严重影响了菌体的生长,即使发酵过程中补加了琥珀酸,但仍然使OSH产量降低[28]。
2.3 代谢途径中反馈抑制的解除在大肠杆菌中,参与L-高丝氨酸代谢合成的多数基因都会受到其终端代谢产物的反馈调控。如图 2所示,L-高丝氨酸代谢途径中的thrA、metL、lysC、asd和metA基因的转录受转录因子MetJ的负调控作用[59];关键基因thrA的转录水平受到L-异亮氨酸与L-苏氨酸的协同反馈阻遏调控[60];基因lysC的转录水平受到L-赖氨酸的反馈阻遏调控;基因metL的转录水平受到L-甲硫氨酸的反馈阻遏调控;基因asd的转录水平受到L-赖氨酸、L-甲硫氨酸和L-苏氨酸3个氨基酸的共价反馈阻遏调控。分子生物学的发展使得在分子水平上研究氨基酸生物合成的代谢调控成为可能,thrA和lysC的反馈抑制表达的机制已在大肠杆菌遗传水平上得到解释[61],反馈抑制天冬氨酸激酶Ⅲ的机制,已经在其三维蛋白质结构上得到阐明[62]。这些信息可作为合理酶修饰的理论基础,使用遗传方法进行酶的变构调节,使得它们失去对抑制剂的敏感性,同时它们的活性不受位点修饰的影响甚至可以增强其活性。在染色体上,由于thrABC这3个基因相邻排列组成了thrABC操纵子,thrBC是L-苏氨酸合成的关键基因。苏氨酸的过量积累会反馈抑制AK I的活性,其抑制机制是变构的并且与L-天冬氨酸竞争,该酶的表达还受到过量积累的L-异亮氨酸的协同阻遏;L-甲硫氨酸的过量积累会反馈抑制AK Ⅱ的活性;L-赖氨酸的过量积累会反馈抑制AK Ⅲ的活性,抑制作用是完全的,其抑制机制是非竞争性和协同作用。生物信息学分析表明,AK Ⅲ可能是AK I和AK Ⅱ的原型[63]。高丝氨酸脱氢酶I也被L-苏氨酸部分抑制,但抑制机制是非竞争性的[60]。此外,AK Ⅲ和高丝氨酸脱氢酶I含有相同的区域,L-赖氨酸和L-苏氨酸对高丝氨酸脱氢酶I均具有一定的抑制作用[64]。在获得AK I和高丝氨酸脱氢酶I的高分辨率结构之前,抑制机制仍然不清楚。在代谢改造过程中,可以通过突变编码酶的基因实现其抗反馈抑制作用[65]。

|
| 图 2 L-天冬氨酸到L-高丝氨酸关键基因的反馈抑制 Fig. 2 Feedback inhibition of key genes involved in the reactions converting L-aspartic acid into L-homoserine. FUM: fumaric acid; ASP: L-aspartic acid; ASP-P: aspartyl-phosphate; ASP-SA: L-aspartate-4-semialdehyde; HS: homoserine; OSH: O-succinyl-L-homoserine; OAA: oxaloacetic acid; LYS: lysine; THR: threonine; ISO: isoleucine; OAH: O-acetyl-L-homoserine; MET: methionine; MetJ: MetJ family transcriptional regulator; aspA: encoding aspartate oxidase; aspC: encoding aspartate aminotransferase; lysC: encoding aspartate kinase; metL: encoding bifunctional aspartokinase/homoserine dehydrogenase; thrA: encoding aspartokinase I and homoserine dehydrogenase I; asd: encoding aspartate-semialdehyde dehydrogenase; metX: encoding homoserine-O-acetyltransferase; metA: encoding homoserine-O-succinyltransferase; metB: encoding O-succinylhomoserine lyase. |
| |
本团队对编码抗反馈抑制AK I的lysCcgC932T基因和编码抗反馈抑制AK Ⅲ和高丝氨酸脱氢酶I融合蛋白的thrAC1034T基因进行了过表达,以提高产物L-高丝氨酸的合成速率,发酵产量从5.9 g/L提高到8.6 g/L,且乙酸、草酰乙酸和α-酸酮戊二酸的积累下降[20]。Kim等使用随机突变和代谢工程等组合策略,已经构建了一系列能够高产OSH的菌株[66]。Kim等以E. coli W3110为底盘菌,通过敲除metB、thrB和metJ和过表达抗反馈抑制的metAfbr基因后,获得OSH摇瓶发酵产量为1.8 g/L[67]。Hong等以L-苏氨酸高产菌株E. coli FTR2533为出发菌株,在阻断OSH的降解途径后,通过过表达metA和抗反馈抑制的thrAfbr基因,增强了L-天冬氨酸的代谢途径,最终在含蔗糖培养基的1 L发酵罐中发酵25 h后得到100 g/L的OSH[1]。
E. coli W3110细胞本身不含有高丝氨酸乙酰转移酶MetX,研究者将外源metX基因转入产L-高丝氨酸菌株中,最终得到产OAH菌株,但metX受到多水平调控机制的严格控制[68-69]。例如,metX在谷氨酸棒杆菌中的表达被转录调节因子McbR严格下调,而其活性被S-腺苷甲硫氨酸和L-半胱氨酸抑制[69]。更重要的是,MetX的活性受到最终产物OAH的强烈反馈抑制,极大地限制了OAH的生物合成[70]。因此,寻找一种高效的MetX对大肠杆菌中OAH的合成至关重要。对酶进行理性设计或半理性设计以改善其热稳定性、增加活性和改进选择性,进而提高目标产物产量是目前研究的常用方法[71-73]。Kase等分离出了谷氨酸棒状杆菌L-甲硫氨酸类似物耐受突变体,该突变体可产生10.5 g/L的OAH[74]。Willke等利用代谢工程技术在E. coli中阻断了OAH的降解途径并引入不受反馈调控的metX基因,利用葡萄糖在摇瓶培养条件下,发酵得到6.5 g/L的OAH,该菌株在补料分批培养条件下产生55 g/L的OAH[75]。Wei等首先通过敲除metB、thrB和metA等基因,并进一步过表达反馈不敏感的thrA基因,重建了OAH代谢途径,在摇瓶发酵48 h后产生了1.68 g/L的OAH,并发现有高丝氨酸、赖氨酸以及乙酸等副产物的积累,后通过敲除lysA基因,OAH产量提高了33.93%,进一步通过不同的人工启动子调节元件调控ppc基因的表达,过表达反馈不敏感的lysCcg基因,使OAH产量提高到4.61 g/L,另外有2.44 g/L的高丝氨酸积累,副产物乙酸降低了43.2%。最后通过对迈氏钩端螺旋体(Leptospira meyeri) 来源的MetX进行酶分子改造并结合OAH代谢途径的改造,最终在7.5 L发酵罐中经过60 h的葡萄糖补料发酵得到62.7 g/L的OAH,糖酸转化率达到0.45 g/g葡萄糖,是目前报道的OAH发酵产量最高的菌株,也是补料分批发酵中糖酸转化率最高的菌株[30]。
本团队在改造OAH发酵菌株的过程中,对途径中thrA基因进行脱敏,解除了途径中L-异亮氨酸与L-苏氨酸的协同反馈阻遏调控,并结合关键酶MetX的代谢改造和调控,使OAH摇瓶发酵产量从原来的6.24 g/L提高至13.18 g/L,糖酸转化率提高至0.33 g/g葡萄糖[76]。若不考虑碳原子损失,OAH的最高转化率为1 mol/mol葡萄糖,理论糖酸转化率为0.89 g/g葡萄糖,但由于整体代谢过程中还原力平衡、能量供应、副产物合成等问题的存在,使糖酸转化率远未达到理论水平。
2.4 辅酶NADPH的循环供应在胞内代谢反应的过程中,辅酶NADPH/ NADP+等是胞内能量传递的重要物质,为细胞生物合成与分解反应提供氧化还原载体,因此控制胞内辅酶平衡是维持细胞正常代谢,实现目标代谢物高效生产的重要前提[77-79]。
在L-高丝氨酸及其衍生物合成过程中,高丝氨酸脱氢酶和天冬氨酸半醛脱氢酶催化的反应需要辅酶NADPH[52]。研究证明,过表达吡啶核苷酸转氢酶编码基因(pntAB) 可有效促进NADPH的再生,已成功应用于L-赖氨酸、L-缬氨酸等氨基酸的代谢改造中,提高目标代谢物的产量[80-82]。如图 3所示,与NADPH生物合成相关的基因有zwf、fpr、fabI、pntAB和sthA等,研究表明过表达pntAB可显著提高大肠杆菌生产L-高丝氨酸的能力[22-23, 52]。PntAB酶活性的提高可以显著增加NADPH的代谢通量,但在代谢过程中酰基-ACP/CoA会对PntAB酶产生反馈抑制,因此,研究学者尝试动态调控fabI基因以降低烯酰基-ACP还原酶的表达水平,从而减轻该抑制提高NADPH通量[84-85]。由udhA基因编码的可溶性反氢酶会消耗代谢过程中所产生的NADPH,因此,降低UdhA酶表达水平可以增加NADPH通量[85]。如图 3所示,降低zwf基因编码的葡萄糖-6-磷酸脱氢酶水平会激活SoxRS调控的NADPH库,并增加丙酮酸铁氧还蛋白氧化还原酶(YdbK) 的表达,导致乙酰-CoA通量增加[86]。此外,通过YdbK氧化丙酮酸生成的还原铁氧还蛋白可用于还原NADP+ (通过fpr基因编码的铁氧还蛋白NADP+还原酶的作用),这可使NADPH通量增加[87-90]。Li等构建了ydbK和fpr缺失菌株,结果表明ydbK和fpr的敲除,确实会引起NADPH通量升高[87]。中国科学院微生物研究所于波课题组系统地分析了大肠杆菌中L-高丝氨酸的代谢网络,设计了一条由葡萄糖发酵高产L-高丝氨酸的辅酶平衡路线。采用乙醛酸循环途径作为TCA循环的回补途径,提高延胡索酸到L-天冬氨酸的通量,并以此满足代谢过程中的氧化还原平衡,进一步通过微调途径中aspA、aspC和pntAB基因的表达来平衡辅酶供应,构建了一株L-高丝氨酸高产菌株,在补料分批发酵中L-高丝氨酸产量达到84.1 g/L,生产效率和糖酸转化率分别达到1.96 g/(L·h) 和0.50 g/g,是目前报道的L-高丝氨酸最高发酵水平[22]。综合上述研究可知,在L-高丝氨酸及其衍生物细胞工厂构建过程中,胞内氧化还原平衡是必须要考虑的重要问题,而其中辅酶NADPH的再生也是影响目标产物合成的关键,新型辅酶再生基因的挖掘和应用也是该领域研究的重要方向。
为了防止目标产物在细胞内大量积累,出现产物抑制,氨基酸产物必须迅速运出细胞,目标产物转运系统的改造可以有效增加目标产物的产量[91]。1974年,Templeton等首次证明L-高丝氨酸和L-苏氨酸是通过同一个系统运输的,然而,这2种氨基酸的转运体在当时并没有被鉴定出来,随着对氨基酸转运系统研究的不断深入,在大肠杆菌中至少发现了3种L-苏氨酸转运体[92]。1990年,在大肠杆菌中发现了L-苏氨酸转运系统由tdcC基因编码[93]。Sumantran等对比tdcC基因失活和过表达情况发现,当tdcC基因失活后,L-高丝氨酸的利用率由13.78 mg/(L·h·g DCW)下降至10.55 mg/(L·h·g DCW),而过表达tdcC基因时,L-高丝氨酸的利用率提高至18.34 mg/(L·h·g DCW)[93],上述结果表明TdcC转运蛋白在大肠杆菌L-高丝氨酸摄取中具有重要作用。Livshits和Kruse等发现了两个与L-苏氨酸同源的L-高丝氨酸输出基因rhtA和rhtB[94-95],RhtA被认为是药物/代谢物转运蛋白超家族的成员,而RhtB属于RhtB转运蛋白超家族成员[96],研究结果表明rhtA基因的过表达和rhtA23基因的突变提高了L-高丝氨酸的输出能力,显著降低了细胞内L-高丝氨酸积累,使菌株L-高丝氨酸生产能力和细胞生长速率均得到显著提高[25, 94, 97]。Zakataeva等在重组大肠杆菌NZ10中过表达rhtB基因,提高L-高丝氨酸输出能力,弥补了L-高丝氨酸高产菌株有限的产物外排能力,使菌株在48 h积累10.6 g/L的L-高丝氨酸[98]。Zhang等构建了过表达质粒pTrc99A-rhtA,摇瓶发酵L-高丝氨酸产量提高60.2%,达到12.5 g/L,生物量提高12.6%,表明增强L-高丝氨酸输出可有效提高其产量并增强菌株对L-高丝氨酸的抗性[52]。本研究团队研究表明增强rhtA和eamA基因(编码丝氨酸外运蛋白) 的表达都可以提高菌株生产L-高丝氨酸的能力[11]。
谷氨酸棒杆菌中已发现有多种氨基酸输出系统,包括L-赖氨酸输出系统(LysE)、L-苏氨酸输出系统(ThrE) 和支链氨基酸双组分输出系统(BrnFE)[99]。这些系统在输出相应的氨基酸时消耗大量的能量,而且需要调节因子的调节。LysE蛋白的生物合成受LysG因子调控[100],而BrnFE则负责L-甲硫氨酸和支链氨基酸(L-缬氨酸、L-亮氨酸和L-异亮氨酸) 的输出,受Lrp家族的调控因子调控[101]。Li等发现编码双组分输出系统的brnFE操纵子负责L-高丝氨酸的输出,在C. glutamicum ATCC13032菌株中过表达brnFE后,L-高丝氨酸产量提高了2倍[26]。于波课题组在前期研究的基础上采用Ptac启动子驱动rhtB基因,促进L-高丝氨酸的外排,发现其效果并不明显,进一步过表达来源于C. glutamicum ATCC13032的brnFE基因后,发现该菌株在含有高浓度L-高丝氨酸的LB培养基中细胞生物量增加了335%,表明该基因对L-高丝氨酸耐受性具有显著的影响[22]。
综上所述,转运蛋白是生物合成L-高丝氨酸及其衍生物的研究热点之一,目前已发现了若干L-高丝氨酸的外运蛋白,包括RhtA、RhtB、EamA和BrnFE等,这些转运蛋白对L-高丝氨酸产量的提高有着重要的意义,也为解决高浓度L-高丝氨酸胁迫下如何提高产量和缩短发酵时间等问题指明了方向。
3 总结与展望L-高丝氨酸及其衍生物(O-乙酰-L-高丝氨酸和O-琥珀酰-L-高丝氨酸) 是合成多种C4化合物的前体,并可以合成含硫氨基酸(L-甲硫氨酸) 与含磷氨基酸(L-草铵膦),也与微生物细胞内多种必需氨基酸的合成相关联,是重要的工业平台化学品。本文综述了代谢工程改造大肠杆菌高效生产L-高丝氨酸及其衍生物的生物合成途径和代谢改造策略,并结合本实验室前期的研究工作阐明了高丝氨酸及其衍生物合成途径中关键途径改造后的效果,为提高L-高丝氨酸及其衍生物的生产效率,下游产品的延伸奠定了一定的研究基础。
氨基酸的微生物合成系统是一个精密的细胞工厂,目标产物的合成是由整个代谢网络中多种酶的协同作用实现的。在现有研究基础上可以结合代谢网络分析、转录组学分析和代谢通量分析等技术对细胞的L-高丝氨酸及其衍生物的代谢合成途径进行全局分析和优化,找到不同代谢通路之间的联系,进而理性地设计细胞代谢通路,从而协调目标产物积累与中间产物生成、目标产物积累与菌体生长之间的关系,进一步提高菌体L-高丝氨酸及其衍生物的生产性能。我们相信,随着代谢工程技术的不断发展,L-高丝氨酸及其衍生物将在微生物细胞中实现高效生物合成和工业化生产。
| [1] |
Hong KK, Kim JH, Yoon JH, et al. O-succinyl-L-homoserine-based C4-chemical production: succinic acid, homoserine lactone, γ-butyrolactone, γ-butyrolactone derivatives, and 1, 4-butanediol. J Ind Microbiol Biotechnol, 2014, 41(10): 1517-1524. DOI:10.1007/s10295-014-1499-z
|
| [2] |
Ferla MP, Patrick WM. Bacterial methionine biosynthesis. Microbiology (Reading), 2014, 160(Pt 8): 1571-1584.
|
| [3] |
Song CW, Kim JW, Cho IJ, et al. Metabolic engineering of Escherichia coli for the production of 3-hydroxypropionic acid and malonic acid through β-alanine route. ACS Synth Biol, 2016, 5(11): 1256-1263. DOI:10.1021/acssynbio.6b00007
|
| [4] |
Liu ZQ, Dong SC, Yin HH, et al. Enzymatic synthesis of an ezetimibe intermediate using carbonyl reductase coupled with glucose dehydrogenase in an aqueous-organic solvent system. Bioresour Technol, 2017, 229: 26-32. DOI:10.1016/j.biortech.2016.12.098
|
| [5] |
Liu ZQ, Wu L, Zhang XJ, et al. Directed evolution of carbonyl reductase from Rhodosporidium toruloides and its application in stereoselective synthesis of tert-butyl(3R, 5S)-6-chloro-3, 5-dihydroxyhexanoate. J Agric Food Chem, 2017, 65(18): 3721-3729. DOI:10.1021/acs.jafc.7b00866
|
| [6] |
Liu ZQ, Lu MM, Zhang XH, et al. Significant improvement of the nitrilase activity by semi-rational protein engineering and its application in the production of iminodiacetic acid. Int J Biol Macromol, 2018, 116: 563-571. DOI:10.1016/j.ijbiomac.2018.05.045
|
| [7] |
Liu ZQ, Wu L, Zheng L, et al. Biosynthesis of tert-butyl(3R, 5S)-6-chloro-3, 5-dihydroxyhexanoate by carbonyl reductase from Rhodosporidium toruloides in mono and biphasic media. Bioresour Technol, 2018, 249: 161-167. DOI:10.1016/j.biortech.2017.09.204
|
| [8] |
Zheng YG, Yin HH, Yu DF, et al. Recent advances in biotechnological applications of alcohol dehydrogenases. Appl Microbiol Biotechnol, 2017, 101(3): 987-1001. DOI:10.1007/s00253-016-8083-6
|
| [9] |
战俊杰, 杜丽红. 代谢工程改造大肠杆菌生产L-色氨酸的研究进展. 山东化工, 2021, 50(1): 85-87, 89. Zhan JJ, Du LH. Metabolic engineering for L-tryptophan production in Escherichia coli. Shandong Chem Ind, 2021, 50(1): 85-87, 89 (in Chinese). DOI:10.3969/j.issn.1008-021X.2021.01.034 |
| [10] |
Park JH, Oh JE, Lee KH, et al. Rational design of Escherichia coli for L-isoleucine production. ACS Synth Biol, 2012, 1(11): 532-540. DOI:10.1021/sb300071a
|
| [11] |
刘鹏. 生物合成L-高丝氨酸及其衍生物的细胞工厂构建[D]. 杭州: 浙江工业大学, 2020. Liu P. Construction of cell factories for bioproduction of L-homoserine and its derivatives[D]. Hangzhou: Zhejiang University of Technology, 2020 (in Chinese). |
| [12] |
Minh DP, Besson M, Pinel C, et al. Aqueous-phase hydrogenation of biomass-based succinic acid to 1, 4-butanediol over supported bimetallic catalysts. Top Catal, 2010, 53(15/16/17/18): 1270-1273.
|
| [13] |
Walther T, Calvayrac F, Malbert Y, et al. Construction of a synthetic metabolic pathway for the production of 2, 4-dihydroxybutyric acid from homoserine. Metab Eng, 2018, 45: 237-245. DOI:10.1016/j.ymben.2017.12.005
|
| [14] |
Nielsen J, Keasling JD. Engineering cellular metabolism. Cell, 2016, 164(6): 1185-1197. DOI:10.1016/j.cell.2016.02.004
|
| [15] |
Lee JH, Wendisch VF. Production of amino acids-genetic and metabolic engineering approaches. Bioresour Technol, 2017, 245: 1575-1587. DOI:10.1016/j.biortech.2017.05.065
|
| [16] |
Hirasawa T, Shimizu H. Recent advances in amino acid production by microbial cells. Curr Opin Biotechnol, 2016, 42: 133-146. DOI:10.1016/j.copbio.2016.04.017
|
| [17] |
Iranmanesh E, Asadollahi MA, Biria D. Improving L-phenylacetylcarbinol production in Saccharomyces cerevisiae by in silico aided metabolic engineering. J Biotechnol, 2020, 308: 27-34. DOI:10.1016/j.jbiotec.2019.11.008
|
| [18] |
Born TL, Franklin M, Blanchard JS. Enzyme-catalyzed acylation of homoserine: mechanistic characterization of the Haemophilus influenzae met2-encoded homoserine transacetylase. Biochemistry, 2000, 39(29): 8556-8564. DOI:10.1021/bi000462p
|
| [19] |
Zhong WQ, Zhang Y, Wu WJ, et al. Metabolic engineering of a homoserine-derived non-natural pathway for the de novo production of 1, 3-propanediol from glucose. ACS Synth Biol, 2019, 8(3): 587-595. DOI:10.1021/acssynbio.9b00003
|
| [20] |
张博, 姚臻豪, 柳志强, 等. 代谢工程改造大肠杆菌生产L-高丝氨酸. 生物工程学报, 2021, 37(4): 1287-1297. Zhang B, Yao ZH, Liu ZQ, et al. Metabolic engineering of Escherichia coli for L-homoserine production. Chin J Biotech, 2021, 37(4): 1287-1297 (in Chinese). DOI:10.13345/j.cjb.200434 |
| [21] |
Liu P, Zhang B, Yao ZH, et al. Multiplex design of the metabolic network for production of L-homoserine in Escherichia coli. Appl Environ Microbiol, 2020, 86(20): e01477-20.
|
| [22] |
Mu QX, Zhang SS, Mao XJ, et al. Highly efficient production of L-homoserine in Escherichia coli by engineering a redox balance route. Metab Eng, 2021, 67: 321-329. DOI:10.1016/j.ymben.2021.07.011
|
| [23] |
Zhang Y, Wei MH, Zhao GH, et al. High-level production of L-homoserine using a non-induced, non-auxotrophic Escherichia coli chassis through metabolic engineering. Bioresour Technol, 2021, 327: 124814. DOI:10.1016/j.biortech.2021.124814
|
| [24] |
Liu M, Lou JL, Gu JL, et al. Increasing L-homoserine production in Escherichia coli by engineering the central metabolic pathways. J Biotechnol, 2020, 314/315: 1-7. DOI:10.1016/j.jbiotec.2020.03.010
|
| [25] |
Li H, Wang BS, Zhu LH, et al. Metabolic engineering of Escherichia coli W3110 for L-homoserine production. Process Biochem, 2016, 51(12): 1973-1983. DOI:10.1016/j.procbio.2016.09.024
|
| [26] |
Li N, Xu S, Du GC, et al. Efficient production of L-homoserine in Corynebacterium glutamicum ATCC 13032 by redistribution of metabolic flux. Biochem Eng J, 2020, 161: 107665. DOI:10.1016/j.bej.2020.107665
|
| [27] |
Zhu WY, Niu K, Liu P, et al. Enhanced O-succinyl-L-homoserine production by recombinant Escherichia coli ΔIJBB*Trc-metL/pTrc-metAfbr-Trc- thrAfbr-yjeH via multilevel fermentation optimization. J Appl Microbiol, 2021, 130(6): 1960-1971. DOI:10.1111/jam.14884
|
| [28] |
Liu P, Liu JS, Zhu WY, et al. O-Succinyl-L-homoserine overproduction with enhancement of the precursor succinyl-CoA supply by engineered Escherichia coli. J Biotechnol, 2021, 325: 164-172. DOI:10.1016/j.jbiotec.2020.11.002
|
| [29] |
Huang JF, Zhang B, Shen ZY, et al. Metabolic engineering of E. coli for the production of O-succinyl-L-homoserine with high yield. 3 Biotech, 2018, 8(7): 1-9.
|
| [30] |
Wei L, Wang Q, Xu N, et al. Combining protein and metabolic engineering strategies for high-level production of O-acetylhomoserine in Escherichia coli. ACS Synth Biol, 2019, 8(5): 1153-1167. DOI:10.1021/acssynbio.9b00042
|
| [31] |
Liu P, Liu JS, Zhang B, et al. Increasement of O-acetylhomoserine production in Escherichia coli by modification of glycerol-oxidative pathway coupled with optimization of fermentation. Biotechnol Lett, 2021, 43(1): 105-117. DOI:10.1007/s10529-020-03031-8
|
| [32] |
Li N, Zeng WZ, Zhou JW, et al. O-Acetyl-L-homoserine production enhanced by pathway strengthening and acetate supplementation in Corynebacterium glutamicum. Biotechnol Biofuels Bioprod, 2022, 15(1): 27. DOI:10.1186/s13068-022-02114-0
|
| [33] |
Escalante A, Salinas Cervantes A, Gosset G, et al. Current knowledge of the Escherichia coli phosphoenolpyruvate-carbohydrate phosphotransferase system: peculiarities of regulation and impact on growth and product formation. Appl Microbiol Biotechnol, 2012, 94(6): 1483-1494. DOI:10.1007/s00253-012-4101-5
|
| [34] |
Gosset G. Improvement of Escherichia coli production strains by modification of the phosphoenolpyruvate: sugar phosphotransferase system. Microb Cell Fact, 2005, 4(1): 14. DOI:10.1186/1475-2859-4-14
|
| [35] |
Mu Y, Teng H, Zhang DJ, et al. Microbial production of 1, 3-propanediol by Klebsiella pneumoniae using crude glycerol from biodiesel preparations. Biotechnol Lett, 2006, 28(21): 1755-1759. DOI:10.1007/s10529-006-9154-z
|
| [36] |
柳志强, 刘鹏, 张博, 等. 一种产O-乙酰L-高丝氨酸菌株发酵方法: CN112094872A. 2020-12-18. Liu ZQ, Liu P, Zhang B, et al. The invention relates to a ermentation method for O-acetyl-L-homoserine strain: CN112094872A. 2020-12-18 (in Chinese). |
| [37] |
Sauer U, Eikmanns BJ. The PEP—pyruvate—oxaloacetate node as the switch point for carbon flux distribution in bacteria. FEMS Microbiol Rev, 2005, 29(4): 765-794. DOI:10.1016/j.femsre.2004.11.002
|
| [38] |
Li YJ, Wei HB, Wang T, et al. Current status on metabolic engineering for the production of L-aspartate family amino acids and derivatives. Bioresour Technol, 2017, 245: 1588-1602. DOI:10.1016/j.biortech.2017.05.145
|
| [39] |
Somasundaram S, Jeong J, Irisappan G, et al. Enhanced production of malic acid by co-localization of phosphoenolpyruvate carboxylase and malate dehydrogenase using synthetic protein scaffold in Escherichia coli. Biotechnol Bioprocess Eng, 2020, 25(1): 39-44. DOI:10.1007/s12257-019-0269-1
|
| [40] |
Lee KH, Park JH, Kim TY, et al. Systems metabolic engineering of Escherichia coli for L-threonine production. Mol Syst Biol, 2007, 3: 149. DOI:10.1038/msb4100196
|
| [41] |
赵慧. 代谢工程改造大肠杆菌生产L-苏氨酸[D]. 无锡: 江南大学, 2018. Zhao H. Metabolic engineering of Escherichia coli for L-threonine production[D]. Wuxi: Jiangnan University, 2018 (in Chinese). |
| [42] |
周茜, 郑会明, 刘辉, 等. 增强乙醛酸循环对大肠杆菌合成L-苏氨酸的影响. 现代食品科技, 2015, 31(10): 109-114. Zhou Q, Zheng HM, Liu H, et al. Effect of enhancing glyoxylate cycle on L-threonine production in Escherichia coli. Mod Food Sci Technol, 2015, 31(10): 109-114 (in Chinese). DOI:10.13982/j.mfst.1673-9078.2015.10.019 |
| [43] |
Song CW, Lee J, Ko YS, et al. Metabolic engineering of Escherichia coli for the production of 3-aminopropionic acid. Metab Eng, 2015, 30: 121-129. DOI:10.1016/j.ymben.2015.05.005
|
| [44] |
Xu JZ, Zhang JL, Guo YF, et al. Genetically modifying aspartate aminotransferase and aspartate ammonia-lyase affects metabolite accumulation in L-lysine producing strain derived from Corynebacterium glutamicum ATCC13032. J Mol Catal B Enzym, 2015, 113: 82-89. DOI:10.1016/j.molcatb.2014.12.015
|
| [45] |
Chae TU, Kim WJ, Choi S, et al. Metabolic engineering of Escherichia coli for the production of 1, 3-diaminopropane, a three carbon diamine. Sci Rep, 2015, 5: 13040. DOI:10.1038/srep13040
|
| [46] |
Park SJ, Kim EY, Noh W, et al. Metabolic engineering of Escherichia coli for the production of 5-aminovalerate and glutarate as C5 platform chemicals. Metab Eng, 2013, 16: 42-47. DOI:10.1016/j.ymben.2012.11.011
|
| [47] |
Chao YP, Lo TE, Luo NS. Selective production of L-aspartic acid and L-phenylalanine by coupling reactions of aspartase and aminotransferase in Escherichia coli. Enzyme Microb Technol, 2000, 27(1/2): 19-25.
|
| [48] |
Piao XY, Wang L, Lin BX, et al. Metabolic engineering of Escherichia coli for production of L-aspartate and its derivative β-alanine with high stoichiometric yield. Metab Eng, 2019, 54: 244-254. DOI:10.1016/j.ymben.2019.04.012
|
| [49] |
Roe AJ, O'Byrne C, Mclaggan D, et al. Inhibition of Escherichia coli growth by acetic acid: a problem with methionine biosynthesis and homocysteine toxicity. Microbiology (Reading), 2002, 148(Pt 7): 2215-2222.
|
| [50] |
Sang YL, Jin HP. Integration of systems biology with bioprocess engineering: L-threonine production by systems metabolic engineering of Escherichia coli. Adv Biochem Eng /Biotechnol, 2010, 120: 1-19.
|
| [51] |
Ikeda M, Ohnishi J, Mitsuhashi S. Genome breeding of an amino acid-producing Corynebacterium glutamicum mutant. Microbial Processes and Products. Totowa, NJ: Humana Press, 2005: 179-190.
|
| [52] |
张宇, 夏利, 林蓓蓓, 等. 高效合成L-高丝氨酸大肠杆菌基因工程菌株的构建. 食品科学, 2022, 43(6): 81-88. Zhang Y, Xia L, Lin BB, et al. Construction of Escherichia coli chassis for efficient production of L-homoserine. Food Sci, 2022, 43(6): 81-88 (in Chinese). |
| [53] |
王晨, 赵雨佳, 李春, 等. 动态转录调控微生物代谢途径研究进展. 化工进展, 2019, 38(9): 4238-4246. Wang C, Zhao YJ, Li C, et al. Advances in dynamic transcriptional regulation of microbial metabolic pathways. Chem Ind Eng Prog, 2019, 38(9): 4238-4246 (in Chinese). DOI:10.16085/j.issn.1000-6613.2019-0197 |
| [54] |
Salinas F, Rojas V, Delgado V, et al. Fungal light-oxygen-voltage domains for optogenetic control of gene expression and flocculation in yeast. mBio, 2018, 9(4): e00626-e00618.
|
| [55] |
方宇. 温控开关调控大肠杆菌中心碳流生产L-苏氨酸[D]. 无锡: 江南大学, 2021. Fang Y. Central carbon flux regulation of Escherichia coli for L-threonine production using a thermal switch[D]. Wuxi: Jiangnan University, 2021 (in Chinese). |
| [56] |
Long MF, Xu MJ, Ma ZF, et al. Significantly enhancing production of trans-4-hydroxy-L-proline by integrated system engineering in Escherichia coli. Sci Adv, 2020, 6(21): eaba2383. DOI:10.1126/sciadv.aba2383
|
| [57] |
Yu BJ, Sung BH, Lee JY, et al. sucAB and sucCD are mutually essential genes in Escherichia coli. FEMS Microbiol Lett, 2006, 254(2): 245-250. DOI:10.1111/j.1574-6968.2005.00026.x
|
| [58] |
Zhao M, Huang DX, Zhang XJ, et al. Metabolic engineering of Escherichia coli for producing adipic acid through the reverse adipate-degradation pathway. Metab Eng, 2018, 47: 254-262. DOI:10.1016/j.ymben.2018.04.002
|
| [59] |
Nakamori S, Kobayashi S, Nishimura T, et al. Mechanism of L-methionine overproduction by Escherichia coli: the replacement of Ser-54 by Asn in the MetJ protein causes the derepression of L-methionine biosynthetic enzymes. Appl Microbiol Biotechnol, 1999, 52(2): 179-185. DOI:10.1007/s002530051506
|
| [60] |
Chassagnole C, Raïs B, Quentin E, et al. An integrated study of threonine-pathway enzyme kinetics in Escherichia coli. Biochem J, 2001, 356(Pt 2): 415-423.
|
| [61] |
Grundy FJ, Lehman SC, Henkin TM. The L box regulon: lysine sensing by leader RNAs of bacterial lysine biosynthesis genes. PNAS, 2003, 100(21): 12057-12062. DOI:10.1073/pnas.2133705100
|
| [62] |
Kotaka M, Ren JS, Lockyer M, et al. Structures of R-and T-state Escherichia coli aspartokinase Ⅲ: mechanisms of the allosteric transition and inhibition by lysine. J Biol Chem, 2006, 281(42): 31544-31552.
|
| [63] |
Fondi M, Brilli M, Fani R. On the origin and evolution of biosynthetic pathways: integrating microarray data with structure and organization of the common pathway genes. BMC Bioinformatics, 2007, 8(Suppl 1): S12.
|
| [64] |
James CL, Viola RE. Production and characterization of bifunctional enzymes. substrate channeling in the aspartate pathway. Biochemistry, 2002, 41(11): 3726-3731.
|
| [65] |
Lee JH, Lee DE, Lee BU, et al. Global analyses of transcriptomes and proteomes of a parent strain and an L-threonine-overproducing mutant strain. J Bacteriol, 2003, 185(18): 5442-5451. DOI:10.1128/JB.185.18.5442-5451.2003
|
| [66] |
Kim CH, Kim SY, Shin YU, et al. Microorganism producing L-methionine precursor and the method of producing L-methionine precursor using the microorganism: US08465952B2. 2015-04-14
|
| [67] |
Kim S, Cho K, Shin Y, et al. Microorganism producing L-methionine precursor and method of producing L-methionine and organic acid from the L-methionine precursor: US20150211034. 2015-07-30.
|
| [68] |
Lee HS, Hwang BJ. Methionine biosynthesis and its regulation in Corynebacterium glutamicum: parallel pathways of transsulfuration and direct sulfhydrylation. Appl Microbiol Biotechnol, 2003, 62(5/6): 459-467.
|
| [69] |
Park SD, Lee JY, Sim SY, et al. Characteristics of methionine production by an engineered Corynebacterium glutamicum strain. Metab Eng, 2007, 9(4): 327-336. DOI:10.1016/j.ymben.2007.05.001
|
| [70] |
Shim J, Shin Y, Lee I, et al. L-methionine production. Adv Biochem Eng Biotechnol, 2017, 159: 153-177.
|
| [71] |
Chica RA, Doucet N, Pelletier JN. Semi-rational approaches to engineering enzyme activity: combining the benefits of directed evolution and rational design. Curr Opin Biotechnol, 2005, 16(4): 378-384. DOI:10.1016/j.copbio.2005.06.004
|
| [72] |
Liu Y, Yan Z, Lu X, et al. Improving the catalytic activity of isopentenyl phosphate kinase through protein coevolution analysis. Sci Reports, 2016, 6: 24117.
|
| [73] |
Steiner K, Schwab H. Recent advances in rational approaches for enzyme engineering. Comput Struct Biotechnol J, 2012, 2: e201209010. DOI:10.5936/csbj.201209010
|
| [74] |
Kase H, Nakayama K. Fermentation production of L-methionine and regulation of L-methionine biosynthesis on Corynebacterium glutamicum. I. Production of O-acetyl-L-homoserine by methionine analogresisttant mutants and regulation of homoserine-O-transacetylase in Corynebacterium glutamicum. Agric Biol Chem, 1974, 38(10): 2021-2030.
|
| [75] |
Willke T. Methionine production—a critical review. Appl Microbiol Biotechnol, 2014, 98(24): 9893-9914. DOI:10.1007/s00253-014-6156-y
|
| [76] |
刘吉颂. O-乙酰高丝氨酸发酵培养基优化及其发酵动力学研究[D]. 杭州: 浙江工业大学, 2020. Liu JS. Study on fermentation medium and fermentation kinetics of O-acetyl-homoserine[D]. Hangzhou: Zhejiang University of Technology, 2020 (in Chinese). |
| [77] |
Wang YP, San KY, Bennett GN. Cofactor engineering for advancing chemical biotechnology. Curr Opin Biotechnol, 2013, 24(6): 994-999. DOI:10.1016/j.copbio.2013.03.022
|
| [78] |
Heux S, Cachon R, Dequin S. Cofactor engineering in Saccharomyces cerevisiae: expression of a H2O-forming NADH oxidase and impact on redox metabolism. Metab Eng, 2006, 8(4): 303-314. DOI:10.1016/j.ymben.2005.12.003
|
| [79] |
Vadali RV, Bennett GN, San KY. Cofactor engineering of intracellular CoA/acetyl-CoA and its effect on metabolic flux redistribution in Escherichia coli. Metab Eng, 2004, 6(2): 133-139. DOI:10.1016/j.ymben.2004.02.001
|
| [80] |
Hao YN, Ma Q, Liu XQ, et al. High-yield production of L-valine in engineered Escherichia coli by a novel two-stage fermentation. Metab Eng, 2020, 62: 198-206. DOI:10.1016/j.ymben.2020.09.007
|
| [81] |
Lama SM, Kim Y, Nguyen DT, et al. Production of 3-hydroxypropionic acid from acetate using metabolically-engineered and glucose-grown Escherichia coli. Bioresour Technol, 2021, 320: 124362. DOI:10.1016/j.biortech.2020.124362
|
| [82] |
Li Z, Ding DQ, Wang HY, et al. Engineering Escherichia coli to improve tryptophan production via genetic manipulation of precursor and cofactor pathways. Synth Syst Biotechnol, 2020, 5(3): 200-205. DOI:10.1016/j.synbio.2020.06.009
|
| [83] |
Ye ZX, Li S, Hennigan JN, et al. Two-stage dynamic deregulation of metabolism improves process robustness & scalability in engineered E. coli. New York: Cold Spring Harbor Laboratory Press, 2020.
|
| [84] |
Lynch MD, Lipscomb T, Trahan AD, et al. Microbial production of chemical products and related compositions, methods and systems: EP2689020B1. 201209-27.
|
| [85] |
Jawed K, Mattam AJ, Fatma Z, et al. Engineered production of short chain fatty acid in Escherichia coli using fatty acid synthesis pathway. PLoS ONE, 2016, 11(7): e0160035. DOI:10.1371/journal.pone.0160035
|
| [86] |
Li S, Ye ZX, Lebeau J, et al. Dynamic control over feedback regulation identifies pyruvate-ferredoxin oxidoreductase as a central metabolic enzyme in stationary phase E. coli. New York: Cold Spring Harbor Laboratory Press, 2020
|
| [87] |
Li S, Ye ZX, Moreb EA, et al. Dynamic control over feedback regulatory mechanisms improves NADPH flux and xylitol biosynthesis in engineered E. coli. Metab Eng, 2021, 64: 26-40. DOI:10.1016/j.ymben.2021.01.005
|
| [88] |
Akhtar MK, Jones PR. Construction of a synthetic YdbK-dependent pyruvate: H2 pathway in Escherichia coli BL21 (DE3). Metab Eng, 2009, 11(3): 139-147. DOI:10.1016/j.ymben.2009.01.002
|
| [89] |
Fàbrega A, Rosner JL, Martin RG, et al. SoxS-dependent coregulation of ompN and ydbK in a multidrug-resistant Escherichia coli strain. FEMS Microbiol Lett, 2012, 332(1): 61-67. DOI:10.1111/j.1574-6968.2012.02577.x
|
| [90] |
Nakayama T, Yonekura SI, Yonei S, et al. Escherichia coli pyruvate: flavodoxin oxidoreductase, YdbK-regulation of expression and biological roles in protection against oxidative stress. Genes Genet Syst, 2013, 88(3): 175-188. DOI:10.1266/ggs.88.175
|
| [91] |
Karkhanis VA, Mascarenhas AP, Martinis SA. Amino acid toxicities of Escherichia coli that are prevented by leucyl-tRNA synthetase amino acid editing. J Bacteriol, 2007, 189(23): 8765-8768. DOI:10.1128/JB.01215-07
|
| [92] |
Templeton BA, Savageau MA. Transport of biosynthetic intermediates: homoserine and threonine uptake in Escherichia coli. J Bacteriol, 1974, 117(3): 1002-1009. DOI:10.1128/jb.117.3.1002-1009.1974
|
| [93] |
Sumantran VN, Schweizer HP, Datta P. A novel membrane-associated threonine permease encoded by the tdcC gene of Escherichia coli. J Bacteriol, 1990, 172(8): 4288-4294. DOI:10.1128/jb.172.8.4288-4294.1990
|
| [94] |
Livshits VA, Zakataeva NP, Aleshin VV, et al. Identification and characterization of the new gene rhtA involved in threonine and homoserine efflux in Escherichia coli. Res Microbiol, 2003, 154(2): 123-135. DOI:10.1016/S0923-2508(03)00036-6
|
| [95] |
Kruse D, Krämer R, Eggeling L, et al. Influence of threonine exporters on threonine production in Escherichia coli. Appl Microbiol Biotechnol, 2002, 59(2/3): 205-210.
|
| [96] |
Eggeling L, Sahm H. New ubiquitous translocators: amino acid export by Corynebacterium glutamicum and Escherichia coli. Arch Microbiol, 2003, 180(3): 155-160. DOI:10.1007/s00203-003-0581-0
|
| [97] |
Kang Z, Wang Y, Gu PF, et al. Engineering Escherichia coli for efficient production of 5-aminolevulinic acid from glucose. Metab Eng, 2011, 13(5): 492-498. DOI:10.1016/j.ymben.2011.05.003
|
| [98] |
Zakataeva NP, Aleshin VV, Tokmakova IL, et al. The novel transmembrane Escherichia coli proteins involved in the amino acid efflux. FEBS Lett, 1999, 452(3): 228-232. DOI:10.1016/S0014-5793(99)00625-0
|
| [99] |
Lange C, Mustafi N, Frunzke J, et al. Lrp of Corynebacterium glutamicum controls expression of the brnFE operon encoding the export system for L-methionine and branched-chain amino acids. J Biotechnol, 2012, 158(4): 231-241. DOI:10.1016/j.jbiotec.2011.06.003
|
| [100] |
Bellmann A, Vrljić M, Pátek M, et al. Expression control and specificity of the basic amino acid exporter LysE of Corynebacterium glutamicum. Microbiology (Reading), 2001, 147(Pt 7): 1765-1774.
|
| [101] |
Yin L, Shi F, Hu X, et al. Increasing L-isoleucine production in Corynebacterium glutamicum by overexpressing global regulator Lrp and two-component export system BrnFE. J Appl Microbiol, 2013, 114(5): 1369-1377. DOI:10.1111/jam.12141
|
 2022, Vol. 38
2022, Vol. 38