中国科学院微生物研究所、中国微生物学会主办
文章信息
- 蔡韬, 刘玉万, 朱蕾蕾, 苏浩, 王钰, 王国坤, 张玲玲, 朱之光, 盛翔, 毕昌昊, 马红武, 田朝光, 张学礼, 吴洽庆, 孙媛霞, 江会锋, 马延和
- CAI Tao, LIU Yuwan, ZHU Leilei, SU Hao, WANG Yu, WANG Guokun, ZHANG Lingling, ZHU Zhiguang, SHENG Xiang, BI Changhao, MA Hongwu, TIAN Chaoguang, ZHANG Xueli, WU Qiaqing, SUN Yuanxia, JIANG Huifeng, MA Yanhe
- 二氧化碳人工生物转化
- Artificial bioconversion of carbon dioxide
- 生物工程学报, 2022, 38(11): 4101-4114
- Chinese Journal of Biotechnology, 2022, 38(11): 4101-4114
- 10.13345/j.cjb.220889
-
文章历史
- Received: November 9, 2022
- Accepted: November 12, 2022
2. 国家合成生物技术创新中心,天津 300308
2. National Technology Innovation Center of Synthetic Biology, Tianjin 300308, China
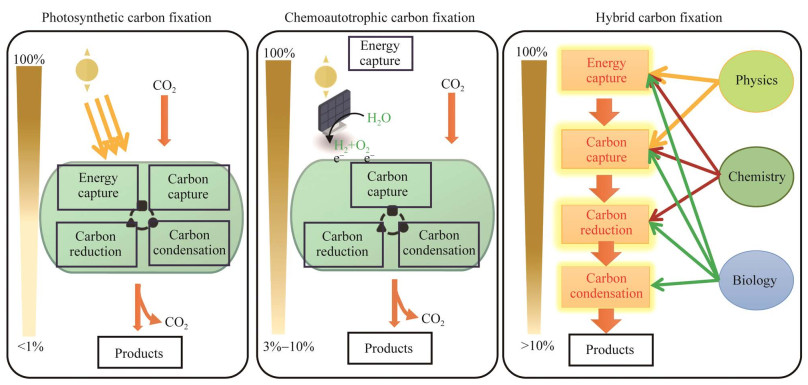
地球亿万年生命进化已形成了二氧化碳循环的复杂生物系统,主要包括吸收太阳能以CO2为原料合成碳水化合物的光合作用系统,以及利用糖酵解等路径将碳水化合物分解成CO2和能量的有机物分解系统,其科学本质就是以碳元素为载体的能量吸收与释放循环。虽然自然光合生物每年固定CO2超过2 000亿t,为地球生命体系演化繁衍提供了物质基础(图 1:光合固碳),但是光合生物CO2利用的全过程在一个细胞内进行了高度集成,整体上太阳能转换效率不足1%,同时CO2转化速率也只有工业微生物发酵的1%,难以满足高强度工业生产需求。
|
| 图 1 不同生物固碳模式 Fig. 1 Different modes of biological carbon fixation. |
| |
近年来世界各国科学家都在探索高效的CO2人工生物转化技术[1]。例如,中国科学院分子植物科学卓越创新中心姜卫红、顾阳研究组通过对自养细菌——食气永达尔梭菌(Clostridium ljungdahlii) 的组合代谢工程改造,实现了利用富含CO2/CO的合成气同步高效合成异丙醇、乙醇和3-羟基丁酸等重要产物,展示了食气梭菌在工业含碳气体生物转化制备高值长碳链化合物方面的良好应用潜力[2]。LanzaTech与首钢集团利用梭菌气体发酵技术,建立了世界上第一座合成气梭菌发酵产乙醇的工厂,近期又实现了中试规模利用CO2生产丙酮和异丙醇[3]。利用化学能可将CO2转化速率提高到工业发酵相当水平,但直接化能利用的能量来源不可再生,通过光电与化能高效转化耦合实现化能自养固碳并合成特定化学品是化能固碳的新选择。哈佛大学报道了无机催化剂-生物相容的体系,偶联光伏系统通过电解水产氢,再利用嗜氢自养固碳生物转化CO2合成生物质及化学品,光能利用效率达到3%‒10%[4] (图 1:化能固碳)。美国劳伦斯-伯克利国家实验室杨培东团队通过半导体纳米材料介导电能转换为化能驱动自养固碳菌株实现CO2到乙酸的转化[5]。光电与化能耦合转化固碳系统利用太阳能效率较自然光合作用有大幅提高,但能量利用、CO2还原、多碳生物转化都被整合在一个细胞里面,不仅受气液传质、电极-生物界面相容等工艺影响,还受生物电能利用机制、自养生物代谢规律不清所限,整体效率依然较低。
与生物固碳相比,化学固碳可实现更高的固碳速率和能量转化效率。中国科学院上海高等研究院在化学CO2转化方面取得了一系列进展,实现了CO2转化为甲醇、乙醇、甲酸、碳氢化合物等[6-10]。中国科学院大连化学物理研究所李灿院士团队,突破了太阳能电解水制取绿氢和二氧化碳加氢制甲醇的关键技术,实现了可再生能源利用和CO2减排[11]。厦门大学王野团队开发了金属表面修饰活化水分子促进CO2还原制甲酸的策略,在不同电流范围内可维持85%以上的甲酸选择性[12]。化学催化可以将CO2快速转化为短碳链化合物,但难以实现长碳链化合物的选择性合成。因此,充分发挥物理、化学、生物等技术在能量转换和固碳转化方面的优势,建立工程化杂合固碳技术体系是实现CO2人工转化利用的最佳选择(图 1:杂合固碳)。
中国科学院天津工业生物技术研究所(以下简称“天津工业生物所”) 在立所之初就聚焦绿色工业制造,重点布局高效人工固碳生物系统的创建。历经10年耕耘,研究所在人工固碳元件、途径与系统的设计与构建等前沿基础领域取得重要进展,特别是充分结合物理、化学、生物等技术优势,利用光伏发电制氢,然后化学还原CO2,再利用人工途径合成碳水化合物(图 1:杂合固碳),国际上首次不依赖光合作用实现CO2到淀粉分子的全合成,取得了标志性成果[13]。因此以CO2为原料进行人工生物转化利用,通过人工固碳元件设计与改造、人工固碳途径组装与适配、人工固碳系统的构建与调控,创建超越自然生物的人工固碳技术体系,创新增加碳汇的技术路径,可推动工业原料路线的战略转移,促进“碳达峰、碳中和”目标实现,服务于全球可持续发展,具有重大战略意义。
1 人工固碳元件设计与改造自然生物固碳元件是自然界亿万年进化的结果,早已经适应了空气中低浓度CO2、低密度太阳能的自然条件。如何解析自然界CO2还原、羧化、聚合等生物酶催化机制,开发蛋白酶从头设计改造技术方法,获得高效人工固碳新元件是CO2人工生物转化的关键。天津工业生物所盛翔团队的研究方向是酶催化机理研究,解析了多种脱羧(羧化) 酶的底物特异性及化学反应机理;天津工业生物所朱敦明、吴洽庆团队的研究方向是生物催化与绿色化工,利用酶催化的羧化反应实现了多种羧酸分子的合成;天津工业生物所朱之光团队的研究方向主要是生物燃料电池、生物电化学合成,构建的生物电催化体系实现了CO2的高效还原;天津工业生物所江会锋团队的研究方向是新酶设计,设计了能够催化甲醛缩合为羟基乙醛的羟基乙醛合成酶;天津工业生物所朱蕾蕾团队的研究方向是蛋白质定向进化,利用发展的高通量方法,提高了聚糖酶活性,改变了羟基乙醛合成酶的化学选择性。
1.1 羧化固碳元件机理解析与应用由于CO2具有较强的化学惰性,底物分子亲核进攻CO2分子需要克服较高的活化能,因此脱羧(羧化) 酶往往采用独特的反应机制,使得受体底物本身处于活性较高的非稳定态,并且通过酶与底物的相互作用稳定反应过渡态、降低活化能。从微观层面对不同脱羧(羧化) 酶催化机制的了解将有助于理解CO2的活化机理,并为相关酶的理性改造奠定重要的理论基础。天津工业生物所盛翔团队采用量子化学方法,在原子层面上系统研究了多种脱羧(羧化) 酶的底物特异性及化学反应机理,明确了碳碳键形成过程中稳定过渡态的关键氨基酸残基,阐明了不同体系中受体底物分子的高效活化机制。对5-羧基香草酸盐脱羧酶、2, 6-二羟基苯甲酸脱羧酶等二价金属依赖非氧化脱羧(羧化) 酶的研究发现,底物通过与金属离子双齿螯合配位提高亲核性,利于底物与CO2间的碳碳键形成[14-16]。对于辅酶依赖性的脱羧(羧化) 酶,底物往往通过与辅酶分子共价结合形成高亲核性的不稳定中间体,实现CO2的高效固定,例如在硫胺素二磷酸依赖性(thiamine diphosphate, ThDP) 脱羧(羧化) 酶中高活性的ThDP叶立德可高效活化底物分子形成碳负离子中间体[17],而在异戊二烯化黄素单核苷酸(prenylation flavin mononucleotide, prFMN) 依赖的脱羧(羧化) 酶中底物分子则通过与prFMN辅酶形成1, 3-环化中间体或高度共轭的类半醌中间体[18]。此外,一些非金属和非辅酶依赖性的脱羧(羧化) 酶,活性中心的氨基酸残基通过酸碱催化活化底物分子,如酚酸脱羧酶等[19]。通过羧化酶固定CO2生成高值化学品或中间体是CO2生物转化利用中最有效的途径之一。羧化酶在生物体代谢途径中广泛存在[20-21],其催化的化学反应是受体底物与CO2分子的碳碳键形成。由于这一转化是热力学不利过程,因此反应通常需要ATP提供能量,并且羧化活性普遍不高。近期,德国马克思普朗克陆地微生物研究所的Tobias Erb团队对天然羟乙酰辅酶A羧化酶进行改造,构建了具有较高活性的突变体酶,实现了二碳(乙醇酸) 转化为三碳化合物(甘油酸盐) 的高效固碳过程[22]。
近期研究发现多种类型的非氧化脱羧(羧化) 酶在压缩CO2气体或高浓度碳酸氢盐条件下具备较好的羧化活性,可以实现不依赖ATP等能量供体的CO2固定反应[18]。例如德国慕尼黑工业大学的Arne Skerra团队通过使用压缩CO2气体促进KdcA脱羧酶的羧化反应,将工业上常见的大宗中间体3-甲硫基丙醛直接羧化合成l-蛋氨酸[10, 17]。奥地利格拉兹大学的Faber Kurt团队则研究了多种脱羧酶催化的芳香族化合物羧化反应,提供了芳香羧酸类化合物合成的新思路[23]。天津工业生物所朱敦明、吴洽庆团队发现来源于真菌尖孢镰刀菌(Fusarium oxysporum) 的2, 3-二羟基苯甲酸脱羧酶具有较好的催化活性及温度耐受性,该酶在脱羧方向和羧化方向的催化效率都高于已经报道过的芳香羧酸脱羧酶[24],分别为203 min/(mmol·L) 和1.88 min/(mmol·L)。通过对该酶及其与邻苯二酚或2, 5-二羟基苯甲酸复合物的晶体结构解析及定点突变,从分子水平揭示了该酶的底物识别机制[25];同时,采用巧妙的方法在体系中添加季铵盐沉淀剂,解决了可逆平衡反应转化率低的问题,大大推动了反应平衡向羧化方向移动,使该酶对间苯二酚和邻苯二酚的羧化转化率达到97%,为二氧化碳绿色固定及有机芳香酸的绿色合成提供了新途径[26]。
1.2 二氧化碳还原原理与应用CO2还原需要电子,生物体通过光合作用、有机物分解等复杂过程提供CO2还原所需的电子,设计可以直接利用电极电子的人工酶元件有望实现更简单、高效的CO2还原。根据电子向酶元件反应中心传递方式的不同,可以分为直接电子转移和间接电子转移两种方式。前者是电子以共价结合的电子传递链接分子和蛋白为电子传递递质,直接被注入催化反应中心。依据Marcus的理论,直接电子转移的距离与速率成反比关系,要获得满足反应的高电子传递速率,电极距离反应中心在14 Å之内较为理想[27]。这一限定在一定程度上限制了直接电子转移方法的应用,并且酶在电极上的固定方式也会对酶的构象造成影响,从而影响酶活。后者则是电子由电极先转移到中介分子上,然后再由中介分子带入酶的反应中心。间接电子传递需要酶的催化中心或者电子传递中继点具备能够接纳电子介质分子的氧化还原反应中心以及稳定的溶液pH环境以维持反应电位的稳定[28]。与直接电子转移方式相比,间接电子转移方式可能更容易实现[29]。天津工业生物所朱之光团队设计了基于硫杆菌属(Thiobacillus sp.) KNK65MA来源的甲酸脱氢酶(formate dehydrogenase, FDH) 偶联铜纳米粒子的生物电催化体系用于CO2还原[30]。通过铜和酶表面的半胱氨酸残基之间形成的Cu-S键可以实现FDH在电极上的高效定向固定;通过聚乙二醇作为摆臂可以将辅因子NADH固定在FDH表面,便于辅因子NADH的自由穿梭。这种含有固定化FDH-辅因子复合物与铜纳米粒子构成的杂合体系,可以有效提升CO2还原效率,实现了甲酸产量为8.5 mmol/L,生产速率为11.8 μmol/(mU·h),为当时报道的最高水平。
中国科学院生物物理研究所王江云团队研究发现,通过使用基因密码子扩展技术可以将非天然氨基酸插入荧光蛋白,从而改造发色团提高光驱动还原活性;进一步在蛋白表面特定位置引入小分子镍配合物,可以驱动二氧化碳光还原[31]。基于Rosetta建模,该团队还设计了一种33 kDa光催化CO2还原酶,包含可以产生高还原活性物种生色团蛋白域和含2个铁硫簇的催化域。利用电化学检测手段,通过对还原电位的微调,优化了从生色团蛋白到铁硫簇的多步电子跃迁,最终得到了1.43%的CO2到甲酸的转换量子效率[32]。
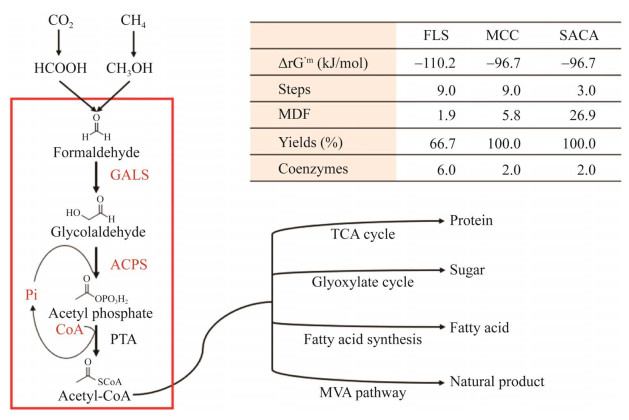
1.3 新型碳聚合酶元件设计与应用CO2被还原为简单的C1化合物后,下一步的挑战就是如何将其定向、高效地聚合成复杂的多碳化合物。实现一碳化合物的聚合反应具有很大挑战性,自然界不存在能够催化一碳化合物聚合反应的酶,在化学催化中只有卡宾类催化剂能够活化羰基碳,通过羰基碳的极性反转,实现C–C键延长。ThDP也是一种类卡宾催化剂。美国David Baker团队[33]基于ThDP催化原理设计了甲醛缩合酶(formolase, FLS),实现了3分子甲醛聚合生成1, 3-二羟基丙酮的聚糖反应,设计了线性一碳聚合途径将C1化合物同化至中心碳代谢网络。人工酶FLS的设计是现代蛋白质工程应用于全新生物合成途径构建的成功范例,已成功应用于CO2的固定[34]。天津工业生物所江会锋团队应用新酶设计策略[35],从脱羧酶出发,创建了两分子甲醛聚合生成一分子羟基乙醛的人工反应,实现了一碳化合物到二碳的线性转化。该研究组根据ThDP依赖酶的催化机制构建理论模型,基于理论模型对Protein Data Bank (PDB) 数据中的酶进行虚拟筛选,进一步根据ThDP催化反应原理,分析出ThDP分子中C2原子活性中心和聚合产物羟基乙醛的空间距离是触发催化反应的关键因素,通过分子对接根据空间距离筛选出6个备选蛋白。最终通过实验验证了能够催化甲醛聚合生成羟基乙醛的苯甲酰甲酸脱羧酶(benzoylformate decarboxylase, BFD)。经过4轮定向进化筛选提高了BFD催化甲醛缩合的催化效率,其最终突变体动力学参数kcat/Km为9.6 mol/s,甲醛缩合活性是FLS的2倍,被定义为羟基乙醛合成酶(glycolaldehyde synthase, GALS)。GALS的设计使人工C1转化途径的“出口”更加多样化,不仅限于C3化合物为同化节点。
天津工业生物所朱蕾蕾团队分别从FLS和GALS (BFD-M3) 出发,创建了新的甲醛聚合酶高通量筛选方法,通过定向进化的方式获得了高底物亲和力突变体FLS-M3[13]和耐受高浓度甲醛的突变体BFD-M6[36]、BFD-M4V2[37]。其中,突变体FLS-M3在低浓度甲醛(2 mmol/L) 条件下,活性提高至原来的3倍(催化效率kcat/Khalf为10 mol/s),2C/3C产物比例反转,成功应用于“CO2人工合成淀粉”途径[6]。FLS-M3的应用解除了该路径中一碳到三碳的催化活性瓶颈,很大程度避免了甲醛与二碳产物GA的毒性,有效推动了CO2人工合成淀粉途径的创建,同时使总酶使用量降低45%[13]。突变体BFD-M6具有大幅度提高C3产物活性(活性提高19倍)、甲醛耐受性(可耐受0.5 mol/L甲醛vs. FLS在0.2 mol/L甲醛浓度下丧失活性[38]) 和热稳定性(Tm值提高9.5 ℃)。进而,通过晶体解析和分子动力学模拟,研究了结构功能关系。在此基础上,成功设计了一条由甲醛生成乳酸的最短途径,利用BFD-M6和廉价NaOH建立了由甲醛合成乳酸的化学-酶法两步级联新反应,即将甲醛通过BFD-M6催化转化为DHA,再利用NaOH催化DHA进一步转化为乳酸,实现了乳酸的总收率为82.9%[36]。突变体BFD-M4V2催化产二碳产物GA的活性提高了11.7倍,可耐受甲醛浓度提高至1 mol/L,达到文献报道最高水平。在此基础上,设计了一锅两步的化学-酶法将甲醛转化为乙醇酸的新路径。该路径中,甲醛经BFD-M4V2转化为二碳产物GA和三碳产物DHA,进一步在绿色安全氧化剂NaClO2的作用下转化为乙醇酸[37]。甲醛聚合酶的设计与应用是人工一碳代谢途径设计以及生物催化领域的显著成果,随着未来合成生物学和蛋白质工程技术的发展,这一领域将有更加瞩目的成绩。
2 人工固碳途径组装与适配自然界已知固碳途径有6条,但是自然进化的固碳途径存在途径复杂、能耗高等问题,研究自然界的生物碳代谢规律,解析上下游生物催化反应机制,通过人工设计的固碳元件,创建新型高效的人工固碳途径,突破自然生物固碳路线局限,提高碳原子经济性,具有重要科学意义。天津工业生物所马红武团队的研究方向是计算生物学,利用开发的算法,挖掘了多条人工固碳途径;利用设计的羟基乙醛合成酶,天津工业生物所江会锋团队构建了合成乙酰辅酶A路径;天津工业生物所孙媛霞团队主要从事功能糖及天然活性物质的生物合成研究,利用人工聚糖路径,合成了多种稀有糖;天津工业生物所马延和团队利用光伏发电制氢,然后化学还原CO2,再利用聚糖路径,国际上首次不依赖光合作用实现CO2到淀粉分子的全合成。
2.1 人工固碳途径设计方法随着计算系统生物学的发展,借助算法和模型进行途径的批量计算和挖掘,使人工碳一生物转化途径进入系统理性设计阶段。以色列魏茨曼科学研究所的Ron Milo团队[39]将5 000个KEGG来源反应加入衣藻代谢网络模型,利用基于约束的通量平衡分析[40] (flux balance analysis, FBA) 计算预测了CO2固定新途径MOG (malonyl-CoA-oxaloacetate-glyoxylate),动力学分析显示该途径理论上比卡尔文循环速度快2–3倍,但并没有进行实验验证。天津工业生物所马红武团队基于大肠杆菌基因组尺度代谢网络模型iJO1366,利用FBA计算了一条新型固碳途径—苏氨酸循环[41],将该途径在大肠杆菌体内表达后,乙酰辅酶A衍生产品聚3-羟基丁酸酯得率提升3.3倍,但是途径步骤仍然复杂,能耗较高。
针对一碳利用途径存在步骤多、碳损失和能量消耗大等缺陷,马红武团队[42]通过代谢网络模型构建和comb-FBA算法开发,实现了无碳损、无ATP和还原力消耗、未知反应个数可控的甲醛吸收途径的系统性挖掘,以ATLAS数据库[43-44]中78个醛缩酶反应为组合子集,以MetaCyc数据集为主反应集,经过102 425次计算获得59条符合上述系列特征的目标途径。并通过实验验证了其中3条途径的可行性,其中羟基乙醛同化途径碳得率达到88%,超过已报道的甲醇聚合途径[45]。后续研究中,该团队根据醛缩酶反应机理,进一步将ATLAS中缺失的28个非天然反应添加到组合子集中,利用comb-FBA计算得到9条新途径,经过实验验证,GAPA途径可以催化乙醇醛高效转化为乙酸[46]。基于上述开发的comb-FBA算法,进一步对CO2产淀粉合成途径进行系统计算[13],以23个一碳利用反应,包括18个天然反应和5个已发表的非天然反应为组合子集,进行了667次组合计算,设计了10条可行途径,基于此预测结果,成功体外构建了仅需要11步反应催化的人工淀粉合成途径,极大地缩减了通过天然卡尔文循环合成淀粉所需的约60步反应。
2.2 人工固碳途径设计与构建中国科学院微生物研究所李寅团队设计了一个全新的最小化的POAP人工固碳循环[47]。这个循环只包含4步反应,分别由丙酮酸羧化酶、草酰乙酸乙酰基水解酶、乙酸-CoA连接酶和丙酮酸合酶催化。在4步反应中,由丙酮酸合酶和丙酮酸羧化酶催化的这两步反应均为固碳反应。POAP循环每运行一轮,可以转化2分子CO2生成1分子草酸,消耗2分子ATP和1分子还原力。由于途径短,在酮酸合酶活性远远低于CETCH循环固碳酶活性的情况下[48],POAP循环的CO2固定速率仍然超过了含有12步反应的CETCH循环。POAP循环可以在厌氧和较高温度(50 ℃) 下实现CO2固定,它为了解和研究地球早期生物如何进行CO2固定提供一个新的模型,也为CO2的人工生物转化提供了一条新的可选途径。
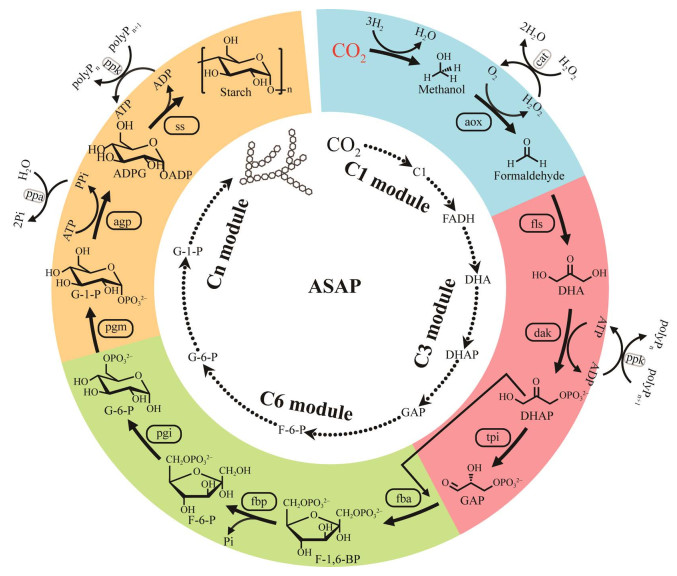
天津工业生物所江会锋团队在GALS催化的聚合反应基础上[35],设计了能够催化羟基乙醛直接生成乙酰磷酸的乙酰磷酸合成酶,结合天然代谢途径中的乙酰磷酸转移酶,实现了甲醛经3步催化反应生成乙酰辅酶A的人工乙酰辅酶A合成途径(synthetic acetyl-CoA, SACA) (图 2)。利用体外酶催化、体内同位素标记、细胞生长等实验,证明SACA途径无论在体外还是体内,都可以有效地将一碳转化成乙酰辅酶A,展示了合成生物学技术在突破自然进化限制方面的强大能力。人工设计的SACA途径突破了生物体固有代谢网络限制,具有化学驱动力大、不需要能量输入、与中心代谢正交和没有碳损失等优点,是第一条线性乙酰辅酶A人工生物合成途径,也是迄今为止最短的乙酰辅酶A生物合成途径。CO2可以通过化学或者生物转化为甲醛,因此,SACA途径的构建为CO2的转化利用奠定了基础。
天津工业生物所孙媛霞团队采用碳素叠加延伸碳链的策略,设计一碳转化路线。通过数据库挖掘和分子改造,获得了关键C–C键连接酶及突变体元件,通过耦合甲醛连接酶和醛缩酶设计了生物转化一碳单元甲醛迭代缩合合成l-赤藓酮糖的新路线[49],采用了分步级联反应策略,体外转化甲醛合成l-赤藓酮糖,转化率达到92%,在细胞中引入碳碳缩合酶,采用全细胞转化方法,甲醛耐受浓度可提高至3 mol/L,最终l-赤藓酮糖产量达到252 g/L,时空生产速率为126 g/(L·h),为l-赤藓酮糖生物制备提供了非常有效的合成策略。在此基础上,考虑到合成路线中DHA可磷酸化合成磷酸二羟丙酮(dihydroxyacetone phosphate, DHAP),结合前期筛选DHAP-依赖的醛缩酶,进一步设计建立了由醛缩酶和磷酸酶组成的支链酮糖合成路线[38],构建了基于聚磷酸激酶的辅酶再生体系,采用分步级联反应策略,实现转化甲醛合成支链酮糖,转化率达到86%,支链酮糖作为自然界非常稀有的一种化合物,可用于制备抗生素和液体燃料。综上所述,通过途径设计、组装和适配,实现从C1碳链延伸至C6化合物,为其他多碳糖的制备提供借鉴。
2.3 人工淀粉合成途径淀粉是粮食最主要的成分,同时也是重要的工业原料。目前淀粉主要由玉米等农作物通过自然光合作用固定CO2生产,淀粉合成与积累涉及60余步生化反应以及复杂的生理调控,理论能量转化效率仅为2%左右。农作物的种植通常需要较长周期,需要使用大量土地、淡水等资源以及肥料、农药等农业生产资料。天津工业生物所马延和团队从头设计了11步反应的非自然CO2固定与淀粉合成新途径(图 3),在实验室中首次实现了从CO2到淀粉分子的全合成[13]。研究团队采用了一种类似“搭积木”的方式,联合中国科学院大连化学物理研究所,利用化学催化剂将高浓度CO2在高密度氢能作用下还原成一碳(C1) 化合物,然后通过设计一碳聚合新酶,依据化学聚糖反应原理将一碳化合物聚合成三碳(C3) 化合物,最后通过生物途径优化,将三碳化合物又聚合成六碳(C6)化合物,再进一步合成支链和直链淀粉(Cn化合物)。这一人工途径的淀粉合成速率是玉米淀粉合成速率的8.5倍,系统适配跨越了自然途径数亿年的进化,向设计自然、超越自然目标的实现迈进了一大步,为创建新功能的生物系统提供了新的科学基础。
研究团队通过耦合化学催化与生物催化模块体系,创新了高密度能量与高浓度CO2利用的生物过程技术,通过反应时空分离优化,解决了人工途径中底物竞争、产物抑制、热/动力学匹配等问题,扩展了人工光合作用的能力。按照目前技术参数,理论上1 m3大小的生物反应器年产淀粉量相当于3 333.35 m2土地玉米种植的淀粉产量。这一成果使淀粉生产的传统农业种植模式向工业车间生产模式转变成为可能,并为CO2原料合成复杂分子开辟了新的技术路线。该成果为从CO2到淀粉生产的工业车间制造打开了一扇窗,如果未来该系统过程成本能够降低到与农业种植相比具有经济可行性,将会节约90%以上的耕地和淡水资源,避免农药、化肥等对环境的负面影响,提高人类粮食安全水平,促进碳中和的生物经济发展,推动形成可持续的生物基社会。
3 人工固碳底盘的构建与优化CO2需较高的能量和还原力才能实现转化利用。耦合电能或者生物质能固碳的杂合生物固碳,具有能量利用效率高、物质转化速率快等优势,成为生物固碳研究热点。生物质耦合固碳主要利用微生物发酵过程中生物质代谢产生的多余还原力及能量固定CO2合成化学品。电能直接固碳是利用可再生资源(包括光、风、潮汐、水力和地热) 产生的电能结合生物转化将CO2转化为有机化合物。还原性一碳生物转化,主要是利用化学电氢催化,还原CO2产生还原性C1能量载体,例如甲酸盐、一氧化碳、甲醇、甲烷等,以支持细胞生长或生产化学品。天津工业生物所田朝光团队、张学礼团队主要从事生物质的高效催化转化,结合CO2固定途径,实现了苹果酸、丁二酸的高效生物合成;天津工业生物所毕昌昊团队的研究方向主要是基因组和碱基编辑技术的开发,通过代谢工程改造,构建了高电活性的大肠杆菌电能固碳细胞;天津工业生物所孙际宾、郑平团队长期致力于谷氨酸棒杆菌(Corynebacterium glutamicum) 的人工甲醇生物转化研究,取得了一系列成果。
3.1 生物质耦合固碳在生物制造过程中,以生物质等原料物质主要为有机酸合成提供前体,同时产生大量的额外能量与还原力,受细胞代谢、目标合成途径的限制,这些能量、还原力往往通过代谢旁路或呼吸作用消耗掉,降低底物的利用效率。CO2的固定是一个高度消耗能量及还原力的过程,通过代谢途径人工重构,在微生物中人工导入CO2固定途径,将发酵原料产生的能量及还原力与CO2固定偶联,实现CO2的生物固定进入有机酸合成途径,增加目标产物的得率。天津工业生物所田朝光团队通过代谢工程手段在嗜热毁丝霉中搭建基于rTCA途径的l-苹果酸合成途径,创建了苹果酸发酵菌株,实现了生物质耦合固定CO2合成苹果酸[50]。在发酵罐中,直接以纤维素为原料,苹果酸产量达到181 g/L,以更为廉价的生物质玉米芯渣为碳源,苹果酸产量达到105 g/L。天津工业生物所张学礼团队将蓝藻的碳浓缩机制引入大肠杆菌细胞,提高了大肠杆菌胞内无机碳CO2的浓度,磷酸烯醇丙酮酸羧化激酶催化的羧化反应速率提高1.6倍,比以葡萄糖为原料合成丁二酸的速率提高了44%。
生物质水解产物主要包括六碳糖(葡萄糖)和木糖(木糖和阿拉伯糖),其五碳糖(木糖和阿拉伯糖) 主要通过磷酸戊糖途径进行代谢,代谢中间物5-磷酸核酮糖可以通过存在于光合生物卡尔文循环中磷酸核酮糖激酶和1, 5-二磷酸核酮糖羧化酶的催化,固定1分子CO2,同时形成2分子3-磷酸甘油酸。田朝光团队在嗜热毁丝霉苹果酸发酵菌株中导入了外源卡尔文循环途径的磷酸核酮糖激酶和1, 5-二磷酸核酮糖羧化酶,在原有的代谢途径上添加一条从5-磷酸核酮糖到3-磷酸甘油酸的代谢通路,有助于推动嗜热毁丝霉中原有的磷酸戊糖途径的代谢,同时提升CO2固定效率[51]。随后,结合木糖转运模块强化,进一步提升了菌株对木糖的利用效率,显著缩短了发酵周期。以粉碎的玉米芯为唯一碳源,苹果酸产量达到40 g/L,转化率为0.53 g/g。其中,CO2固定速率为33.8 mg/(L·h),每生产1 t苹果酸,消耗1.89 t玉米芯,并固定0.14 t CO2,为生物质耦合固碳提供了新策略。
3.2 电能直接固碳随着“碳达峰”和“碳中和”双碳目标的提出,微生物电合成利用电能作为还原力,可将CO2还原合成为各种目标化学品,越来越成为科学家们研究的热点[52-53]。天津工业生物所毕昌昊团队设计和搭建了以中性红作为电子载体介导的微生物电合成平台,并将来源于希瓦氏菌MR-1的电子传递途径相关的MtrABC膜蛋白复合体引入大肠杆菌,构建了高电活性的大肠杆菌电能细胞[54]。然后通过改造黄素腺嘌呤二核苷酸(flavin adenine dinucleotide, FAD) 的合成途径,提高了胞内FAD的水平,从而增加了大肠杆菌的电活性,还原性产物(乳酸、乙醇等) 和丁二酸转化率均得到了较大提升[55]。电子科技大学夏川团队与中国科学院深圳先进技术研究院于涛团队、中国科学技术大学曾杰团队合作,通过电催化将CO2和水合成高纯乙酸,再以乙酸及乙酸盐为碳源经生物发酵合成葡萄糖和脂肪酸等长碳链分子[56]。
3.3 一碳生物转化利用可再生能源化学还原CO2生成C1化合物(甲酸、甲醇等),可以为生物转化提供原料,是发展碳中和社会的一个重要组成方案。以甲醇利用速度快的天然甲基营养细菌和甲醇酵母为底盘,改造下游产物合成途径,构建天然甲基营养细胞工厂[57-59];以遗传背景清晰的工业平台微生物大肠杆菌和谷氨酸棒杆菌等为底盘,改造上游甲醇同化途径,构建人工甲基营养菌[60-63]。美国加州大学洛杉矶分校James Liao团队在人工甲基营养菌研究方面取得突破性进展,结合适应性进化等策略,该团队成功构建了可以以甲醇为唯一碳源的大肠杆菌[[64]。由于天然甲基营养菌缺乏相应的遗传操作工具,改造模式微生物大肠杆菌[64]、谷氨酸棒状杆菌[65]、解脂耶氏酵母[66]等同化甲醇,创建非天然甲基营养型底盘菌株,进行甲醇高效生物转化,成为了近年来的研究重点。
4 总结与展望自然生物系统每年利用光合作用固定CO2超2 000亿t,但空气中CO2依然逐年增加。全球碳素循环不平衡的本质是碳排放速度远高于自然生物的固碳转化速度。因此在阐明生命碳代谢原理基础上,需要进一步“减排加固”,一方面利用物理、化学与生物的杂合生物固碳系统提升人工生物固碳途径能效与速度,加快CO2固定与利用的速度;另一方面通过全新人工碳代谢途径设计,提供碳元素利用的原子经济性,减少必需产品合成的碳排放,最终建立以CO2为原料的新型生物制造工业体系,实现粮食、蛋白、材料等大宗产品的绿色生物制造路线,为“双碳”、粮食安全、新材料合成等提供全新的解决方案,为实现中国式现代化提供科技助力。
| [1] |
Liu Z, Wang K, Chen Y, et al. Third-generation biorefineries as the means to produce fuels and chemicals from CO2. Nat Catal, 2020, 3(3): 274-288. DOI:10.1038/s41929-019-0421-5
|
| [2] |
Jia DC, He MY, Tian Y, et al. Metabolic engineering of gas-fermenting Clostridium ljungdahlii for efficient co-production of isopropanol, 3-hydroxybutyrate, and ethanol. ACS Synth Biol, 2021, 10(10): 2628-2638. DOI:10.1021/acssynbio.1c00235
|
| [3] |
Liew FE, Nogle R, Abdalla T, et al. Carbon-negative production of acetone and isopropanol by gas fermentation at industrial pilot scale. Nat Biotech, 2022, 40(3): 335-344. DOI:10.1038/s41587-021-01195-w
|
| [4] |
Liu C, Colón BC, Ziesack M, et al. Water splitting-biosynthetic system with CO2 reduction efficiencies exceeding photosynthesis. Science, 2016, 352(6290): 1210-1213. DOI:10.1126/science.aaf5039
|
| [5] |
Su Y, Cestellos-Blanco S, Kim JM, et al. Close-packed nanowire-bacteria hybrids for efficient solar-driven CO2 fixation. Joule, 2020, 4(4): 800-811. DOI:10.1016/j.joule.2020.03.001
|
| [6] |
Zhang S, Liu X, Luo H, et al. Morphological modulation of Co2C by surface-adsorbed species for highly effective low-temperature CO2 reduction. ACS Catal, 2022, 12(14): 8544-8557. DOI:10.1021/acscatal.2c02020
|
| [7] |
Su X, Jiang Z, Zhou J, et al. Complementary operando spectroscopy identification of in-situ generated metastable charge-asymmetry Cu2-CuN3 clusters for CO2 reduction to ethanol. Nat Commun, 2022, 13(1): 1322. DOI:10.1038/s41467-022-29035-8
|
| [8] |
Bai X, Chen W, Zhao C, et al. Exclusive formation of formic acid from CO2 electroreduction by a tunable Pd-Sn alloy. Angew Chem Int Ed Engl, 2017, 56(40): 12219-12223. DOI:10.1002/anie.201707098
|
| [9] |
Song Y, Chen W, Zhao C, et al. Metal-free nitrogen-doped mesoporous carbon for electroreduction of CO2 to ethanol. Angew Chem Int Ed Engl, 2017, 56(36): 10840-10844. DOI:10.1002/anie.201706777
|
| [10] |
Gao P, Li S, Bu X, et al. Direct conversion of CO2 into liquid fuels with high selectivity over a bifunctional catalyst. Nat Chem, 2017, 9(10): 1019-1024. DOI:10.1038/nchem.2794
|
| [11] |
Wang J, Li G, Li Z, et al. A highly selective and stable ZnO-ZrO2 solid solution catalyst for CO2 hydrogenation to methanol. Sci Adv, 2017, 3(10): e1701290. DOI:10.1126/sciadv.1701290
|
| [12] |
Ma W, Xie S, Zhang XG, et al. Promoting electrocatalytic CO2 reduction to formate via sulfur-boosting water activation on indium surfaces. Nat Commun, 2019, 10(1): 892. DOI:10.1038/s41467-019-08805-x
|
| [13] |
Cai T, Sun H, Qiao J, et al. Cell-free chemoenzymatic starch synthesis from carbon dioxide. Science, 2021, 373(6562): 1523-1527. DOI:10.1126/science.abh4049
|
| [14] |
Sheng X, Himo F. Mechanisms of metal-dependent non-redox decarboxylases from quantum chemical calculations. Comput Struct Biotechnol J, 2021, 19: 3176-3186. DOI:10.1016/j.csbj.2021.05.044
|
| [15] |
Sheng X, Patskovsky Y, Vladimirova A, et al. Mechanism and structure of γ-resorcylate decarboxylase. Biochem, 2018, 57(22): 3167-3175. DOI:10.1021/acs.biochem.7b01213
|
| [16] |
Sheng X, Zhu W, Huddleston J, et al. A combined experimental-theoretical study of the LigW-catalyzed decarboxylation of 5-carboxyvanillate in the metabolic pathway for lignin degradation. ACS Catal, 2017, 7(8): 4968-4974. DOI:10.1021/acscatal.7b01166
|
| [17] |
Martin J, Eisoldt L, Skerra A. Fixation of gaseous CO2 by reversing a decarboxylase for the biocatalytic synthesis of the essential amino acid l-methionine. Nat Catal, 2018, 1(7): 555-561. DOI:10.1038/s41929-018-0107-4
|
| [18] |
Aleku GA, Roberts GW, Titchiner GR, et al. Synthetic enzyme-catalyzed CO2 fixation reactions. ChemSusChem, 2021, 14(8): 1781-1804. DOI:10.1002/cssc.202100159
|
| [19] |
Sheng X, Lind ME, Himo F. Theoretical study of the reaction mechanism of phenolic acid decarboxylase. FEBS J, 2015, 282(24): 4703-13. DOI:10.1111/febs.13525
|
| [20] |
Bernhardsgrütter I, Stoffel GMM, Miller TE, et al. CO2-converting enzymes for sustainable biotechnology: from mechanisms to application. Curr Opin in Biotech, 2021, 67: 80-87. DOI:10.1016/j.copbio.2021.01.003
|
| [21] |
Glueck SM, Gümüs S, Fabian WM, et al. Biocatalytic carboxylation. Chem Soc Rev, 2010, 39(1): 313-28. DOI:10.1039/B807875K
|
| [22] |
Scheffen M, Marchal DG, Beneyton T, et al. A new-to-nature carboxylation module to improve natural and synthetic CO2 fixation. Nat Catal, 2021, 4(2): 105-115. DOI:10.1038/s41929-020-00557-y
|
| [23] |
Payer SE, Faber K, Glueck SM. Non-oxidative enzymatic (de) carboxylation of (hetero) aromatics and acrylic acid derivatives. Adv Synth Catal, 2019, 361(11): 2402-2420. DOI:10.1002/adsc.201900275
|
| [24] |
Zhang X, Ren J, Yao P, et al. Biochemical characterization and substrate profiling of a reversible 2, 3-dihydroxybenzoic acid decarboxylase for biocatalytic Kolbe-Schmitt reaction. Enzyme Microb Technol, 2018, 113: 37-43. DOI:10.1016/j.enzmictec.2018.02.008
|
| [25] |
Song M, Zhang X, Liu W, et al. 2, 3-dihydroxybenzoic acid decarboxylase from Fusarium oxysporum: crystal structures and substrate recognition mechanism. Chembiochem, 2020, 21(20): 2950-2956. DOI:10.1002/cbic.202000244
|
| [26] |
Ren J, Yao P, Yu S, et al. An unprecedented effective enzymatic carboxylation of phenols. ACS Catal, 2016, 6(2): 564-567. DOI:10.1021/acscatal.5b02529
|
| [27] |
Ma S, Ludwig R. Direct electron transfer of enzymes facilitated by cytochromes. ChemElectroChem, 2019, 6(4): 958-975. DOI:10.1002/celc.201801256
|
| [28] |
Tasca F, Gorton L, Harreither W, et al. Comparison of direct and mediated electron transfer for cellobiose dehydrogenase from phanerochaete sordida. Anal Chem, 2009, 81(7): 2791-2798. DOI:10.1021/ac900225z
|
| [29] |
Algov I, Alfonta L. Use of protein engineering to elucidate electron transfer pathways between proteins and electrodes. ACS Meas Sci Au, 2022, 2(2): 78-90. DOI:10.1021/acsmeasuresciau.1c00038
|
| [30] |
Song H, Ma C, Liu P, et al. A hybrid CO2 electroreduction system mediated by enzyme-cofactor conjugates coupled with Cu nanoparticle-catalyzed cofactor regeneration. J CO2 Util, 2019, 34: 568-575. DOI:10.1016/j.jcou.2019.08.007
|
| [31] |
Liu X, Kang F, Hu C, et al. A genetically encoded photosensitizer protein facilitates the rational design of a miniature photocatalytic CO2-reducing enzyme. Nat Chem, 2018, 10(12): 1201-1206. DOI:10.1038/s41557-018-0150-4
|
| [32] |
Kang F, Yu L, Xia Y, et al. Rational design of a miniature photocatalytic CO2-reducing enzyme. ACS Catal, 2021, 11(9): 5628-5635. DOI:10.1021/acscatal.1c00287
|
| [33] |
Siegel JB, Smith AL, Poust S, et al. Computational protein design enables a novel one-carbon assimilation pathway. PNAS, 2015, 112(12): 3704-9. DOI:10.1073/pnas.1500545112
|
| [34] |
Hu G, Li Z, Ma D, et al. Light-driven CO2 sequestration in Escherichia coli to achieve theoretical yield of chemicals. Nature Catalysis, 2021, 4(5): 395-406. DOI:10.1038/s41929-021-00606-0
|
| [35] |
Lu X, Liu Y, Yang Y, et al. Constructing a synthetic pathway for acetyl-coenzyme A from one-carbon through enzyme design. Nat Commun, 2019, 10(1): 1378. DOI:10.1038/s41467-019-09095-z
|
| [36] |
Li T, Tang Z, Wei H, et al. Totally atom-economical synthesis of lactic acid from formaldehyde: combined bio-carboligation and chemo-rearrangement without the isolation of intermediates. Green Chem, 2020, 22(20): 6809-6814. DOI:10.1039/D0GC02433C
|
| [37] |
Li T, Tan Z, Tang Z, et al. One-pot chemoenzymatic synthesis of glycolic acid from formaldehyde. Green Chem, 2022, 24(13): 5064-5069. DOI:10.1039/D2GC00688J
|
| [38] |
Yang J, Zhu Y, Qu G, et al. Biosynthesis of dendroketose from different carbon sources using in vitro and in vivo metabolic engineering strategies. Biotechnol Biofuels, 2018, 11: 290. DOI:10.1186/s13068-018-1293-7
|
| [39] |
Bar-Even A, Noor E, Lewis NE, et al. Design and analysis of synthetic carbon fixation pathways. PNAS, 2010, 107(19): 8889-8894. DOI:10.1073/pnas.0907176107
|
| [40] |
Orth JD, Thiele I, Palsson BØ. What is flux balance analysis?. Nat Biotech, 2010, 28(3): 245-248. DOI:10.1038/nbt.1614
|
| [41] |
Lin Z, Zhang Y, Yuan Q, et al. Metabolic engineering of Escherichia coli for poly(3-hydroxybutyrate) production via threonine bypass. Microb Cell Fact, 2015, 14: 185. DOI:10.1186/s12934-015-0369-3
|
| [42] |
Yang X, Yuan Q, Luo H, et al. Systematic design and in vitro validation of novel one-carbon assimilation pathways. Metab Eng, 2019, 56: 142-153. DOI:10.1016/j.ymben.2019.09.001
|
| [43] |
Hadadi N, Hafner J, Shajkofci A, et al. ATLAS of biochemistry: a repository of all possible biochemical reactions for synthetic biology and metabolic engineering studies. ACS Synth Biol, 2016, 5(10): 1155-1166. DOI:10.1021/acssynbio.6b00054
|
| [44] |
Hafner J, MohammadiPeyhani H, Sveshnikova A, et al. Updated ATLAS of biochemistry with new metabolites and improved enzyme prediction power. ACS Synth Biol, 2020, 9(6): 1479-1482. DOI:10.1021/acssynbio.0c00052
|
| [45] |
Bogorad IW, Chen CT, Theisen MK, et al. Building carbon-carbon bonds using a biocatalytic methanol condensation cycle. PNAS, 2014, 111(45): 15928-15933. DOI:10.1073/pnas.1413470111
|
| [46] |
Mao Y, Yuan Q, Yang X, et al. Non-natural aldol reactions enable the design and construction of novel one-carbon assimilation pathways in vitro. Front Microbiol, 2021, 12: 677596. DOI:10.3389/fmicb.2021.677596
|
| [47] |
Xiao L, Liu G, Gong F, et al. A minimized synthetic carbon fixation cycle. ACS Catal, 2022, 12(1): 799-808. DOI:10.1021/acscatal.1c04151
|
| [48] |
Schwander T, Schada von Borzyskowski L, Burgener S, et al. A synthetic pathway for the fixation of carbon dioxide in vitro. Science, 2016, 354(6314): 900-904. DOI:10.1126/science.aah5237
|
| [49] |
Yang J, Sun S, Men Y, et al. Transformation of formaldehyde into functional sugars via multi-enzyme stepwise cascade catalysis. Catal Sci Technol, 2017, 7(16): 3459-3463. DOI:10.1039/C7CY01062A
|
| [50] |
Li J, Lin L, Sun T, et al. Direct production of commodity chemicals from lignocellulose using Myceliophthora thermophila. Metab Eng, 2020, 61: 416-426. DOI:10.1016/j.ymben.2019.05.007
|
| [51] |
Li J, Chen B, Gu S, et al. Coordination of consolidated bioprocessing technology and carbon dioxide fixation to produce malic acid directly from plant biomass in Myceliophthora thermophila. Biotechnol Biofuels, 2021, 14(1): 186. DOI:10.1186/s13068-021-02042-5
|
| [52] |
Qi X, Jia X, Wang Y, et al. Development of a rapid startup method of direct electron transfer-dominant methanogenic microbial electrosynthesis. Bioresour Technol, 2022, 358: 127385. DOI:10.1016/j.biortech.2022.127385
|
| [53] |
Sadhukhan J, Lloyd JR, Scott K, et al. A critical review of integration analysis of microbial electrosynthesis (MES) systems with waste biorefineries for the production of biofuel and chemical from reuse of CO2. Renew Sustain Energy Rev, 2016, 56: 116-132. DOI:10.1016/j.rser.2015.11.015
|
| [54] |
Wu Z, Wang J, Liu J, et al. Engineering an electroactive Escherichia coli for the microbial electrosynthesis of succinate from glucose and CO(2). Microb Cell Fact, 2019, 18(1): 15. DOI:10.1186/s12934-019-1067-3
|
| [55] |
Wu Z, Wang J, Zhang X, et al. Engineering an electroactive Escherichia coli for the microbial electrosynthesis of succinate by increasing the intracellular FAD pool. Biochem Eng J, 2019, 146: 132-142. DOI:10.1016/j.bej.2019.03.015
|
| [56] |
Zheng T, Zhang M, Wu L, et al. Upcycling CO2 into energy-rich long-chain compounds via electrochemical and metabolic engineering. Nat Catal, 2022, 5(5): 388-396. DOI:10.1038/s41929-022-00775-6
|
| [57] |
Yuan XJ, Chen WJ, Ma ZX, et al. Rewiring the native methanol assimilation metabolism by incorporating the heterologous ribulose monophosphate cycle into Methylorubrum extorquens. Metab Eng, 2021, 64: 95-110. DOI:10.1016/j.ymben.2021.01.009
|
| [58] |
Gao J, Gao N, Zhai X, et al. Recombination machinery engineering for precise genome editing in methylotrophic yeast Ogataea polymorpha. iScience, 2021, 24(3): 102168. DOI:10.1016/j.isci.2021.102168
|
| [59] |
Zhang M, Yuan XJ, Zhang C, et al. Bioconversion of methanol into value-added chemicals in native and synthetic methylotrophs. Curr Issues Mol Biol, 2019, 33: 225-236.
|
| [60] |
Müller JEN, Meyer F, Litsanov B, et al. Engineering Escherichia coli for methanol conversion. Metab Eng, 2015, 28: 190-201. DOI:10.1016/j.ymben.2014.12.008
|
| [61] |
Wang J, Jian X, Xing XH, et al. Empowering a methanol-dependent Escherichia coli via adaptive evolution using a high-throughput microbial microdroplet culture system. Front Bioeng Biotechnol, 2020, 8: 570. DOI:10.3389/fbioe.2020.00570
|
| [62] |
Tuyishime P, Wang Y, Fan L, et al. Engineering Corynebacterium glutamicum for methanol-dependent growth and glutamate production. Metab Eng, 2018, 49: 220-231. DOI:10.1016/j.ymben.2018.07.011
|
| [63] |
Dai Z, Gu H, Zhang S, et al. Metabolic construction strategies for direct methanol utilization in Saccharomyces cerevisiae. Bioresource Technology, 2017, 245: 1407-1412. DOI:10.1016/j.biortech.2017.05.100
|
| [64] |
Chen FY, Jung HW, Tsuei CY, et al. Converting Escherichia coli to a synthetic methylotroph growing solely on methanol. Cell, 2020, 182(4): 933-946.e14. DOI:10.1016/j.cell.2020.07.010
|
| [65] |
Fan L, Wang Y, Qian J, et al. Transcriptome analysis reveals the roles of nitrogen metabolism and sedoheptulose bisphosphatase pathway in methanol-dependent growth of Corynebacterium glutamicum. Microb Biotechnol, 2021, 14(4): 1797-1808. DOI:10.1111/1751-7915.13863
|
| [66] |
Wang G, Olofsson-Dolk M, Hansson FG, et al. Engineering yeast Yarrowia lipolytica for methanol assimilation. ACS Synth Biol, 2021, 10(12): 3537-3550. DOI:10.1021/acssynbio.1c00464
|
 2022, Vol. 38
2022, Vol. 38