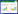中国科学院微生物研究所,中国微生物学会
文章信息
- 卢亚兰, 唐标, 杨华, 孙东昌. 2022
- LU Yalan, TANG Biao, YANG Hua, SUN Dongchang.
- CRISPR-Cas系统转录调控机制的研究进展
- Advances in transcriptional regulation mechanisms of CRISPR-Cas systems
- 微生物学报, 62(4): 1308-1321
- Acta Microbiologica Sinica, 62(4): 1308-1321
-
文章历史
- 收稿日期:2021-08-19
- 修回日期:2021-12-02
- 网络出版日期:2021-12-17
2. 浙江省农业科学院农产品质量标准研究所, 浙江 杭州 310021
2. Institute of Quality and Standard for Agro-products, Zhejiang Academy of Agricultural Sciences, Hangzhou 310021, Zhejiang, China
为了抵御噬菌体或质粒等的侵染,原核生物进化产生多种防御机制[1]。由成簇的规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)和CRISPR相关(CRISPR-associated,Cas)蛋白组成的CRISPR-Cas系统是原核生物进化产生的适应性免疫系统[2–5]。其中,CRISPR序列包括一个前导序列(leader)、一个或多个间隔序列(spacer)和多个重复序列(repeat)。CRISPR-Cas系统的作用机制可分为3个过程:适应(adaptation)、表达(expression)和干扰(interference)[6–10]。在适应阶段,外源核酸首次入侵细胞时,Cas整合酶将其序列中的部分核酸整合至CRISPR序列中,形成新的间隔序列[11–14];当宿主再次遇到类似外源核酸入侵时,cas基因表达生成Cas蛋白,同时CRISPR序列转录产生的前体crRNA (pre-crRNA)经Cas蛋白加工后形成成熟的crRNA[15–16];在干扰过程中,成熟的crRNA与Cas蛋白形成的核酸蛋白复合体(ribonucleoprotein complex,RNP)可识别并降解进入宿主菌的外源核酸[17–18]。基于其作用机制,可将CRISPR-Cas系统分为两大类(class 1和class 2)[19–22]。第一类CRISPR-Cas系统利用多亚基Cas蛋白和成熟的CRISPR RNA (crRNA)组成的效应复合物识别和切割靶标核酸,包括Ⅰ、Ⅲ和Ⅳ型;第二类CRISPR-Cas系统通过单个Cas蛋白和crRNA组成的效应复合物发挥作用,包括Ⅱ、Ⅴ和Ⅵ型[23]。
CRISPR-Cas系统可防御质粒编码的转座子或噬菌体的入侵。然而,CRISPR-Cas系统的持续性表达不仅增加了原核生物的代谢负担,而且可引发自我免疫(autoimmunity),导致原核生物自身的基因组DNA遭受来自CRISPR-Cas系统的损伤。因此,原核生物需要通过精准地调控CRISPR-Cas系统平衡外源核酸的摄取和防御。虽然人们对CRISPR-Cas系统的作用机制已开展了十分深入的研究,但是对其转录调控机制的研究相对较少[1]。探讨CRISPR/Cas系统的调控机制,不仅有助于正确认识原核生物利用适应性免疫防范水平基因转移风险的生理机制,和进一步完善原核生物转录调控理论;而且在开发更精准的基因编辑系统和利用细菌内源CRISPR-Cas系统防御耐药基因转移具有重要应用价值。近年来,人们在Ⅰ型、Ⅱ型以及Ⅲ型CRISPR-Cas系统中陆续开展了转录调控机制的研究(表 1)。其中,Ⅰ型CRISPR-Cas系统的转录调控机制相对较深入。因此,本文将重点阐述Ⅰ型CRISPR-Cas系统转录调控模式的最新研究,并简要介绍Ⅱ型和Ⅲ型系统转录调控机制的进展。
| Types | cis-acting elements | trans-acting factors | Regulatory mechanism | Species |
| Ⅰ-A | Pcsa1 | Csa3a[24–25] | Transcriptional activator | Sulfolobus islandicus |
| Csa3b[26–27] | Transcriptional repressor | Sulfolobus islandicus | ||
| Pcas | Csa3b[26] | Transcriptional repressor | Sulfolobus islandicus | |
| Ⅰ-E | Pcas3 | H-NS[28] | Transcriptional repressor | Escherichia coli |
| cAMP-CRP[29] | Transcriptional activator | Escherichia coli | ||
| Pcas | BaeSR[30] | Transcriptional activator | Escherichia coli | |
| H-NS[31] | Transcriptional repressor | Escherichia col | ||
| StpA[32] | Transcriptional activator in low level | Escherichia coli | ||
| LeuO[33] | Relieves repression by H-NS | Escherichia coli | ||
| LRP[34] | Transcriptional repressor | Salmonella enterica Serovar Typhi | ||
| cAMP-CRP[35] | Transcriptional repressor | Escherichia coli | ||
| SmaIR[36] | Transcriptional activator mediated by QS in high cell density | Serratia sp. | ||
| PCRISPR | H-NS[31] | Transcriptional repressor | Escherichia coli | |
| Ⅰ-F | Pcas1 | cAMP-CRP[37] | Transcriptional activator | Pectobacterium atrosepticum |
| Pcas1, Pcsy | SmaIR[36], LasIR, RhlIR[38] | Transcriptional activator mediated by QS in high cell density | Pseudomonas aeruginosa | |
| Pcsy | AlgU, AlgR, AmrZ[39] | Transcriptional repressor | Pseudomonas aeruginosa | |
| Pcas1 | PvdS[40] | Iron responsive transcriptional activator | Pseudomonas aeruginosa | |
| Ⅱ-A | Pcas9 | trL[41] | Guide Cas9 autoimmune suppression | Staphylococcus aureus |
| Ⅲ-A | cas operon | SmaIR[36] | Transcriptional activator mediated by QS in high cell density | Serratia sp. |
| Ⅲ-B | cmr operon | DdvS, CarD/G[42] | Transcriptional activator | Myxococcus xanthus |
| Csa3b[27] | Transcriptional activator | Sulfolobus islandicus |
1 Ⅰ型CRISPR-Cas系统的转录调控途径
Ⅰ型CRISPR-Cas系统广泛存在于细菌和古菌中,主要标志是一个N端含HD磷酸水解酶结构域和C端含DExH解旋酶结构域的Cas3蛋白[43–44]。在Cas3介导下,成熟的crRNA与CasA-D蛋白形成的cascade (CRISPR-associated complex for antiviral defence)复合物可靶标并切割DNA。Ⅰ型CRISPR-Cas系统包含6种亚型(Ⅰ-A、Ⅰ-B、Ⅰ-C、Ⅰ-D、Ⅰ-E和Ⅰ-F型)。目前,人们对Ⅰ-A、Ⅰ-E和Ⅰ-F 3种亚型的CRISPR-Cas系统调控机制了解较深入。后文将在简要说明表达上述3种亚型CRISPR-Cas系统操纵子结构的基础上,分别介绍它们的转录调控模式。
1.1 调控Ⅰ型CRISPR-Cas系统表达的顺式作用元件古菌冰岛硫化叶菌(Sulfolobus islandicus)是研究Ⅰ-A型CRISPR-Cas系统的模式菌株。在冰岛硫化叶菌REY15A中,Ⅰ-A型CRISPR-Cas系统的cas基因表达由2个启动子控制:参与适应过程的csa1、cas1、cas2和cas4 (又称acas操纵子)基因拥有一个共同的csa1启动子[24]。Pcas启动子控制由csa5、cas7、cas5、cas3、cas3HD、cas8a和cas6 (以下称为干扰基因簇)组成的操纵子的转录[25] (图 1)。干扰基因簇表达的蛋白与crRNA组成的cascade复合物可靶向切割外源核酸[26]。
|
| 图 1 CRISPR-Cas操纵子结构图 Figure 1 Structure diagram of CRISPR-Cas operons. Arrow boxes represent cas genes; black arrows represent promoters; black diamonds represent repeats; small rectangular boxes represent spacers. |
大肠杆菌(Escherichia coli)是研究Ⅰ-E型CRISPR-Cas系统的模式菌株。在大肠杆菌中,Ⅰ-E型CRISPR-Cas系统由2个CRISPR阵列和8个cas基因(cas1、cas2、cas3和casA–E)编码的Cas蛋白组成(图 1)[31]。Ⅰ-E型CRISPR-Cas系统的表达由3个启动子控制:Pcas3控制cas3基因的转录,cas1-2和casA–E组成的操纵子由位于cas1上游的Pcas启动子启动转录,位于前导序列的PCRISRR启动子控制CRISPR基因座的转录[31]。
大气果胶杆菌(Pectobacterium atrosepticum)和铜绿假单胞菌(Pseudomonas aeruginosa)均是研究Ⅰ-F型CRISPR-Cas系统调控的模式菌株。在Ⅰ-A和Ⅰ-E型CRISPR-Cas系统中,cas基因的转录由多个启动子控制。而在Ⅰ-F型CRISPR-Cas系统中,其cas基因的转录由一个或多个启动子控制[45]。在大气果胶杆菌的Ⅰ-F型CRISPR-Cas系统中,其所有的cas基因(cas1、cas3和csy1–4)组成的cas操纵子受单一启动子调控(图 1)[37];而在铜绿假单胞菌的Ⅰ-F型CRISPR-Cas系统中,cas1和cas2–3组成的操纵子和csy1–4组成的操纵子分别受启动子Pcas1和Pcsy控制[39]。
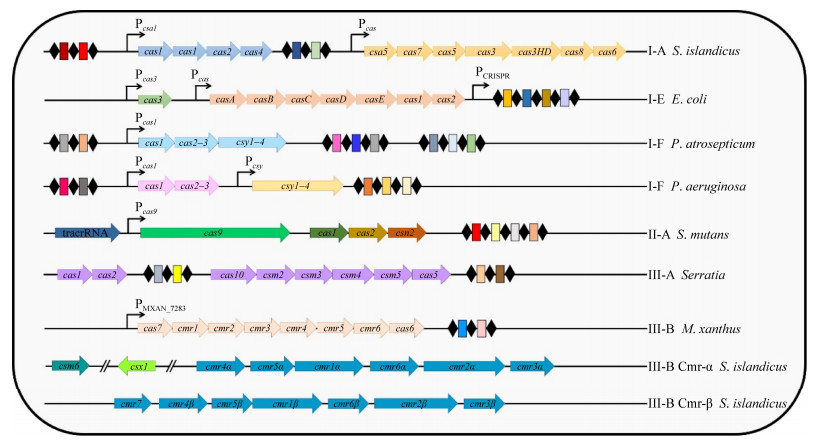
1.2 Ⅰ-A型CRISPR-Cas系统的调控途径在冰岛硫化叶菌REY15A中,Ⅰ-A型CRISPR-Cas系统受转录调控因子Csa3a和Csa3b调节(图 2)。当无病毒感染时,Csa3b不仅与Pcsa1启动子结合,抑制acas操纵子表达,从而降低Ⅰ-A型CRISPR-Cas系统获取新间隔区的能力[26–27];而且与Cascade复合物共结合在Pcas上,使干扰基因的转录完全被抑制[26]。当噬菌体侵袭宿主时,Csa3a与Pcsa1启动子和CRISPR前导序列结合,从而激活acas操纵子和CRISPR序列的转录,进而提高Ⅰ-A型CRISPR-Cas系统的适应和干扰能力[24–25];冰岛硫化叶菌REY15A中,Ⅲ-B型CRISPR-Cas的Cas10蛋白介导产生的信号分子环状寡聚腺苷酸(cyclic oligoadenylate,cOAs)可增强Csa3b与Pcsa1启动子的结合,这表明cOAs可能增强Csa3b对acas基因的抑制作用,进而可能影响Ⅰ-A型CRISPR-Cas系统的免疫活性[27]。
|
| 图 2 Ⅰ-A型CRISPR-Cas系统的转录调控机制 Figure 2 The mechanism of transcriptional regulation of the type Ⅰ-A CRISPR-Cas system. L: leader sequence; acas operon (adaptation genes) includes csa1, cas1, cas2 and cas4; gene cassettes: interference gene cassettes, consisting of csa5, cas7, cas5, cas3, cas3HD, cas8a and cas6. |
1.3 Ⅰ-E型CRISPR-Cas系统的调控途径
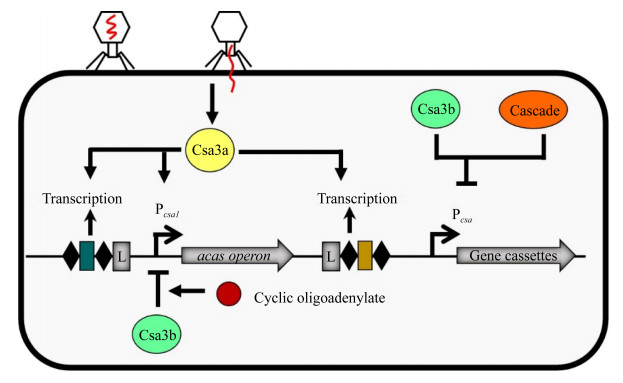
在大肠杆菌中,类组蛋白H-NS、StpA和转录调控因子LeuO等DNA结合蛋白调控Ⅰ-E型CRISPR-Cas系统的表达(图 3)。通常,Ⅰ-E型CRISPR-Cas系统在类组蛋白H-NS的转录抑制作用下处于沉默状态[31]。H-NS与Pcas3[28]、Pcas和PCRISPR结合抑制Ⅰ-E型CRISPR-Cas系统的活性[31]。H-NS的同源蛋白StpA与Pcas结合后激活cas基因的表达,增加Ⅰ-E型CRISPR-Cas系统对靶标DNA的切割能力[32]。LeuO与Pcas结合可解除H-NS的抑制作用,从而激活Ⅰ-E型CRISPR-Cas系统表达[33]。虽然亮氨酸反应性调节蛋白(leucine-responsive regulatory protein,LRP)与H-NS同属类组蛋白,但是在大肠杆菌中,LRP不参与Ⅰ-E型CRISPR-Cas系统的调控[31]。然而,在伤寒沙门氏菌(Salmonella enterica Serovar Typhi)中,LRP与H-NS均与cas启动子结合抑制casA基因的转录,进而影响Ⅰ-E型CRISPR-Cas系统的活性[34]。
某些调控因子(如双组分系统BaeSR、异源二聚体RcsB-BglJ、全局调控因子cAMP-CRP等)可通过H-NS或LeuO间接调节Ⅰ-E型CRISPR-Cas系统(图 3)。双组分调控系统BaeSR (two-component BaeSR regulatory system)由位于细胞膜上的组氨酸激酶和胞内的反应调控因子BaeR组成。膜压力作用下,活化的BaeS导致BaeR磷酸化,进而缓解H-NS对cas基因的抑制,激活Ⅰ-E型CRISPR-Cas系统的活性[30]。RcsB-BglJ可通过提高LeuO的转录水平,从而激活启动子Pcas[46]。cAMP-CRP (cAMP activated global transcriptional regulator)可双向调控Ⅰ-E型CRISPR-Cas系统的表达。cAMP-CRP可与LeuO竞争Pcas启动子的结合位点,从而抑制cas操纵子的转录,降低宿主菌对P1噬菌体感染的免疫能力[35]。此外,cAMP-CRP可与Pcas3启动子结合,激活cas3基因的表达,进而提高Ⅰ-E型CRISPR-Cas系统抵御质粒入侵的能力[29]。
|
| 图 3 Ⅰ-E型CRISPR-Cas系统的转录调控机制 Figure 3 The mechanism of the transcriptional regulation of the type Ⅰ-E CRISPR-Cas system. H-NS: heat-stable nucleoid-structuring protein; StpA: H-NS paralogue; LeuO: LysR-type transcription factors; LRP: leucine-responsive regulatory protein; cAMP: cyclic adenosine 3, 5-monophosphate; CRP: cAMP regulatory protein; RcsB-BglJ: heterodimers; BaeSR: envelope stress responsive two-component regulatory system; SmaIR: quorum sensing system; AI: autoinducer; P: phosphorylated; Imipenem: antibiotic. |
大肠杆菌中的Ⅰ-E型CRISPR-Cas系统不仅在转录水平上受严密的调控,而且在翻译后水平上也受调控。Cas3蛋白是影响CRISPR-Cas系统活性的关键蛋白。热稳定蛋白G (heat shock protein G,HptG)可通过增强Cas3蛋白的稳定性,进而提高Ⅰ-E型CRISPR-Cas系统对靶标DNA的干扰作用[47]。
在沙雷氏菌ATCC 39006中,群体感应(quorum sensing)参与Ⅰ-E型CRISPR-Cas系统的调控[36]。当细胞密度升高时,smaI基因的表达增加,导致信号分子N-酰基高丝氨酸内酯(AHL)的产量增多,AHL与SmaR结合后阻碍了SmaR对cas操纵子和CRISPR序列转录的抑制作用,从而激活了Ⅰ-E型CRISPR-Cas系统对靶标质粒的适应和干扰能力。
1.4 Ⅰ-F型CRISPR-Cas系统的调控途径在Ⅰ-E型和Ⅰ-A型中,人们对胞内因子(如Csa3a、H-NS和LeuO等)调控CRISPR-Cas系统表达的研究较多,而对外界信号如何影响CRISPR-Cas系统表达的认识相对不足。在调查Ⅰ-F型CRISPR-Cas系统调控机制过程中,人们发现外界信号(如葡萄糖水平、铁含量、温度等)可通过多种途径影响胞内因子,进而调节CRISPR-Cas系统表达(图 4)。在大气果胶杆菌中,环境中葡萄糖水平可调控Ⅰ-F型CRISPR-Cas系统的表达[37]。葡萄糖的添加可降低细胞内cAMP-CRP的水平,进而释放cAMP-CRP对Pcas1启动子的激活作用,从而降低Ⅰ-F型CRISPR-Cas活性对靶标质粒的适应和干扰能力。此外,GalM和GalK是参与乳糖代谢的酶,均可降低细胞内的CyaA (cAMP合成酶)水平,从而下调cas操纵子表达[48]。在铜绿假单胞菌UCBPP-PA14 (以下简称PA14)中,外界环境中的温度和铁含量可影响Ⅰ-F型CRISPR-Cas系统表达。在低温环境中,Ⅰ-F型CRISPR-Cas系统介导的将外源核酸片段整合至CRISPR阵列(适应)的能力增强[49]。在细胞缺铁时,细胞外γ功能因子PvdS的表达被上调,PvdS与核心RNA聚合酶(core RNA polymerase,cRNAP)结合形成复合物,通过与Pcas1上的结合位点结合增强cas基因的表达,并影响CRISPR-Cas系统的活性[40]。参与调控藻酸盐生物合成的双组分调控系统KinB-AlgB是Ⅰ-F型CRISPR-Cas系统的调控因子[39]。环境因素导致的膜蛋白KinB失活会促使AlgB磷酸化,从而激活周质蛋白酶AlgW降解MucA,进而导致与MucA结合的AlgU被释放,随后游离的AlgU激活AlgR和AmrZ。AlgU、AlgR和AmrZ均可抑制csy1–4操纵子的转录水平,进而影响Ⅰ-F型CRISPR-Cas系统防御噬菌体感染的能力[39]。此外,细胞密度也与Ⅰ-F型CRISPR-Cas系统的调控有关。铜绿假单胞菌PA14中,高细胞密度会增加噬菌体感染的风险;同时,群体感应系统的SmaIR、LasIR和RhlIR激活Ⅰ-F型系统cas基因的表达,增强其对噬菌体感染的免疫能力[36, 38]。
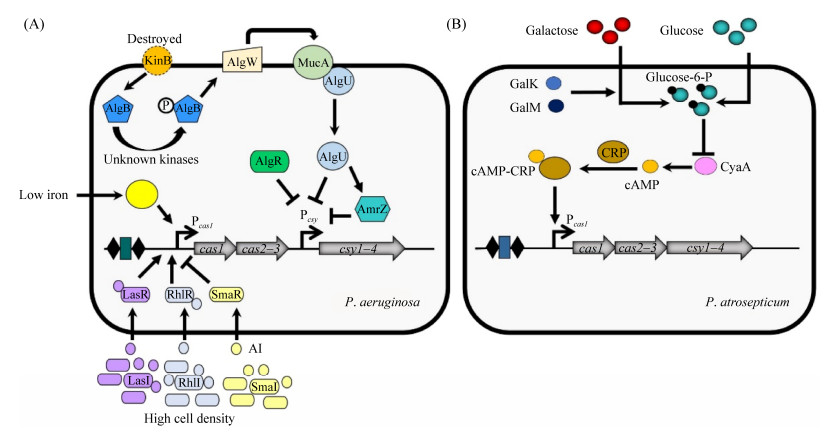
|
| 图 4 Ⅰ-F型CRISPR-Cas系统的转录调控机制 Figure 4 The mechanism of transcriptional regulation of the type Ⅰ-F CRISPR-Cas system. A: the regulation pathway of the Ⅰ-F CRISPR-Cas system of Pseudomonas aeruginosa; B: the regulation pathway of the Ⅰ-F CRISPR-Cas system of Pectobacterium atrosepticum; P: phosphate group. |
细菌内的小调节RNA (small regulatory RNAs,sRNAs)可快速响应外界环境信号,调控相应基因的表达[50]。在铜绿假单胞菌中,sRNAs参与Ⅰ-F型CRISPR-Cas系统的调控。PhrS作为sRNA的一种,可阻碍Rho蛋白与前导序列的结合,降低Rho蛋白对CRISPR序列转录的终止作用,促进CRISPR阵列的表达,进而增强Ⅰ-F型CRISPR-Cas系统防御噬菌体入侵的能力[50]。
2 Ⅱ型CRISPR-Cas系统的转录调控途径Ⅱ型CRISPR-Cas系统中,人们较为熟知的是Ⅱ-A型CRISPR-Cas9系统。该系统包括反式激活CRISPR-RNA (trans-activating crRNA,tracrRNA)基因,Cas蛋白的编码基因(cas9、cas1、cas2和csn2)和CRISPR阵列,其中Pcas9启动子控制由cas9、cas1、cas2和csn2组成的操纵子转录[51–52]。tracrRNA可与前体crRNA中的重复序列配对结合,随后被细胞内核糖核酸内切酶RNAse Ⅲ处理形成成熟crRNA。成熟crRNA与大分子蛋白Cas9组成核糖核蛋白复合体对目标序列进行识别和切割。CRISPR-Cas9系统介导的基因编辑技术已广泛应用于生命科学研究与应用。然而,直到最近,人们才初步认识其调控机制。
在变形链球菌(Streptococcus mutans) UA159中发现CRISPR-Cas9系统受生物被膜形成有关的组氨酸传感器激酶VicK和反应调节器VicR组成的双组分系统调控[53],暗示VicK可感应环境中某信号并将其传递给VicR,进而影响CRISPR-Cas9系统表达。除此之外,Ⅱ型CRISPR-Cas9系统还含有一条RNA介导的自我免疫途径[41]。在金黄色葡萄球菌(Staphylococcus aureus)的Ⅱ型CRISPR-Cas9系统中,tracrRNA基因有2个启动子,转录后形成长短2种构型的tracrRNA。其中,短构型的tracrRNA(trS)与前体crRNA互补配对,长构型的tracrRNA(trL)能够引导Cas9蛋白与Pcas9的结合,从而抑制cas操纵子的表达,进而影响CRISPR-Cas9系统的免疫能力[41]。然而,trL并不完全抑制CRISPR-Cas9系统,cas操纵子仍可维持低水平表达[41]。这种调控模式既能减轻细胞的代谢负担以及防止细胞产生自我免疫,又能维持一定的免疫防御外源核酸入侵的能力。
3 Ⅲ型CRISPR-Cas系统的转录调控途径Ⅲ型CRISPR-Cas系统虽多发现于古菌中,但也在少数细菌中存在,主要标志是Cas10蛋白。Ⅲ型CRISPR-Cas系统存在与Ⅰ型的cascade类似的干扰复合物[3]。Ⅲ-A/D型CRISPR-Cas系统中,成熟crRNA与Csm蛋白(Ⅲ-B/C型是Cmr蛋白)组成干扰复合物[54]。然而,与其他Ⅰ型和Ⅱ型CRISPR-Cas系统不同的是,Ⅲ型CRISPR-Cas系统同时靶标入侵DNA的单链DNA (ssDNA)和DNA的转录产物RNA[54]。Ⅲ型CRISPR-Cas系统的干扰复合物可与靶标RNA结合,并由具有RNA酶活性的Csm3 (或Cmr4)降解靶标RNA。ssDNA则由具有DNA酶活性的Cas10降解。目前,在沙雷氏菌(Serratia sp.) ATCC 39006、黄色粘球菌(Myxococcus xanthus) DK1050和冰岛硫化叶菌REY15A中揭示了Ⅲ-A和Ⅲ-B型CRISPR-Cas系统的调控机制。
在沙雷氏菌ATCC 39006中,Ⅲ-A型系统由cas1、cas2、cas10、csm3–5、cas6和2个CRISPR序列组成[36]。该系统受群体感应系统SmaIR调控。当细胞密度升高时,smaI基因的表达增加,导致信号分子N-酰基高丝氨酸内酯(AHL)的产量增多,AHL与SmaR结合后阻碍了SmaR对cas操纵子和CRISPR序列转录的抑制作用,从而激活了Ⅲ-A型CRISPR-Cas系统对靶标质粒的适应和干扰能力[36]。
在黄色粘球菌DK1050中,Ⅲ-B型CRISPR-Cas系统由cas6、cmr1-6和CRISPR阵列组成(图 1)[42]。CRISPR阵列与cmr操纵子共用一个启动子,其转录产物为多顺反子mRNA,经Cas6加工后形成成熟的crRNA。Ⅲ-B型CRISPR-Cas系统受σ因子(DdvS)和全局调控复合物(CarD-CarG)的调节。DdvS和CarD-CarG共同驱动cas基因的转录[42]。DdvS通常被膜结合的抗σ因子DdvA隔离,导致Ⅲ-B型CRISPR-Cas系统沉默。外部刺激(例如噬菌体或细胞外DNA入侵)导致DdvA的失活,使得DdvS从DdvA中释放出来;在CarD-CarG的协助下,DdvS与RNA聚合酶结合并激活cmr操纵子的转录,同时提高成熟crRNA的表达水平,从而激活CRISPR-Cas适应性免疫系统。目前,黄色粘球菌中CarD-CarG和DvdS激活Ⅲ-B型CRISPR-Cas系统表达的具体机制仍有待进一步研究。
在冰岛硫化菌REY15A中,除Ⅰ-A型CRISPR-Cas系统外,还存在2个Ⅲ-B型CRISPR-Cas系统(Ⅲ-B Cmr α和Ⅲ-B Cmr β)[27]。Ⅲ-B Cmr α的cmr基因包括csm4α、csm5α、csm1α、csm6α、csm2α和csm3α。Ⅲ-B Cmr β的cmr基因包括csm7、csm4β、csm5β、csm1β、csm6β、csm2β和csm3β。Csa3b是Ⅲ-B型CRISPR-Cas系统的激活因子[27]。Csa3b可与cmr基因的启动子(cmr4启动子和csm7启动子)结合,从而激活cmr基因的表达,进而增强Ⅲ-B型CRISPR-Cas对靶标基因的干扰能力。但需要注意的是,Csa3b仅能增强Ⅲ-B型CRISPR-Cas对靶标RNA的切割能力,而对Ⅲ-B型CRISPR-Cas介导的DNA干扰能力没有影响[27]。
4 应答环境因子平衡CRISPR-Cas适应性免疫与水平基因转移水平基因转移(horizontal gene transfer,HGT)是驱动原核生物进化的重要推动力[55]。在自然界中,水平基因转移包括3种方式:接合、自然转化和转导[55]。CRISPR-Cas系统可选择性降解通过水平基因转移进入细胞的外源核酸,保护宿主免受噬菌体等的侵害。然而CRISPR-Cas系统在细胞内持续性地表达会增加代谢负担并引起宿主的自身免疫,从而降低宿主在环境中的竞争力。因此,原核生物需要应答环境信号平衡水平基因转移和CRISPR-Cas介导的适应性免疫。目前,人们对于原核生物如何应答环境因子协调水平基因转移和CRISPR-Cas介导的适应性免疫的认知仍十分有限。
有线索表明环境因子参与平衡CRISPR-Cas介导的适应性免疫与自然转化介导的水平基因转移。在大肠杆菌中,环境中碳源(如葡萄糖)可调节细胞中cAMP的水平。cAMP与其受体蛋白CRP组成的复合物cAMP-CRP,不仅通过降低自然转化的激活因子RpoS的水平抑制自然转化[56],而且还通过与Ⅰ-E型CRISPR-Cas系统的激活剂LeuO竞争来抑制宿主对P1噬菌体入侵的免疫能力[35]。同时,cAMP-CRP通过激活cas3基因的转录水平提高大肠杆菌防御质粒入侵的能力[31]。因此,cAMP-CRP可同时影响自然转化和CRISPR-Cas系统,这提示环境中的碳源可通过cAMP-CRP调控自然转化介导和适应性免疫系统。类组蛋白H-NS和同源物StpA不仅调节大肠杆菌的免疫活性[32–33],而且在沙门氏菌中影响自然转化的调控因子RpoS的表达水平[57–58]。我们最新的研究表明H-NS可同时抑制大肠杆菌自然转化和CRISPR-Cas系统[59]。环境中抗生素可能也是参与平衡水平基因转移和CRISPR-Cas介导的适应性免疫的重要因子。在某些细菌中,抗生素可加速自然转化和接合介导的水平基因转移[60–61]。而在另外的细菌中,抗生素可通过H-NS抑制CRISPR-Cas系统[62]。例如,在肺炎克雷伯氏菌中,抗生素亚胺培南(imipenem)引起的细胞压力诱导转录沉默因子H-NS表达,降低cas3的表达水平,进而降低Ⅰ-E型CRISPR-Cas系统对靶标质粒的切割能力[62] (图 3)。群体感应也可能是调控CRISPR-Cas适应性免疫和自然遗传转化所共有的环境信号。细胞密度的增加可增加某些细菌被环境中噬菌体感染的风险[36]。在铜绿假单胞菌中,群体感应会激活Ⅰ-F型CRISPR-Cas系统,从而抵御噬菌体的入侵[38]。
5 总结与展望原核生物中CRISPR-Cas系统可降低外源核酸入侵的风险。然而,CRISPR-Cas系统持续表达不仅消耗细菌的资源,降低其环境适应能力;而且增加将细菌自身基因组的DNA片段整合至CRISPR序列,引发自我免疫的风险。因此,当原核生物处于噬菌体入侵的低风险时期时,其CRISPR-Cas系统处于沉默状态或只有较低的表达水平;而在原核生物处于易受外源核酸入侵的高风险情况下,原核生物或通过细胞膜上的膜蛋白,或通过影响细胞内某些基因的表达,将信号从胞外传递至胞内的调控因子,再由胞内的调节因子精细地调控CRISPR-Cas系统表达,降低外源核酸入侵的风险。
除转录调控外,人们还发现了调控CRISPR- Cas系统的其他机制。近年来,研究揭示了Ⅲ-A和Ⅲ-B型CRISPR-Cas系统的功能调控机制[63–65]。Ⅲ-A和Ⅲ-B型CRISPR-Cas系统的干扰复合物与靶标RNA结合,导致Cas10蛋白将ATP转化为环形寡聚腺苷酸(cyclic oligoadenylates,cOAs)。cOAs可激活Csm6 (或Csx1)的RNA酶活性,使得目标RNA被非特异性地降解[54]。在清除入侵者后,cOAs被宿主细胞内的环状核酸酶降解[54]。抗CRISPR蛋白是人们在噬菌体中发现的能抑制CRISPR-Cas系统活性的天然抑制剂,可帮助噬菌体逃逸CRISPR-Cas系统的攻击[66]。最近的研究表明,抗CRISPR蛋白(anti-CRISPR protein)—AcrⅢ-1蛋白可特异性降解cOA4,表明抗CRISPR蛋白可通过cOA4调控CRISPR-Cas系统[67]。另外,人们发现转录调控因子Csa3a可同时上调冰岛硫化叶菌的Ⅰ-A型CRISPR-Cas系统适应基因和DNA损伤修复相关基因的表达,从而修复由Ⅰ-A型CRISPR-Cas系统造成的宿主基因组的损伤,加深了对CRISPR-Cas介导的自我免疫与基因组稳定性的认知[68]。
综上所述,原核生物中CRISPR-Cas系统的转录调控方式多样,且细菌多通过全局或多效应调控因子(如H-NS、cAMP-CRP和BaeSR等)控制CRISPR-Cas系统的表达。在大肠杆菌中,全局调控因子H-NS和cAMP-CRP均可调控Ⅰ-E型CRISPR-Cas系统和自然转化介导的水平基因转移[35, 56, 59]。这暗示了当受到外界刺激时,细菌可能通过多效应因子偶联自然转化介导的水平基因转移与CRISPR-Cas介导的适应性免疫,从而使得宿主在摄取外源核酸的同时,启动胞内的CRISPR-Cas系统,降低危险核酸入侵的风险。此外,噬菌体感染会影响细菌的氨基酸代谢[2]。转录调节因子LeuO既能响应氨基酸饥饿,也可通过阻遏H-NS促进Ⅰ-E型CRISPR-Cas的表达[2],从而使得细菌既能防御噬菌体的入侵,又能维持氨基酸代谢的正常进行。然而,目前我们对原核生物利用全局调控因子调节CRISPR-Cas系统的机理认知十分有限,仍有待进一步的探究。
认识细菌利用CRISPR-Cas系统防御核酸入侵机制,将有助于我们了解自然环境中微生物的进化机制,并为控制耐药性和毒力基因在细菌中的传播提供新的策略。大肠杆菌中cAMP-CRP和H-NS同时抑制Ⅰ-E型CRISPR-Cas系统和自然转化,表明某些环境因子(如葡萄糖)可通过细胞内信号途径同时调控CRISPR-Cas系统和自然转化,但详细机理仍有待进一步探索。其他一些环境因子(如细胞密度、抗生素等)可在不同的细菌中分别调控CRISPR-Cas系统和水平基因转移,但仍不清楚在同一体系中这些环境因子是否及如何通过胞内途径同时调控水平基因转移和CRISPR-Cas系统。笔者认为鉴定调控水平基因转移和CRISPR-Cas系统共有的环境信号及相应的细胞内调控网络,将为深入认识在原核生物进化过程中CRISPR-Cas系统的生物学意义具有重要价值。
| [1] | Patterson AG, Yevstigneyeva MS, Fineran PC. Regulation of CRISPR-Cas adaptive immune systems. Current Opinion in Microbiology, 2017, 37: 1-7. DOI:10.1016/j.mib.2017.02.004 |
| [2] | Dorman CJ, Ni Bhriain N. CRISPR-Cas, DNA supercoiling, and nucleoid-associated proteins. Trends in Microbiology, 2020, 28(1): 19-27. DOI:10.1016/j.tim.2019.08.004 |
| [3] | Hille F, Richter H, Wong SP, Bratovič M, Ressel S, Charpentier E. The biology of CRISPR-Cas: backward and forward. Cell, 2018, 172(6): 1239-1259. DOI:10.1016/j.cell.2017.11.032 |
| [4] | Koonin EV, Makarova KS, Zhang F. Diversity, classification and evolution of CRISPR-Cas systems. Current Opinion in Microbiology, 2017, 37: 67-78. DOI:10.1016/j.mib.2017.05.008 |
| [5] | Mojica FJM, Díez-Villaseñor C, Soria E, Juez G. Biological significance of a family of regularly spaced repeats in the genomes of archaea, bacteria and mitochondria. Molecular Microbiology, 2000, 36(1): 244-246. DOI:10.1046/j.1365-2958.2000.01838.x |
| [6] | Behler J, Hess WR. Approaches to study CRISPR RNA biogenesis and the key players involved. Methods, 2020, 172: 12-26. DOI:10.1016/j.ymeth.2019.07.015 |
| [7] | Faure G, Shmakov SA, Yan WX, Cheng DR, Scott DA, Peters JE, Makarova KS, Koonin EV. CRISPR-Cas in mobile genetic elements: counter-defence and beyond. Nature Reviews Microbiology, 2019, 17(8): 513-525. DOI:10.1038/s41579-019-0204-7 |
| [8] | Mohanraju P, Makarova KS, Zetsche B, Zhang F, Koonin EV, Van Der Oost J. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science, 2016, 353(6299): aad5147. DOI:10.1126/science.aad5147 |
| [9] | Shivram H, Cress BF, Knott GJ, Doudna JA. Controlling and enhancing CRISPR systems. Nature Chemical Biology, 2021, 17(1): 10-19. DOI:10.1038/s41589-020-00700-7 |
| [10] | Xiao YB, Luo M, Dolan AE, Liao MF, Ke AL. Structure basis for RNA-guided DNA degradation by Cascade and Cas3. Science, 2018, 361(6397): eaat0839. DOI:10.1126/science.aat0839 |
| [11] | Amitai G, Sorek R. CRISPR-Cas adaptation: insights into the mechanism of action. Nature Reviews Microbiology, 2016, 14(2): 67-76. DOI:10.1038/nrmicro.2015.14 |
| [12] | Jackson SA, Birkholz N, Malone LM, Fineran PC. Imprecise spacer acquisition generates CRISPR-Cas immune diversity through primed adaptation. Cell Host & Microbe, 2019, 25(2): 250-260. |
| [13] | Radovčić M, Killelea T, Savitskaya E, Wettstein L, Bolt EL, Ivančić-Baće I. CRISPR-Cas adaptation in Escherichia coli requires RecBCD helicase but not nuclease activity, is independent of homologous recombination, and is antagonized by 5′ ssDNA exonucleases. Nucleic Acids Research, 2018, 46(19): 10173-10183. |
| [14] | Shiriaeva A, Fedorov I, Vyhovskyi D, Severinov K. Detection of CRISPR adaptation. Biochemical Society Transactions, 2020, 48(1): 257-269. DOI:10.1042/BST20190662 |
| [15] | Brouns SJJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJH, Snijders APL, Dickman MJ, Makarova KS, Koonin EV, Van Der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science, 2008, 321(5891): 960-964. DOI:10.1126/science.1159689 |
| [16] | Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao YJ, Pirzada ZA, Eckert MR, Vogel J, Charpentier E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886 |
| [17] | Nishimasu H, Nureki O. Structures and mechanisms of CRISPR RNA-guided effector nucleases. Current Opinion in Structural Biology, 2017, 43: 68-78. DOI:10.1016/j.sbi.2016.11.013 |
| [18] | Plagens A, Richter H, Charpentier E, Randau L. DNA and RNA interference mechanisms by CRISPR-Cas surveillance complexes. FEMS Microbiology Reviews, 2015, 39(3): 442-463. DOI:10.1093/femsre/fuv019 |
| [19] | Koonin EV, Makarova KS. Mobile genetic elements and evolution of CRISPR-Cas systems: all the way there and back. Genome Biology and Evolution, 2017, 9(10): 2812-2825. DOI:10.1093/gbe/evx192 |
| [20] | Koonin EV, Makarova KS. Origins and evolution of CRISPR-Cas systems. Philosophical Transactions of the Royal Society B: Biological Sciences, 2019, 374(1772): 20180087. DOI:10.1098/rstb.2018.0087 |
| [21] | Makarova KS, Wolf YI, Iranzo J, Shmakov SA, Alkhnbashi OS, Brouns SJJ, Charpentier E, Cheng D, Haft DH, Horvath P, Moineau S, Mojica FJM, Scott D, Shah SA, Siksnys V, Terns MP, Venclovas Č, White MF, Yakunin AF, Yan W, Zhang F, Garrett RA, Backofen R, Van Der Oost J, Barrangou R, Koonin EV. Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nature Reviews Microbiology, 2020, 18(2): 67-83. DOI:10.1038/s41579-019-0299-x |
| [22] | Makarova KS, Wolf YI, Koonin EV. Classification and nomenclature of CRISPR-Cas systems: where from here?. The CRISPR Journal, 2018, 1(5): 325-336. DOI:10.1089/crispr.2018.0033 |
| [23] | Murugan K, Babu K, Sundaresan R, Rajan R, Sashital DG. The revolution continues: newly discovered systems expand the CRISPR-Cas as toolkit. Molecular Cell, 2017, 68(1): 15-25. DOI:10.1016/j.molcel.2017.09.007 |
| [24] | Liu T, Li YJ, Wang XD, Ye Q, Li H, Liang YX, She QX, Peng N. Transcriptional regulator-mediated activation of adaptation genes triggers CRISPR de novo spacer acquisition. Nucleic Acids Research, 2015, 43(2): 1044-1055. DOI:10.1093/nar/gku1383 |
| [25] | Liu T, Liu ZZ, Ye Q, Pan SF, Wang XD, Li YJ, Peng WF, Liang YX, She QX, Peng N. Coupling transcriptional activation of CRISPR-Cas system and DNA repair genes by Csa3a in Sulfolobus islandicus. Nucleic Acids Research, 2017, 45(15): 8978-8992. DOI:10.1093/nar/gkx612 |
| [26] | He F, Vestergaard G, Peng WF, She QX, Peng X. CRISPR-Cas type Ⅰ—a Cascade complex couples viral infection surveillance to host transcriptional regulation in the dependence of Csa3b. Nucleic Acids Research, 2017, 45(4): 1902-1913. |
| [27] | Ye Q, Zhao XQ, Liu JL, Zeng ZF, Zhang ZF, Liu T, Li YJ, Han WY, Peng N. CRISPR-associated factor Csa3b regulates CRISPR adaptation and Cmr-mediated RNA interference in Sulfolobus islandicus. Frontiers in Microbiology, 2020, 11: 2038. DOI:10.3389/fmicb.2020.02038 |
| [28] | Majsec K, Bolt EL, Ivančić-Baće I. Cas3 is a limiting factor for CRISPR-Cas immunity in Escherichia coli cells lacking H-NS. BMC Microbiology, 2016, 16: 28. DOI:10.1186/s12866-016-0643-5 |
| [29] | Yang DH, Wang ZF, Ma JJ, Fu Q, Wu LF, Wang HG, Wang SH, Yan YX, Sun JH. Glycine cleavage system and cAMP receptor protein co-regulate CRISPR/cas3 expression to resist bacteriophage. Viruses, 2020, 12(1): 90. DOI:10.3390/v12010090 |
| [30] | Perez-Rodriguez R, Haitjema C, Huang QQ, Nam KH, Bernardis S, Ke AL, DeLisa MP. Envelope stress is a trigger of CRISPR RNA-mediated DNA silencing in Escherichia coli. Molecular Microbiology, 2011, 79(3): 584-599. DOI:10.1111/j.1365-2958.2010.07482.x |
| [31] | Pul Ü, Wurm R, Arslan Z, René geißen, Hofmann N, Wagner R. Identification and characterization of E. coli CRISPR-Cas promoters and their silencing by H-NS. Molecular Microbiology, 2010, 75(6): 1495-1512. DOI:10.1111/j.1365-2958.2010.07073.x |
| [32] | Sun DC, Mao XD, Fei MY, Chen ZY, Zhu TH, Qiu JP. Histone-like nucleoid-structuring protein (H-NS) paralogue StpA activates the type Ⅰ-E CRISPR-Cas system against natural transformation in Escherichia coli. Applied and Environmental Microbiology, 2020, 86(14): e00731-00720. |
| [33] | Westra ER, Pul Ü, Heidrich N, Jore MM, Lundgren M, Stratmann T, Wurm R, Raine A, Mescher M, Van Heereveld L, Mastop M, Wagner EGH, Schnetz K, Van Der Oost J, Wagner R, Brouns SJJ. H-NS-mediated repression of CRISPR-based immunity in Escherichia coli K12 can be relieved by the transcription activator LeuO. Molecular Microbiology, 2010, 77(6): 1380-1393. DOI:10.1111/j.1365-2958.2010.07315.x |
| [34] | Medina-Aparicio L, Rebollar-Flores JE, Gallego- Hernández AL, Vázquez A, Olvera L, Gutiérrez-Ríos RM, Calva E, Hernández-Lucas I. The CRISPR/Cas immune system is an operon regulated by LeuO, H-NS, and leucine-responsive regulatory protein in Salmonella enterica Serovar Typhi. Journal of Bacteriology, 2011, 193(10): 2396-2407. DOI:10.1128/JB.01480-10 |
| [35] | Yang CD, Chen YH, Huang HY, Huang HD, Tseng CP. CRP represses the CRISPR/Cas system in Escherichia coli: evidence that endogenous CRISPR spacers impede phage P1 replication. Molecular Microbiology, 2014, 92(5): 1072-1091. DOI:10.1111/mmi.12614 |
| [36] | Patterson AG, Jackson SA, Taylor C, Evans GB, Salmond GPC, Przybilski R, Staals RHJ, Fineran PC. Quorum sensing controls adaptive immunity through the regulation of multiple CRISPR-Cas systems. Molecular Cell, 2016, 64(6): 1102-1108. DOI:10.1016/j.molcel.2016.11.012 |
| [37] | Patterson AG, Chang JT, Taylor C, Fineran PC. Regulation of the Type Ⅰ-F CRISPR-Cas system by CRP-cAMP and GalM controls spacer acquisition and interference. Nucleic Acids Research, 2015, 43(12): 6038-6048. DOI:10.1093/nar/gkv517 |
| [38] | Høyland-Kroghsbo NM, Paczkowski J, Mukherjee S, Broniewski J, Westra E, Bondy-Denomy J, Bassler BL. Quorum sensing controls the Pseudomonas aeruginosa CRISPR-Cas adaptive immune system. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(1): 131-135. DOI:10.1073/pnas.1617415113 |
| [39] | Borges AL, Castro B, Govindarajan S, Solvik T, Escalante V, Bondy-Denomy J. Bacterial alginate regulators and phage homologs repress CRISPR-Cas immunity. Nature Microbiology, 2020, 5(5): 679-687. DOI:10.1038/s41564-020-0691-3 |
| [40] | Ahator SD, Wang JH, Zhang LH. The ECF sigma factor PvdS regulates the type Ⅰ-F CRISPR-Cas system in Pseudomonas aeruginosa. bioRxiv, 2020. DOI:10.1101/2020.01.31.929752 |
| [41] | Workman RE, Pammi T, Nguyen BTK, Graeff LW, Smith E, Sebald SM, Stoltzfus MJ, Euler CW, Modell JW. A natural single-guide RNA repurposes Cas9 to autoregulate CRISPR-Cas expression. Cell, 2021, 184(3): 675-688. DOI:10.1016/j.cell.2020.12.017 |
| [42] | Bernal-Bernal D, Abellón-Ruiz J, Iniesta AA, Pajares-Martínez E, Bastida-Martínez E, Fontes M, Padmanabhan S, Elías-Arnanz M. Multifactorial control of the expression of a CRISPR-Cas system by an extracytoplasmic function σ/anti-σ pair and a global regulatory complex. Nucleic Acids Research, 2018, 46(13): 6726-6745. DOI:10.1093/nar/gky475 |
| [43] | Sorek R, Lawrence CM, Wiedenheft B. CRISPR-mediated adaptive immune systems in bacteria and archaea. Annual Review of Biochemistry, 2013, 82: 237-266. DOI:10.1146/annurev-biochem-072911-172315 |
| [44] |
Sun DC, Qiu JP. Advances in molecular mechanisms of adaptive immunity mediated by type Ⅰ-E CRISPR/Cas system—a review. Acta Microbiologica Sinica, 2016, 56(1): 1-7.
(in Chinese) 孙东昌, 裘娟萍. Ⅰ-E型CRISPR/Cas系统介导适应性免疫分子机制研究进展. 微生物学报, 2016, 56(1): 1-7. |
| [45] | Xue CY, Sashital DG. Mechanisms of type Ⅰ-E and Ⅰ-F CRISPR-Cas systems in Enterobacteriaceae. EcoSal Plus, 2019, 8(2). |
| [46] | Arslan Z, Stratmann T, Wurm R, Wagner R, Schnetz K, Pul Ü. RcsB-BglJ-mediated activation of Cascade operon does not induce the maturation of CRISPR RNAs in E. coli K12. RNA Biology, 2013, 10(5): 708-715. DOI:10.4161/rna.23765 |
| [47] | Yosef I, Goren MG, Kiro R, Edgar R, Qimron U. High-temperature protein G is essential for activity of the Escherichia coli clustered regularly interspaced short palindromic repeats (CRISPR)/Cas system. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(50): 20136-20141. DOI:10.1073/pnas.1113519108 |
| [48] | Hampton HG, Patterson AG, Chang JT, Taylor C, Fineran PC. GalK limits type Ⅰ-F CRISPR-Cas expression in a CRP-dependent manner. FEMS Microbiology Letters, 2019, 366(11): fnz137. DOI:10.1093/femsle/fnz137 |
| [49] | Høyland-Kroghsbo NM, Muñoz KA, Bassler BL. Temperature, by controlling growth rate, regulates CRISPR-Cas activity in Pseudomonas aeruginosa. mBio, 2018, 9(6): e02184-18. |
| [50] | Lin P, Pu QQ, Wu Q, Zhou CM, Wang B, Schettler J, Wang ZH, Qin SG, Gao P, Li RP, Li GP, Cheng ZY, Lan LF, Jiang JX, Wu M. High-throughput screen reveals sRNAs regulating crRNA biogenesis by targeting CRISPR leader to repress Rho termination. Nature Communications, 2019, 10(1): 3728. DOI:10.1038/s41467-019-11695-8 |
| [51] | Le Rhun A, Escalera-Maurer A, Bratovič M, Charpentier E. CRISPR-Cas in Streptococcus pyogenes. RNA Biology, 2019, 16(4): 380-389. DOI:10.1080/15476286.2019.1582974 |
| [52] | Mosterd C, Moineau S. Characterization of a type Ⅱ-A CRISPR-Cas system in Streptococcus mutans. mSphere, 2020, 5(3). |
| [53] | Serbanescu MA, Cordova M, Krastel K, Flick R, Beloglazova N, Latos A, Yakunin AF, Senadheera DB, Cvitkovitch DG. Role of the Streptococcus mutans CRISPR-Cas systems in immunity and cell physiology. Journal of Bacteriology, 2015, 197(4): 749-761. DOI:10.1128/JB.02333-14 |
| [54] | Huang FT, Zhu B. The cyclic oligoadenylate signaling pathway of type Ⅲ CRISPR-Cas systems. Frontiers in Microbiology, 2020, 11: 602789. |
| [55] | Sun DC. Pull in and push out: mechanisms of horizontal gene transfer in bacteria. Frontiers in Microbiology, 2018, 9: 2154. DOI:10.3389/fmicb.2018.02154 |
| [56] | Guo MY, Wang HY, Xie NB, Xie ZX. Positive effect of carbon sources on natural transformation in Escherichia coli: role of low-level cyclic AMP (cAMP)-cAMP receptor protein in the derepression of rpoS. Journal of Bacteriology, 2015, 197(20): 3317-3328. DOI:10.1128/JB.00291-15 |
| [57] | Battesti A, Tsegaye YM, Packer DG, Majdalani N, Gottesman S. H-NS regulation of IraD and IraM antiadaptors for control of RpoS degradation. Journal of Bacteriology, 2012, 194(10): 2470-2478. DOI:10.1128/JB.00132-12 |
| [58] | Lucchini S, McDermott P, Thompson A, Hinton JCD. The H-NS-like protein StpA represses the RpoS (σ38) regulon during exponential growth of Salmonella Typhimurium. Molecular Microbiology, 2009, 74(5): 1169-1186. DOI:10.1111/j.1365-2958.2009.06929.x |
| [59] | 陈紫燕. 类组蛋白调控大肠杆菌摄取和免疫防御质粒机制研究. 浙江工业大学硕士论文, 2021. |
| [60] | Lu Y, Zeng JM, Wang LJ, Lan K, Shunmei E, Wang LN, Xiao Q, Luo Q, Huang XZ, Huang B, Chen C. Antibiotics promote Escherichia coli-Pseudomonas aeruginosa conjugation through inhibiting quorum sensing. Antimicrobial Agents and Chemotherapy, 2017, 61(12): e01284-01217. |
| [61] | Prudhomme M, Attaiech L, Sanchez G, Martin B, Claverys JP. Antibiotic stress induces genetic transformability in the human pathogen Streptococcus pneumoniae. Science, 2006, 313(5783): 89-92. DOI:10.1126/science.1127912 |
| [62] | Lin TL, Pan YJ, Hsieh PF, Hsu CR, Wu MC, Wang JT. Imipenem represses CRISPR-Cas interference of DNA acquisition through H-NS stimulation in Klebsiella pneumoniae. Scientific Reports, 2016, 6: 31644. DOI:10.1038/srep31644 |
| [63] | Han WY, Stella S, Zhang Y, Guo T, Sulek K, Peng-Lundgren L, Montoya G, She QX. A type Ⅲ-B Cmr effector complex catalyzes the synthesis of cyclic oligoadenylate second messengers by cooperative substrate binding. Nucleic Acids Research, 2018, 46(19): 10319-10330. |
| [64] | Kazlauskiene M, Kostiuk G, Venclovas Č, Tamulaitis G, Siksnys V. A cyclic oligonucleotide signaling pathway in type Ⅲ CRISPR-Cas systems. Science, 2017, 357(6351): 605-609. DOI:10.1126/science.aao0100 |
| [65] | Niewoehner O, Garcia-Doval C, Rostøl JT, Berk C, Schwede F, Bigler L, Hall J, Marraffini LA, Jinek M. Type Ⅲ CRISPR-Cas systems produce cyclic oligoadenylate second messengers. Nature, 2017, 548(7669): 543-548. DOI:10.1038/nature23467 |
| [66] | Jia N, Patel DJ. Structure-based functional mechanisms and biotechnology applications of anti-CRISPR proteins. Nature Reviews Molecular Cell Biology, 2021, 22(8): 563-579. DOI:10.1038/s41580-021-00371-9 |
| [67] | Athukoralage JS, McMahon SA, Zhang CY, Grüschow S, Graham S, Krupovic M, Whitaker RJ, Gloster TM, White MF. An anti-CRISPR viral ring nuclease subverts type Ⅲ CRISPR immunity. Nature, 2020, 577(7791): 572-575. DOI:10.1038/s41586-019-1909-5 |
| [68] | Liu ZZ, Sun MM, Liu JL, Liu T, Ye Q, Li YJ, Peng N. A CRISPR-associated factor Csa3a regulates DNA damage repair in Crenarchaeon Sulfolobus islandicus. Nucleic Acids Research, 2020, 48(17): 9681-9693. DOI:10.1093/nar/gkaa694 |
 2022, Vol. 62
2022, Vol. 62