中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 谭菲凡, 朱丽雯, 周成, 张桂敏, 贾开志. 2022
- TAN Feifan, ZHU Liwen, ZHOU Cheng, ZHANG Guimin, JIA Kaizhi.
- 微生物源甲硫氨酸γ-裂解酶的研究进展
- Research progress of methionine γ-lyase from microorganism
- 微生物学报, 62(2): 421-433
- Acta Microbiologica Sinica, 62(2): 421-433
-
文章历史
- 收稿日期:2021-04-22
- 修回日期:2021-06-04
- 网络出版日期:2021-06-11
2. 中国科学院微生物研究所, 微生物资源前期开发国家重点实验室, 北京 100101;
3. 北京化工大学生命科学与技术学院, 北京 100029
2. State Key Laboratory of Microbial Resources, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
3. College of Life Science and Technology, Beijing University of Chemical Technology, Beijing 100029, China
甲硫氨酸γ-裂解酶(methionine γ-lyase,MGL)是一种依赖5′-磷酸吡哆醛(PLP)、催化甲硫氨酸γ位C-S键裂解反应的功能酶;催化合成的产物为甲基硫醇、α-酮丁酸和氨,该酶促反应是构成甲硫氨酸分解的重要途径之一[1–3]。MGL广泛存在于多种微生物中,而在哺乳动物组织中却未发现[4]。挖掘微生物来源的MGL,通过食品添加MGL或者体内注射MGL以降低哺乳动物胞内甲硫氨酸浓度成为一项极具发展潜力治疗甲硫氨酸依赖肿瘤的重要手段[5–8]。正常细胞对胞内甲硫氨酸浓度降低具有一定的耐受性,且甲硫氨酸浓度降低将减少胞内活性氧的种类,延长正常细胞的寿命、提高抗胁迫、代谢适应以及抗氧化功能[7–8]。因此,利用MGL降低正常细胞胞内甲硫氨酸含量成为抗衰老研究的热点。MGL催化反应产物甲基硫醇进一步通过自由基反应快速合成二甲基硫、二甲基二硫和二甲基三硫等挥发性有机含硫化合物(VOSCs)[9–15]。VOSCs嗅阈值低、香味活性值高,是构成发酵食品香味的主体成分[9–10]。精细调控发酵微生物胞内MGL的表达是控制食品品质的关键环节。对于一些病原微生物而言,如引起口臭细菌牙龈卟啉单胞菌(Porphyromonas gingivalis)、齿密螺旋体(Treponema dentium)和连翘坦纳氏菌(Tannerella forsythia),其通过胞内的甲基氧化酶进一步将甲基硫醇转化成活性化合物甲醛、过氧化氢和硫化氢;而MGL催化产物氨是口臭气味的另外一个重要来源[16]。因此,从源头上开发MGL活性抑制剂是治疗口腔细菌感染的一个重要手段。此外,MGL也是作为治疗阴道毛滴虫(Trichomonas vaginalis)、溶组织内阿米巴(Entamoeba histolytica)等致病性微生物感染的药物靶标[2]。基于MGL及其催化产物在医药和食品领域的重要性,本文通过系统综述甲硫氨酸裂解酶的挖掘、催化机制和改造方面最新进展,为治疗多种恶性甲硫氨酸依赖性肿瘤、抗衰老和抗致病微生物感染等医疗方面提供优良的酶制剂或者酶抑制剂,同时推动VOSCs绿色制造技术的开发及其动态、精细化调控技术的研制。
1 MGL的挖掘Kreis等于1973年首次从生孢梭菌(Clostridium sporogenes)获得高纯度的MGL,并发现MGL具有抗肿瘤活性[17]。随后的研究从阴道毛滴虫、恶臭假单胞菌(Pseudomonas putida)[18]、溶组织内阿米巴(Entamoeba histolytica)、弗氏柠檬酸杆菌(Citrobacter freundii)[19]等微生物中纯化出天然甲硫氨酸裂解酶并解析了其结构(附表 1)。一些MGL具有较宽泛的底物杂泛性,例如来源于恶臭假单胞菌[18]、卵状假单胞菌(Pseudomonas ovalis)[20]、扩展短杆菌BL2 (Brevibacterium linens BL2)[21]、链霉菌(Streptomyces avermitilis)[22]、弗氏柠檬酸杆菌[23]和溶组织内阿米巴[24]的MGL能催化L-半胱氨酸及其衍生物β位C-S键的裂解反应。目前研究最为系统的是来源于恶臭假单胞菌的甲硫氨酸γ-裂解酶(pMGL),其对甲硫氨酸表现出较高的结合能力和催化能力,因此广泛应用于通过降低胞内甲硫氨酸浓度治疗恶性肿瘤的研究中[25–26]。
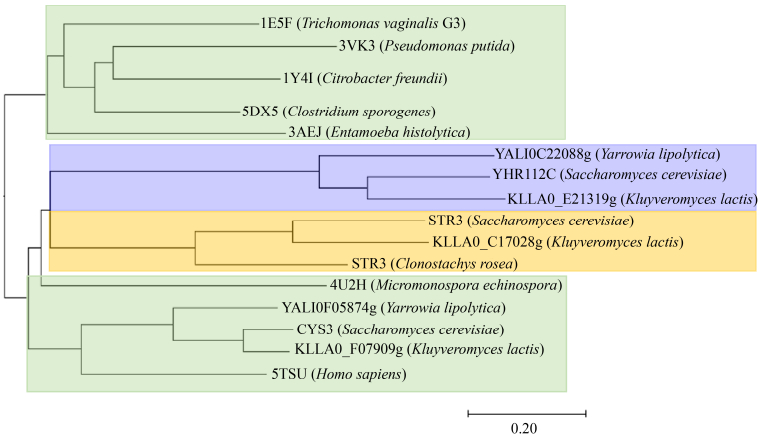
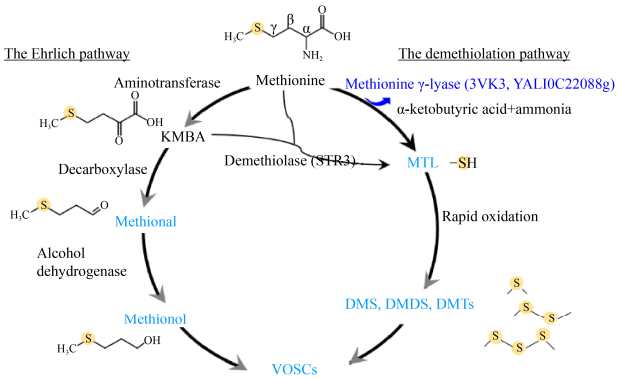
MGL从属于胱硫醚γ-裂解酶(cystathionine- γ-lyase,CGL)亚群,聚类分析发现原核和真核微生物来源的MGL分别聚类于2个不同的分支(图 1),值得注意的是来源于原生动物阴道毛滴虫的MGL与原核微生物的MGL聚类在一起,而来源于棘孢小单孢菌(Micromonospora echinospora)的MGL却位于真核胱硫醚γ-裂解酶亚群与胱硫醚β-裂解酶亚群之间,暗示微生物来源的MGL在自然进化过程中的多样性(图 1)。胱硫醚γ-裂解酶具有特异催化L-胱硫醚、L-甲硫氨酸、L-同型半胱氨酸等含硫氨基酸γ位C-S键裂解的功能[27]。然而模式生物酿酒酵母(Saccharomyces cerevisiae)中与原核MGL同源的功能酶CYS3却不能催化甲硫氨酸γ位C-S键的裂解反应[11];STR3被注释为胱硫醚β-裂解酶编码基因,体外功能鉴定其亦不具有裂解甲硫氨酸的功能[11, 28]。Jia等从名贵食用真菌黑孢块菌的子实体中分离到一株重寄生真菌红粉粘帚霉Tang19 (Clonostachys rosea Tang19),来源于红粉粘帚霉的STR3同源蛋白能够裂解甲硫氨酸合成甲基硫醇,但其活性很弱,底物谱测定表明STR3同源蛋白能够高效催化含硫化合物(L-djenkolate、L-胱氨酸、S-甲基-L-半胱氨酸) β位C-S键的裂解反应,推测STR3同源蛋白催化甲硫氨酸γ位C-S键裂解的功能与其杂泛性有关[29]。STR3同源蛋白能够分解甲硫氨酸转氨产物4-甲硫基-2-氧丁酸(KMBA)合成甲基硫醇等VOSCs组分[29]。这条路线需要两步酶促反应,没有一步分解甲硫氨酸合成甲基硫醇等VOSCs组分的路线更为经济、高效(图 2)。
|
| 图 1 不同来源MGL的进化分析 Figure 1 Evolutionary analysis of MGLs. PDB IDs or entry names from bacteria, yeast, protozoon and human were provided, cystathionine γ-lyase was marked in green, cystathionine β-lyase was marked in yellow, cystathionine γ-lyase identified recently in yeast was marked in blue. |
|
| 图 2 微生物分解甲硫氨酸合成VOSCs途径 Figure 2 The pathway for catabolizing L-methionine into VOSCs by microorganisms. 3VK3 is the PDB ID of pMGL from Pseudomonas putida, YALI0C22088g was a MGL from Y. lipolytica. |
本实验室前期通过途径分析挖掘酵母(酿酒酵母、解脂耶氏酵母(Yarrowia lipolytica)和乳酸克鲁维酵母(Kluyveromyces lactis))基因组中参与甲硫氨酸代谢的功能酶,发现未知功能蛋白YHR112C、KLLA0-E21319g和YALI0C22088g聚类距离胱硫醚β-裂解酶亚群较近,而距离胱硫醚γ-裂解酶亚群较远[9] (图 1)。体外功能鉴定发现来源于解脂耶氏酵母的YALI0C22088g具有一步分解甲硫氨酸合成甲基硫醇的活性,且活性明显高于来源于红粉粘帚霉的STR3同源蛋白;而来源于酿酒酵母的YHR112C具有微弱的裂解甲硫氨酸合成甲基硫醇活性,表明该聚类是一类新型的MGL[9]。
不同来源MGL的生化性质非常相似。MGL的氨基酸序列长度通常为400个氨基酸左右,序列之间具有明显的同源性。MGL的物理性质也较为相似,例如,最适pH在7.0–8.0之间,pH稳定性在5.0–9.0之间,最适温度为30 ℃。该酶在278 nm和420 nm处有吸收峰,在330 nm附近有一个肩峰,且吸收峰不随pH而改变。裂解酶为四聚体,含有一个较大的PLP结合结构域和较小的C-端结构域,其中辅因子PLP在静息态(resting stage)时与多肽链中心部分的赖氨酸(Lys)残基连接,形成内部Schiff碱[27, 30–32]。
2 MGL的催化机制利用计算化学和结构生物学等研究手段解析MGL与底物甲硫氨酸、辅因子PLP的相互作用方式,揭示MGL催化甲硫氨酸一步分解合成VOSCs的分子机制,可以为理性设计和改造MGL提供理论基础。
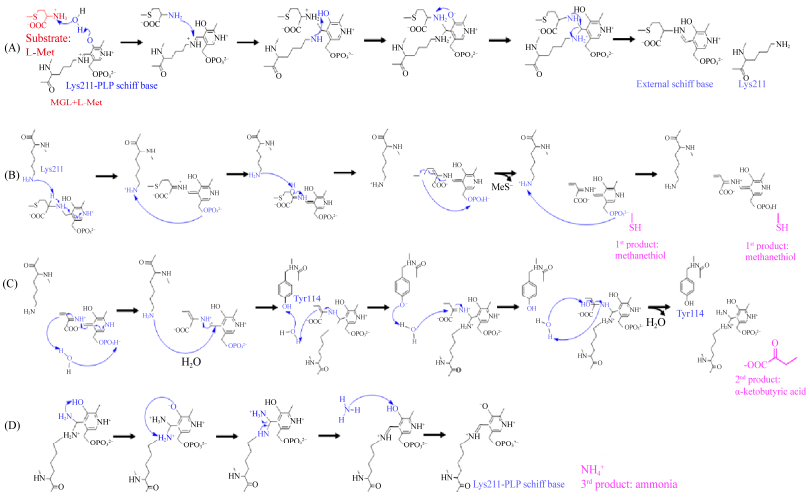
2.1 动力学模拟解析MGL的催化机制量子力学、分子力学结合伞形取样的研究结果表明,MGL与底物甲硫氨酸结合后经过4个阶段共19步反应,将甲硫氨酸裂解为甲基硫醇、α-酮丁酸和氨[33]。阶段1 (transamination reaction of PLP,PLP的转氨反应):是一个加成-消除反应,底物甲硫氨酸加成至CPLP,随后消除Lys211上的NLys,导致底物甲硫氨酸取代Lys211,甲硫氨酸与PLP之间形成外部Schiff碱。阶段2 (E1cB elimination of methyl mercaptan,E1cB消除反应):遵循去质子化-消除途径,即NLys和磷酸基团充当碱,去除甲硫氨酸Cα和Cβ的质子,反应中间体进一步通过E1cB机制消除MeS–。阶段3 (tautomerization and hydrolysis,互变异构化和水解):是一个耦合亲核加成—消除过程的水解反应,连接CPLP和PLP环的共价π-键被打断,Cα原子的亲电活性增强;当一个水分子攻击高亲电性的Cα即可发生亲核加成反应;Owat上的一个质子转移至NMet,NMet随即被从Cα原子消除,产生第二个产物α-酮丁酸。阶段4 (return to the resting state of the enzyme,恢复酶的静息态):NLys上的质子转移至来自甲硫氨酸的氨基基团NMet,导致NMet离去能力增强、随后被消除,产生氨(NH4+),酶恢复静息态(图 3)。PLP在酶促甲硫氨酸裂解反应开始时通过外部的醛亚胺结构与底物甲硫氨酸共价结合,继而通过共振效应稳定不同类型反应中间体的阴离子;最新的研究表明PLP还具有利用羟基基团和磷酸基团帮助质子转移或者临时储存一些中间体产生的额外质子的功能[33–34]。
|
| 图 3 动力学模拟MGL的催化机制 Figure 3 Kinetic simulation of catalytic mechanism for MGL. A: First stage of the reaction: transamination reaction of PLP; B: Second stage of the reaction: ElcB elimination of methyl mercaptan; C: Third stage of the reaction: tautomerization and hydrolysis; D: Fourth stage of the reaction: return to the resting state of the enzyme. |
2.2 控制MGL催化活性的关键氨基酸残基
酶的底物特异性和反应类型通常是由活性中心的一系列氨基酸残基决定,而不是结构上的折叠[35]。通过对YALI0C22088g与结构解析的胱硫醚γ-裂解酶CYS3 (PDB ID:1N8P,来源于酿酒酵母)[27]、甲硫氨酸裂解酶5TSU (PDB ID,人源胱硫醚γ-裂解酶CTH突变体)[36–38]和3VK3 (PDB ID,来源于恶臭假单胞菌C116)[18]以及拟南芥胱硫醚β-裂解酶1IBJ (PDB ID)[32]在氨基酸水平上的保守性进行分析,发现胱硫醚β-裂解酶与胱硫醚γ-裂解酶活性中心的氨基酸残基高度保守,暗示在活性中心之外存在决定裂解酶对底物C-S键裂解的区域选择性的关键因子。CYS3、人源胱硫醚γ-裂解酶CTH和3VK3同属胱硫醚γ-裂解酶亚群,然而CYS3和CTH均不具有裂解甲硫氨酸的功能。人源胱硫醚γ-裂解酶CTH的突变体5TSU能够高效裂解甲硫氨酸,比较CTH与5TSU等突变体结构上的差异,推测位于活性中心入口处loop的运动性增强、入口处于开放状态导致突变体高效裂解甲硫氨酸[36–38]。然而这段loop序列在YALI0C22088g以及其他裂解酶中并不保守[9]。3VK3具有分解甲硫氨酸的功能,其中H116控制C-S键裂解的区域选择特性,H116、K240和D241之间形成的氢键网络控制了裂解反应的底物特异性,H116、K240和D241特异存在于3VK3[18],在YALI0C22088g以及其他裂解酶中亦不保守。这些数据暗示不同来源的甲硫氨酸裂解酶控制底物特异性的分子机制可能存在很大差异。
底物甲硫氨酸需要通过入口和通道才能进入活性中心参与催化反应,对入口、通道和活性中心的氨基酸残基进行功能分析可以快速鉴定控制甲硫氨酸裂解酶底物选择性和催化活性的关键氨基酸残基。基于YALI0C22088g裂解甲硫氨酸的酶活是YHR112C的25.23倍,而KLLA0-E21319g不具有裂解甲硫氨酸功能[9],Zhao等提出假设:YALI0C22088g在入口、通道和活性中心与YHR112C、KLLA0-E21319g存在变异的氨基酸残基可能是控制YALI0C22088g催化活性的关键氨基酸残基[9]。通过对YALI0C22088g、YHR112C和KLLA0-E21319g氨基酸序列进化的保守性和变异性进行分析,在YALI0C22088g入口、通道和活性中心结构共找到12个变异氨基酸残基[9],利用丙氨酸扫描结合酶催化活性测定,共在YALI0C22088g入口和通道处找到8个关键氨基酸残基,其功能可能与底物特异结合、入口和通道瓶颈相关[9]。
底物位阻和构型也是影响MGL底物识别的重要因素。来源于卵状假单胞菌MGL不能催化胱氨酸和胱硫醚的裂解反应,对S-(β-氨基乙基)-L-半胱氨酸催化活性较弱[20]。推测这些底物结构中ω-羧基基团或者ω-氨基基团可能限制了MGL与底物的特异结合,阻碍了裂解反应的发生[20]。另外,MGL不能催化正亮氨酸、戊氨酸以及甲硫氨酸和半胱氨酸D异构体的裂解反应,表明该酶的活性中心只能与α碳为L构型的底物进行结合[16]。
3 MGL的改造及其应用MGL催化甲硫氨酸一步分解反应有效降低胞内甲硫氨酸浓度,是治疗甲硫氨酸依赖性癌症的一种极具发展潜力的方法[36–40]。MGL广泛存在于微生物中,而在哺乳动物中不存在,MGL是开发抗致病微生物感染药物的重要靶标[2, 16]。MGL催化合成的甲基硫醇等VOSCs组分是构成食品香味的主体物质,其组分和浓度决定了食品的品质[12–15]。通过改造MGL以及研究MGL活性调控机制将推动其在医疗健康和食品工业领域中的应用。
3.1 MGL在医疗健康中的应用甲硫氨酸而非其他氨基酸的浓度降低将阻碍胞内甲基化反应,抑制甲硫氨酸依赖性肿瘤细胞的生长和繁殖,进而破坏肿瘤干细胞的自发更新能力[41–44]。正常细胞对胞内甲硫氨酸浓度降低具有耐受能力,同时甲硫氨酸浓度降低将减少胞内活性氧的种类,从而延长正常细胞的寿命、提高抗胁迫、代谢适应以及抗氧化功能[7–8, 45]。注射MGL和食品添加MGL均有效降低甲硫氨酸浓度,且该方法用于癌症治疗不需要考虑药物跨越血脑屏障问题[36–38]。因此,MGL成为治疗癌症的一种极具发展潜力的酶制剂。
目前,用于癌症治疗研究最为深入的pMGL来自恶臭假单胞菌。模式动物体内试验表明pMGL能够降低血浆中甲硫氨酸浓度,严重抑制肿瘤生长。限制pMGL进一步应用的主要因素是其在血液中的稳定性很差、引发动物的高免疫反应[38]。利用酶工程改造pMGL将在一定程度上缓解上述问题[18, 46–47]。开发来源于人类的MGL可以克服pMGL的缺点[18, 46–47]。然而,人类组织中不存在天然MGL。人源CGL与pMGL在氨基酸水平具有61%的一致性,且具备结构相似性[38]。但是人源CGL却不具有裂解甲硫氨酸的功能。Stone等将CGL与来源于阴道毛滴虫的MGL进行蛋白结构上的覆盖分析,发现CGL侧链结构中E59、R119和E339可能是控制底物取向的3个关键氨基酸残基。Stone等利用组合成对饱和突变策略对CGL侧链结构中E59、R119和E339进行改造,通过比色法检测产物α-酮丁酸与3-甲基苯并噻唑啉酮腙的反应物,从文库中筛选获得具有催化甲硫氨酸γ位C-S键裂解功能的突变体(CGL (E59N、R119L、E339V))[38]。CGL (E59N、R119L、E339V)在血液中的稳定性和抗神经母细胞瘤的活性明显优于pMGL[38]。Lu等随后通过饱和突变结合高通量筛选方法将CGL (E59N,R119L,E339V) 升级改造为CGL (E59I/S63L/ L91M/R119A/K268R/T311G/E339V/I353S),新突变体的催化效率(kcat/Km)较CGL (E59N、R119L、E339V)提高了9倍[36]。模式动物体内试验表明新突变体的应用更为高效,只需要一次腹腔给药,就能将血液中甲硫氨酸浓度降低至75%以下,且维持时间超过3 d;同时,被试动物对血液中甲硫氨酸浓度降低表现出更好的耐受能力,且不会出现体重下降、肝酶异常以及其他毒性。血液中的甲硫氨酸浓度降低至阈值5 μmol/L左右时,肿瘤细胞将不能存活[38]。新突变体在处理第2天和第3天只能将血液甲硫氨酸浓度降低至17 μmol/L和26 μmol/L,杀死肿瘤细胞需要开发更为高效的甲硫氨酸裂解酶突变体,提高突变体结合和裂解甲硫氨酸的功能[36]。解脂耶氏酵母被认为是食品安全微生物,分离来自解脂耶氏酵母的甲硫氨酸裂解酶YALI0C22088g具有低免疫潜力,其结合甲硫氨酸底物的能力显著高于pMGL[9];将位于YALI0C22088g入口处的Y59突变成丙氨酸,YALI0C22088g通道瓶颈半径尺寸由1.5提高至2.0,突变体YALI0C22088g (Y59A)结合甲硫氨酸的功能明显高于CGL (E59I/S63L/L91M/R119A/K268R/T311G/E339V/ I353S)[9]。进一步通过酶工程改造其活性中心等关键结构,将有可能推动YALI0C22088g突变体在临床上的应用。
MGL是开发抗致病微生物感染药物的重要靶标,寻找抑制MGL酶活的化合物将推动治疗口腔细菌感染、滴虫病和阿米巴病药物的开发[2, 16]。炔丙基甘氨酸和3-氟-L-甲硫氨酸在体外和体内试验中均表现出高效抑制MGL的功能,其中3-氟-L-甲硫氨酸的作用机制归因于其γ位裂解产物硫代羰基二氟化物抑制了阴道毛滴虫、牙龈卟啉单胞菌、溶组织内阿米巴菌的生长[16, 48–49]。从密花树组织中提取获得的化合物麦西那酸B (myrsinoic acid B)显著抑制具核梭杆菌JCM8532 (Fusobacterium nucleatum JCM8532)、牙龈卟啉单胞菌W83以及对龋垢密螺旋体ATCC35405 (T. denticola ATCC35405) MGL的活性[50]。Batuev等人通过对商业化合物库进行筛选,获得两个抑制MGL活性的化合物((R)-5-胍基-2-(1-(4-甲苯磺酰氨基)环己烷甲酰胺)-戊酮酸和(N-(5-氨基-2-羟苯基)甲苯磺酰氨)[51]。最近的研究发现含硫氨基酸亚砜化合物是MGL的自杀式底物,这类物质与MGL作用生成产物硫代磺酸盐,硫代磺酸盐能够氧化MGL的Cys115,破坏Tyr113与Cys115的相互作用,从而导致Tyr113不能催化甲基硫醇的消除反应[52]。
3.2 MGL在食品工业中的应用潜力天然香味物质拥有巨大的市场需求,通过微生物发酵或者酶催化天然底物合成香味物质是天然香味物质的重要生产方式之一[53]。MGL分解甲硫氨酸合成的甲基硫醇及其衍生物是构成食品香味的主体成分。复杂微生物体系参与了食品发酵过程中甲硫氨酸的分解过程[12–15]。筛选生产VOSCs的优良菌株并对其进行代谢工程改造将快速推动食品风味的改良,提升食品品质。Sreekumar等将益生菌德氏乳杆菌ATCC4749 (Lactobacillus delbrueckii ATCC4797)、植物乳杆菌ATCC14917 (Lactobacillus plantarum ATCC14917)等菌株分别与半胱氨酸和甲硫氨酸混合培养,会发现不同菌株生产VOSCs的产量存在很大差异,其中植物乳杆菌299v和嗜酸乳杆菌NCFM (Lactobacillus acidophilus NCFM)比乳酸球菌MG1363 (Lactococcus lactis MG1363)能产生更多的VOSCs[54]。Hanniffy等通过在乳酸杆菌中异源表达亚麻短杆菌BL21的MGL基因,提高了L-甲硫氨酸、L-半胱氨酸、L-胱硫氨酸和L-胱氨酸等含硫化合物的降解效率,甲基硫醇的生成量远远高于其氧化衍生物(二甲基二硫和二甲基三硫)[21]。鉴于乳酸菌在发酵食品风味开发中的重要意义,调控乳酸菌生理代谢改善发酵乳制品的风味和质地不仅能满足人们消费需求,更能提高我国发酵剂产业的竞争力。因此,使用具有提高VOSCs合成效率的乳酸菌是促进乳酪风味开发的有效途径之一。
甲基硫醇等VOSCs组分和浓度决定了食品的品质,通过控制MGL的表达精确调控VOSCs的合成是控制食品香味的关键环节。Zhang等利用抑制消减杂交技术构建了红粉粘帚霉cDNA文库,用于筛选外源添加甲硫氨酸引发VOSCs生物合成的调控因子,发现过表达E3泛素-蛋白连接酶HUWE1显著增强了甲基硫醇及其衍生物的生物合成,而完全抑制了竞争途径埃利希(Ehrlich)途径产物的合成,实现了对两条通路的方向性调节,研究结果暗示VOSCs的生物合成在转录水平存在复杂的调控机制[55]。转录因子通过影响RNA聚合酶与启动子的结合介导合成酶基因在转录水平的表达,是控制合成酶基因在转录水平表达的关键环节。Xu等通过生物信息学分析结合启动子上游序列的突变和缺失,发现GLN3结合位点(GLN3 binding site,–512/–508)、MSN2基序(MSN2 motif,–1 160/–1 156)和MSN2基序(–1 255/–1 251)分别是控制脱巯基途径主效酶基因STR3及其竞争途径氨基转移酶基因ARO8-2、脱羧酶基因PDC在转录水平表达的关键上游调控元件;MSN2基序和GLN3结合位点是位于氮阻遏信号系统下游的两个关键启动子调节元件,据此推测VOSCs的生物合成受氮阻遏信号系统调控[56]。解脂耶氏酵母具有分解甲硫氨酸合成VOSCs的功能,对奶酪典型风味的形成发挥关键作用[9]。精细控制解脂耶氏酵母发酵产品品质,需要阐明外源甲硫氨酸触发合成酶基因在转录水平表达的分子机制。来源于解脂耶氏酵母的甲硫氨酸裂解酶YALI0C22088g编码基因响应外源甲硫氨酸添加在转录水平出现上调[9],该现象与之前在红粉粘帚霉中研究获得的结果(脱巯基酶STR3编码基因响应外源甲硫氨酸添加而在转录水平的表达出现下调)相反,推测解脂耶氏酵母中甲硫氨酸直接分解合成甲基硫醇及其衍生物途径可能存在新的调控机制[9]。解析甲硫氨酸分解合成VOSCs途径酶基因在转录和转录后水平的表达调控机制,将推动VOSCs组分和浓度精细调控技术的开发,为食品香味的精确调控提供理论基础。
4 展望近70年来,研究人员在MGL的挖掘、酶学性质、催化机制及应用等方面进行了大量的探索,MGL在癌症治疗、抗衰老以及食品香味合成和制造方面表现出巨大的应用潜力。pMGL在癌症治疗研究中表现出的问题主要为半衰期短、引起机体强烈的免疫反应[36–38]。通过酶工程改造人源MGL有效解决了上述问题,模式动物体内试验显示改造后的人源MGL显著抑制肿瘤细胞的生长,且未表现出毒性[36]。杀死肿瘤细胞、减少酶制剂的使用量需要进一步通过酶工程提高人源MGL结合和裂解甲硫氨酸的功能,或者挖掘并改造其他具有低免疫潜力的MGL。降低机体从环境中摄取外源甲硫氨酸能够延长机体寿命,利用MGL降解环境中的甲硫氨酸需要解决MGL在不同复杂环境中的稳定性和保持高催化活性问题。MGL是设计抗致病微生物感染药物的重要靶标,基于不同来源的MGL特异识别底物的机制存在很大差异,今后需要利用计算化学和结构生物学手段解析不同致病微生物MGL特异识别底物的分子机制,进而理性设计靶向特定MGL的抑制剂,达到特异拮抗致病微生物的目的。MGL作用底物甲硫氨酸是合成机体胞内调控分子S-腺苷甲硫氨酸的前体。近期研究表明Covid-19 RNA帽子结构的甲基化依赖宿主胞内S-腺苷甲硫氨酸的浓度,因此利用MGL降低宿主胞内甲硫氨酸浓度可能成为治疗新冠病毒感染的一种辅助手段[57]。MGL催化甲硫氨酸分解产物甲基硫醇及其衍生物是构成食品香味的主体成分,其组分和浓度决定了食品品质和稳定性,解析食品微生物中MGL在转录和转录后水平的调控机制将推动食品品质的精准控制;另外,进一步利用酶工程提高MGL的催化活性也将推动食品香味绿色制造技术的开发。
随着合成生物学和计算化学的发展,研究人员在理解MGL催化甲硫氨酸裂解的具体机制基础上,通过理性设计MGL入口、通道和活性中心等关键结构,将进一步增强MGL对底物L-甲硫氨酸的特异性识别,提高酶促反应效率。研究结果将推动MGL在癌症治疗、抗衰老、抗致病微生物感染、抗新冠病毒以及食品香味合成和制造等方面的应用。
补充材料
附表 1 不同微生物来源的甲硫氨酸γ-裂解酶的性质
本文补充材料见网络版http://journals.im.ac.cn/actamicrocn。补充材料为作者提供的原始数据,作者对其学术质量和内容负责。
| [1] | Raboni S, Revtovich S, Demitri N, Giabbai B, Storici P, Cocconcelli C, Faggiano S, Rosini E, Pollegioni L, Galati S, Buschini A, Morozova E, Kulikova V, Nikulin A, Gabellieri E, Cioni P, Demidkina T, Mozzarelli A. Engineering methionine γ-lyase from Citrobacter freundii for anticancer activity. Biochimica et Biophysica Acta: BBA - Proteines and Proteomics, 2018, 1866(12): 1260-1270. DOI:10.1016/j.bbapap.2018.09.011 |
| [2] | Foo TC, Terentis AC, Venkatachalam KV. A continuous spectrophotometric assay and nonlinear kinetic analysis of methionine γ-lyase catalysis. Analytical Biochemistry, 2016, 507: 21-26. DOI:10.1016/j.ab.2016.05.010 |
| [3] | El-Sayed AS. Microbial l-methioninase: production, molecular characterization, and therapeutic applications. Applied Microbiology and Biotechnology, 2010, 86(2): 445-467. DOI:10.1007/s00253-009-2303-2 |
| [4] | Morozova EA, Kulikova VV, Rodionov AN, Revtovich SV, Anufrieva NV, Demidkina TV. Engineered Citrobacter freundii methionine γ-lyase effectively produces antimicrobial thiosulfinates. Biochimie, 2016, 128/129: 92-98. DOI:10.1016/j.biochi.2016.07.007 |
| [5] | Pokrovsky VS, Yu Anisimova N, Zh Davydov D, Bazhenov SV, Bulushova NV, Zavilgelsky GB, Kotova VY, Manukhov IV. Methionine gamma lyase from Clostridium sporogenes increases the anticancer effect of doxorubicin in A549 cells and human cancer xenografts. Investigational New Drugs, 2019, 37(2): 201-209. DOI:10.1007/s10637-018-0619-4 |
| [6] | Strekalova E, Malin D, Good DM, Cryns VL. Methionine deprivation induces a targetable vulnerability in triple-negative breast cancer cells by enhancing TRAIL receptor-2 expression. Clinical Cancer Research, 2015, 21(12): 2780-2791. DOI:10.1158/1078-0432.CCR-14-2792 |
| [7] | Lee BC, Kaya A, Ma SM, Kim G, Gerashchenko MV, Yim SH, Hu Z, Harshman LG, Gladyshev VN. Methionine restriction extends lifespan of Drosophila melanogaster under conditions of low amino-acid status. Nature Communications, 2014, 5: 3592. DOI:10.1038/ncomms4592 |
| [8] | Ruckenstuhl C, Netzberger C, Entfellner I, Carmona-Gutierrez D, Kickenweiz T, Stekovic S, Gleixner C, Schmid C, Klug L, Sorgo AG, Eisenberg T, Büttner S, Mariño G, Koziel R, Jansen-Dürr P, Fröhlich KU, Kroemer G, Madeo F. Lifespan extension by methionine restriction requires autophagy-dependent vacuolar acidification. PLoS Genetics, 2014, 10(5): e1004347. DOI:10.1371/journal.pgen.1004347 |
| [9] | Zhao QL, Wang ZL, Yang L, Zhang S, Jia KZ. YALI0C22088g from Yarrowia lipolytica catalyses the conversion of L-methionine into volatile organic sulfur-containing compounds. Microbial Biotechnology, 2021: 00. DOI:10.1111/1751–7915.13796 |
| [10] | Hébert A, Forquin-Gomez MP, Roux A, Aubert J, Junot C, Heilier JF, Landaud S, Bonnarme P, Beckerich JM. New insights into sulfur metabolism in yeasts as revealed by studies of Yarrowia lipolytica. Applied and Environmental Microbiology, 2013, 79(4): 1200-1211. DOI:10.1128/AEM.03259-12 |
| [11] | Holt S, Cordente AG, Williams SJ, Capone DL, Jitjaroen W, Menz IR, Curtin C, Anderson PA. Engineering Saccharomyces cerevisiae to release 3-mercaptohexan-1-ol during fermentation through overexpression of an S. cerevisiae gene, STR3, for improvement of wine aroma. Applied and Environmental Microbiology, 2011, 77(11): 3626-3632. DOI:10.1128/AEM.03009-10 |
| [12] | Liu J, Wu Q, Wang P, LinJC, Huang L, Xu Y. Synergistic effect in core microbiota associated with sulfur metabolism in spontaneous Chinese liquor fermentation. Applied and Environmental Microbiology, 2017, 83(24): e01475-e01417. |
| [13] | Splivallo R, Ottonello S, Mello A, Karlovsky P. Truffle volatiles: from chemical ecology to aroma biosynthesis. New Phytologist, 2011, 189(3): 688-699. DOI:10.1111/j.1469-8137.2010.03523.x |
| [14] | Dzialo MC, Park R, Steensels J, Lievens B, Verstrepen KJ. Physiology, ecology and industrial applications of aroma formation in yeast. FEMS Microbiology Reviews, 2017, 41(Supp_1): S95-S128. |
| [15] | Heitmann M, Zannini E, Arendt E. Impact of Saccharomyces cerevisiae metabolites produced during fermentation on bread quality parameters: a review. Critical Reviews in Food Science and Nutrition, 2018, 58(7): 1152-1164. DOI:10.1080/10408398.2016.1244153 |
| [16] | Kandalam U, Ledra N, Laubach H, Venkatachalam KV. Inhibition of methionine gamma lyase deaminase and the growth of Porphyromonas gingivalis: a therapeutic target for halitosis/periodontitis. Archives of Oral Biology, 2018, 90: 27-32. DOI:10.1016/j.archoralbio.2018.02.022 |
| [17] | Kreis W, Hession C. Biological effects of enzymatic deprivation of L-methionine in cell culture and an experimental tumor. Cancer Research, 1973, 33(8): 1866-1869. |
| [18] | Fukumoto M, Kudou DZ, Murano S, Shiba T, Sato D, Tamura T, Harada S, Inagaki K. The role of amino acid residues in the active site of L-methionine γ-lyase from Pseudomonas putida. Bioscience, Biotechnology, and Biochemistry, 2012, 76(7): 1275-1284. DOI:10.1271/bbb.110906 |
| [19] | Mamaeva DV, Morozova EA, Nikulin AB, Revtovich SV, Nikonsov SV, Garber MB, Demidkina TV. Structure of Citrobacter freundii L-methionine gamma-lyase. Acta Crystallographica Section F Structual Biology Communications, 2005, F61: 546-549. |
| [20] | Tanaka H, Esaki N, Soda K. Properties of L-methionine gamma-lyase from Pseudomonas ovalis. Biochemistry, 1977, 16(1): 100-106. DOI:10.1021/bi00620a016 |
| [21] | Hanniffy SB, Philo M, Peláez C, Gasson MJ, Requena T, Martínez-Cuesta MC. Heterologous production of methionine-gamma-lyase from Brevibacterium linens in Lactococcus lactis and formation of volatile sulfur compounds. Applied and Environmental Microbiology, 2009, 75(8): 2326-2332. DOI:10.1128/AEM.02417-08 |
| [22] | Kudou DZ, Yasuda E, Hirai Y, Tamura T, Inagaki K. Molecular cloning and characterization of L-methionine γ-lyase from Streptomyces avermitilis. Journal of Bioscience and Bioengineering, 2015, 120(4): 380-383. DOI:10.1016/j.jbiosc.2015.02.019 |
| [23] | Revtovich SV, Faleev NG, Morozova EA, Anufrieva NV, Nikulin AD, Demidkina TV. Crystal structure of the external aldimine of Citrobacter freundii methionine γ-lyase with glycine provides insight in mechanisms of two stages of physiological reaction and isotope exchange of α-and β-protons of competitive inhibitors. Biochimie, 2014, 101: 161-167. DOI:10.1016/j.biochi.2014.01.007 |
| [24] | Sato D, Shiba T, Karaki T, Yamagata W, Nozaki T, Nakazawa T, Harada S. X-ray snapshots of a pyridoxal enzyme: a catalytic mechanism involving concerted [1, 5]-hydrogen sigmatropy in methionine γ-lyase. Scientific Reports, 2017, 7: 4874. DOI:10.1038/s41598-017-05032-6 |
| [25] | Yang ZJ, Wang JH, Lu Q, Xu JB, Kobayashi Y, Takakura T, Takimoto A, Yoshioka T, Lian CG, Chen CM, Zhang DD, Zhang Y, Li SK, Sun XH, Tan YY, Yagi S, Frenkel EP, Hoffman RM. PEGylation confers greatly extended half-life and attenuated immunogenicity to recombinant methioninase in primates. Cancer Research, 2004, 64(18): 6673-6678. DOI:10.1158/0008-5472.CAN-04-1822 |
| [26] | Sun XH, Yang ZJ, Li SK, Tan YY, Zhang N, Wang XE, Yagi S, Yoshioka T, Takimoto A, Mitsushima K, Suginaka A, Frenkel EP, Hoffman RM. In vivo efficacy of recombinant methioninase is enhanced by the combination of polyethylene glycol conjugation and pyridoxal 5′-phosphate supplementation. Cancer Research, 2003, 63(23): 8377-8383. |
| [27] | Messerschmidt A, Worbs M, Steegborn C, Wahl MC, Huber R, Laber B, Clausen T. Determinants of enzymatic specificity in the cys-met-metabolism PLP-dependent enzyme family: crystal structure of cystathionine γ-lyase from yeast and intrafamiliar structure comparison. Biological Chemistry, 2003, 384(3): 373-386. |
| [28] | Holt S, Cordente AG, Curtin C. Saccharomyces cerevisiae STR3 and yeast cystathionine β-lyase enzymes. Bioengineered, 2012, 3(3): 180-182. DOI:10.4161/bbug.19566 |
| [29] | Jia KZ, Zhang Q, Sun LY, Xu YH, Li HM, Tang YJ. Clonostachys rosea demethiolase STR3 controls the conversion of methionine into methanethiol. Scientific Reports, 2016, 6: 21920. DOI:10.1038/srep21920 |
| [30] | Filipovic MR, Zivanovic J, Alvarez B, Banerjee R. Chemical biology of H2S signaling through persulfidation. Chemical Reviews, 2018, 118(3): 1253-1337. |
| [31] | Sagong HY, Kim B, Joo S, Kim KJ. Structural and functional characterization of cystathionine γ-lyase from Bacillus cereus ATCC 14579. Journal of Agricultural and Food Chemistry, 2020, 68(51): 15267-15274. DOI:10.1021/acs.jafc.0c06503 |
| [32] | Breitinger U, Clausen T, Ehlert S, Huber R, Laber B, Schmidt F, Pohl E, Messerschmidt A. The three-dimensional structure of cystathionine beta-lyase from Arabidopsis and its substrate specificity. Plant Physiology, 2001, 126(2): 631-642. DOI:10.1104/pp.126.2.631 |
| [33] | Li Z, Zhao YS, Zhou HF, Luo HB, Zhan CG. Catalytic roles of coenzyme pyridoxal-5'-phosphate (PLP) in PLP-dependent enzymes: reaction pathway for methionine-γ-lyase-catalyzed L-methionine depletion. ACS Catalysis, 2020, 10(3): 2198-2210. DOI:10.1021/acscatal.9b03907 |
| [34] | Toney MD. Controlling reaction specificity in pyridoxal phosphate enzymes. Biochimica et Biophysica Acta: BBA - Proteines and Proteomics, 2011, 1814(11): 1407-1418. |
| [35] | Song HG, Xu R, Guo ZH. Identification and characterization of a methionine γ-lyase in the calicheamicin biosynthetic cluster of Micromonospora echinospora. ChemBioChem, 2015, 16(1): 100-109. DOI:10.1002/cbic.201402489 |
| [36] | Lu WC, Saha A, Yan WP, Garrison K, Lamb C, Pandey R, Irani S, Lodi A, Lu XY, Tiziani S, Zhang YJ, Georgiou G, DiGiovanni J, Stone E. Enzyme-mediated depletion of serum l-Met abrogates prostate cancer growth via multiple mechanisms without evidence of systemic toxicity. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(23): 13000-13011. DOI:10.1073/pnas.1917362117 |
| [37] | Yan WP, Stone E, Zhang YJ. Structural snapshots of an engineered cystathionine-γ-lyase reveal the critical role of electrostatic interactions in the active site. Biochemistry, 2017, 56(6): 876-885. DOI:10.1021/acs.biochem.6b01172 |
| [38] | Stone E, Paley O, Hu J, Ekerdt B, Cheung NK, Georgiou G. De novo engineering of a human cystathionine-γ-lyase for systemic L-methionine depletion cancer therapy. ACS Chemical Biology, 2012, 7(11): 1822-1829. DOI:10.1021/cb300335j |
| [39] | Sharma B, Singh S, Kanwar SS. L-methionase: a therapeutic enzyme to treat malignancies. BioMed Research International, 2014, 2014: 506287. |
| [40] | Chaturvedi S, Hoffman RM, Bertino JR. Exploiting methionine restriction for cancer treatment. Biochemical Pharmacology, 2018, 154: 170-173. |
| [41] | Wang ZX, Yip LY, Lee JHJ, Wu ZW, Chew HY, Chong PKW, Teo CC, Ang HYK, Peh KLE, Yuan J, Ma SM, Choo LSK, Basri N, Jiang X, Yu Q, Hillmer AM, Lim WT, Lim TKH, Takano A, Tan EH, Tan DSW, Ho YS, Lim B, Tam WL. Methionine is a metabolic dependency of tumor-initiating cells. Nature Medicine, 2019, 25(5): 825-837. |
| [42] | Miao YX, Yang H, Levorse J, Yuan SP, Polak L, Sribour M, Singh B, Rosenblum MD, Fuchs E. Adaptive immune resistance emerges from tumor-initiating stem cells. Cell, 2019, 177(5): 1172-1186.e14. |
| [43] | Batlle E, Clevers H. Cancer stem cells revisited. Nature Medicine, 2017, 23(10): 1124-1134. |
| [44] | Shiraki N, Shiraki Y, Tsuyama T, Obata F, Miura M, Nagae G, Aburatani H, Kume K, Endo F, Kume S. Methionine metabolism regulates maintenance and differentiation of human pluripotent stem cells. Cell Metabolism, 2014, 19(5): 780-794. |
| [45] | Yin J, Ren WK, Chen S, Li YY, Han H, Gao J, Liu G, Wu X, Li TJ, Woo Kim S, Yin YL. Metabolic regulation of methionine restriction in diabetes. Molecular Nutrition & Food Research, 2018, 62(10): 1700951. |
| [46] | Sato D, Shiba T, Yunoto S, Furutani K, Fukumoto M, Kudou DZ, Tamura T, Inagaki K, Harada S. Structural and mechanistic insights into homocysteine degradation by a mutant of methionine γ-lyase based on substrate-assisted catalysis. Protein Science, 2017, 26(6): 1224-1230. |
| [47] | Kudou DZ, Misaki S, Yamashita M, Tamura T, Esaki N, Inagaki K. The role of cysteine116 in the active site of the antitumor enzyme L-methionine γ-lyase from Pseudomonas putida. Bioscience, Biotechnology, and Biochemistry, 2008, 72(7): 1722-1730. |
| [48] | Coombs GH, Mottram JC. Trifluoromethionine, a prodrug designed against methionine γ-lyase-containing pathogens, has efficacy in vitro and in vivo against Trichomonas vaginalis. Antimicrobial Agents and Chemotherapy, 2001, 45(6): 1743-1745. |
| [49] | Sato D, Kobayashi S, Yasui H, Shibata N, Toru T, Yamamoto M, Tokoro G, Ali V, Soga T, Takeuchi T, Suematsu M, Nozaki T. Cytotoxic effect of amide derivatives of trifluoromethionine against the enteric protozoan parasite Entamoeba histolytica. International Journal of Antimicrobial Agents, 2010, 35(1): 56-61. |
| [50] | Ito S, Narise A, Shimura S. Identification of a methioninase inhibitor, myrsinoic acid B, from Myrsine seguinii Lév., and its inhibitory activities. Bioscience, Biotechnology, and Biochemistry, 2008, 72(9): 2411-2414. |
| [51] | Batuev EA, Lisunov AY, Morozova EA, Klochkov VV, Anufrieva NV, Demidkina TV, Polshakov VI. NMR screening of potential inhibitors of methionine γ-lyase from Citrobacter freundii. Molecular Biology, 2014, 48(6): 896-905. |
| [52] | Revtovich S, Morozova E, Kulikova V, Koval V, Anufrieva N, Nikulin A, Demidkina T. Sulfoxides of sulfur-containing amino acids are suicide substrates of Citrobacter freundii methionine γ-lyase. Structural bases of the enzyme inactivation. Biochimie, 2020, 168: 190-197. |
| [53] | Che YX, Yin S, Wang H, Yang HQ, Xu RX, Wang Q, Wu YP, Boutet J, Huet R, Wang CT. Production of methionol from 3-methylthiopropionaldehyde by catalysis of the yeast alcohol dehydrogenase Adh4p. Journal of Agricultural and Food Chemistry, 2020, 68(16): 4650-4656. |
| [54] | Sreekumar R, Al-Attabi Z, Deeth HC, Turner MS. Volatile sulfur compounds produced by probiotic bacteria in the presence of cysteine or methionine. Letters in Applied Microbiology, 2009, 48(6): 777-782. |
| [55] | Zhang Q, Jia KZ, Xia ST, Xu YH, Liu RS, Li HM, Tang YJ. Regulating Ehrlich and demethiolation pathways for alcohols production by the expression of ubiquitin-protein ligase gene HUWE1. Scientific Reports, 2016, 6: 20828. |
| [56] | Xu YH, Jia KZ, Tang YJ. Regulatory networks governing methionine catabolism into volatile organic sulfur-containing compounds in Clonostachys rosea. Applied and Environmental Microbiology, 2018, 84(22): e01840-18. |
| [57] | Hoffman RM, Han QH. Oral methioninase for Covid-19 methionine-restriction therapy. In vivo: Athens, Greece, 2020, 34(3 Suppl): 1593-1596. |
 2022, Vol. 62
2022, Vol. 62