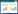中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 李兴, 欧一新, 康前进, 白林泉, 邓子新. 2021
- Xing Li, Yixin Ou, Qianjin Kang, Linquan Bai, Zixin Deng. 2021
- 抗细菌药物默诺霉素的化学生物学研究进展
- Advances in chemical and biological research of the antibacterial drug moenomycins
- 微生物学报, 61(7): 1839-1855
- Acta Microbiologica Sinica, 61(7): 1839-1855
-
文章历史
- 收稿日期:2020-08-01
- 修回日期:2020-10-13
- 网络出版日期:2021-02-03
2. 上海交通大学代谢与发育国际联合合作实验室, 上海 200040
2. Joint International Research Laboratory of Metabolic & Developmental Sciences, Shanghai Jiao Tong University, Shanghai 200240, China
默诺霉素(moenomycin)又被称为黄霉素(flavomycin)、黄磷脂醇(flavophospholipid)和斑堡霉素(bambermycin),是由加纳链霉菌(Streptomyces ghanaensis)、斑堡链霉菌(Streptomyces bambergiensis)、喷泉链霉菌(Streptomyces geysiriensis)和埃德尔链霉菌(Streptomycesederensis)等放线菌产生的寡糖类抗生素[1]。许多寡糖类抗生素及其衍生物具有重要的生理活性,例如:阿卡波糖是重要的α-葡萄糖苷酶抑制剂[2],阿泊拉霉素对多种革兰氏阳性菌有抑制作用[3-4],假寡糖类井冈霉素是防治水稻纹枯病的重要农药等[5-6]。默诺霉素家族抗生素的化学结构含有1个3-磷酸甘油酸单元、4–5个糖基和1个特殊的异戊烯基脂肪链,也被称为磷糖脂类抗生素(图 1)。该类化合物可以直接抑制细菌细胞壁生物合成中的肽聚糖糖基转移酶(PGTs),对众多的革兰氏阳性细菌均具有较好的杀灭或抑制作用,针对有些菌株的最低抑菌浓度(MIC)仅为10–100 ng/mL,生物学活性是万古霉素的约近10–1000倍[7],具有重要的应用开发潜力。
|
| 图 1 默诺霉素家族类化合物的化学结构 Figure 1 The chemical structures of moenomycins. |
抗生素的大量使用以及病原微生物的不断进化,导致新型感染性疾病不断出现,抗生素的耐药性现象日益严重[8],因而高效抑菌抗生素的研究和开发倍受关注。默诺霉素类抗生素的化学结构复杂新颖,而且在已经发现的10多种天然磷糖脂类化合物中,许多成员都具有显著的抗菌活性和一定的抗肿瘤活性,是抗生素新药研发的热点化合物。随着对默诺霉素类抗生素研究的不断深入,该类抗生素抑制细菌生长的分子机制和细菌耐受机制得到了较为详细的解析。该类抗生素的生物合成基因簇也已被克隆,其详细的生物合成机制也得到一定解析。这些系统的化学生物学研究,不仅为它们的药物研发提供了理论依据,也为利用组合生物合成或合成生物学技术实现高活性默诺霉素类抗生素的定向结构创制奠定了基础。近年来,我们课题组也针对默诺霉素的耐受性、生物合成及异源生产开展了相关的研究工作。本文对默诺霉素类抗生素的化学结构特征、生物活性机制、生物合成研究进展、外排机制以及默诺霉素新结构创制等方面进行了系统综述,为默诺霉素类抗生素临床药物的研究与开发提供了借鉴。
1 默诺霉素的结构特征与药效关系 1.1 默诺霉素的结构特征默诺霉素A (moenomycin A)(图 1)是1965年在加纳链霉菌中,被发现的第一个默诺霉素家族化合物,其结构也被进行了详细的鉴定[9]。随后,研究者又通过对相关链霉菌发酵产物的分离纯化,得到了其他多种默诺霉素类的组分[10-11]。默诺霉素类家族的化合物(moenomycins)是典型的磷酸糖脂类抗生素[1],包含了一个磷酸甘油酸脂肪链和经过修饰的B-C-E-F四个来源于葡萄糖或半乳糖糖基的单元。主体结构中B-C-E-F四个糖基通过1–4或1–2糖苷键相连,在F糖基的C-1位,通过磷脂键连接了一个3-磷酸甘油酸(3-phosphoglyceric acid,3-PG),在3-PG的C-2羟基上还连接了一个异戊二烯为结构单元的特殊脂肪链(图 1)。默诺霉素家族化合物的结构多样性主要来自于B-C-E-F四个糖基上R1、R2、R3、R4和R5 5个取代基的变化,包括:在大多数默诺霉素家族化合物中,半乳糖醛酸单位B环的C-6位羧酸基既可以与2-氨基环戊烷-1, 3-二酮(C5N)发色团单位A环连接,也可以转化为羧基或被氨基取代;糖单位C环和E环的结构可以是N-乙酰氨基葡萄糖或它的6-脱氧衍生物N-乙酰氨基半乳糖;E糖基环C-6位R3基团可被葡萄糖单位D环或羟基取代。烯脲酰胺F环C-4位的立体化学结构为D-半乳糖结构或D-葡萄糖结构,后者有一个轴向甲基。根据R1、R2、R3、R4和R5 5个取代基的不同,默诺霉素类化合物又分成:R1基团被2-氨基环戊烷-1, 3-二酮(C5N)发色团单位A环取代的moenomycin系列化合物、pholipomycin和从Actinomyces sp. AC326菌株中分离得到的AC326-α;以及R1基团不被A环取代而修饰以羟基或氨基的nosokomycin系列化合物。在默诺霉素类抗生素中糖基单元和磷酸甘油酸脂链的完整性对该类化合物发挥生物活性具有重要的作用。
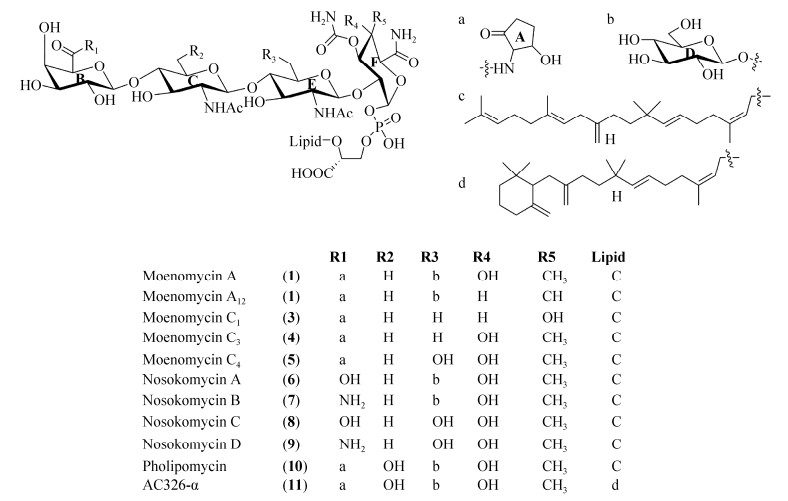
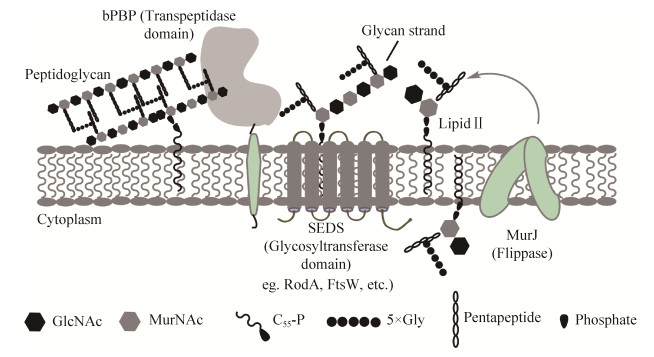
1.2 默诺霉素的活性作用机制默诺霉素家族抗生素是通过抑制革兰氏阳性细菌细胞壁的生物合成,来发挥其杀菌或抑菌作用。革兰氏阳性细菌细胞壁的主要成分是肽聚糖(peptidoglycan,PG),由N-乙酰氨基葡萄糖(N-acetylglucosamine,GlcNAc)和N-乙酰胞壁酸(N-acetylmuramic acid,MurNAc)形成了一种包围内膜的网状结构(图 2)。肽聚糖聚合物在革兰氏阳性细菌细胞表面的组装过程中,需要青霉素结合蛋白(penicillin-binding proteins,PBPs)对肽聚糖单体进行交联[12-14]。大部分PBPs为多结构域蛋白酶,根据其结构和功能分为A、B、C三类。在革兰氏阳性细菌中含量最多的为A类PBPs,该蛋白包括肽聚糖糖基转移酶(peptidoglycan transferase,PGT)和转肽酶(transpeptidase,TP)两个结构域。其中,PGTs结构域利用连接肽聚糖单体的十一烷基焦磷酸脂——脂质Ⅱ (lipid Ⅱ)作为底物,在肽聚糖分子的还原端添加脂质Ⅱ来合成N-乙酰氨基葡萄糖和N-乙酰胞壁酸交替连接的线性糖链,并释放运输肽聚糖单体的十一烷基焦磷酸脂质载体;在TP结构域的作用下这些线性糖链进一步交联,在肽链和邻近单元的D-丙氨酸之间形成共价键(图 2)。B类PBPs仅包括一个转肽酶结构域,常与A类PBPs协同完成细胞壁合成后期肽聚糖单体的聚合。C类PBPs为单官能团蛋白,负责肽聚糖聚合物的维持、重构和回收。一般来说A类PBPs和B类PBPs对细菌的生存是必要的,而C类PBPs的缺失不会对细菌造成致命性伤害。
|
| 图 2 默诺霉素抑制细胞壁合成的机理 Figure 2 Mechanism of moenomycins inhibiting the biosynthesis of cell wall. MurA-MurF: synthetase proteins; DdlA: D, D-carboxypeptidase; MurG: translocase; C55-P: lipid carrier (undecaprenylphosphate); UDP: uracil diphosphate. |
默诺霉素的作用靶点为A类PBPs的肽聚糖糖基转移酶结构域和革兰氏阳性细菌中单功能的肽聚糖糖基转移酶(如金黄色葡萄球菌中)[15](图 2),阻碍细菌细胞壁合成过程中肽聚糖单体的转糖基作用,进而阻止线性肽聚糖链的形成和十一烷基焦磷酸脂质载体的释放。其原因是由于默诺霉素和N-乙酰氨基葡萄糖与N-乙酰胞壁酸组成的肽聚糖单体在结构上具有一定相似性,被认为是一种竞争性抑制剂,而且能与十一烷基焦磷酸脂的肽聚糖单体(脂质Ⅱ)竞争性结合肽聚糖糖基转移酶,从而阻断细胞壁后续合成过程中肽聚糖链的合成和交联[14-16]。但也有研究表明,由于默诺霉素与PGTs结合的解离常数和半抑制浓度(IC50)稳定在纳摩尔范围内,脂质Ⅱ根本无法与默诺霉素形成有效竞争[17],其详细的作用机制依然值得深入探讨。
此外,在作用于革兰氏阳性细菌细胞壁的所有抗生素中,默诺霉素家族化合物是目前已知化合物中唯一作用于肽聚糖糖基转移酶的抗生素[14]。常见的β-内酰胺类药物(青霉素)和糖肽类药物(万古霉素)作用靶点分别为转肽酶结构域和肽聚糖单体五肽末端的D-丙氨酰-D-丙氨酸[18](图 2)。
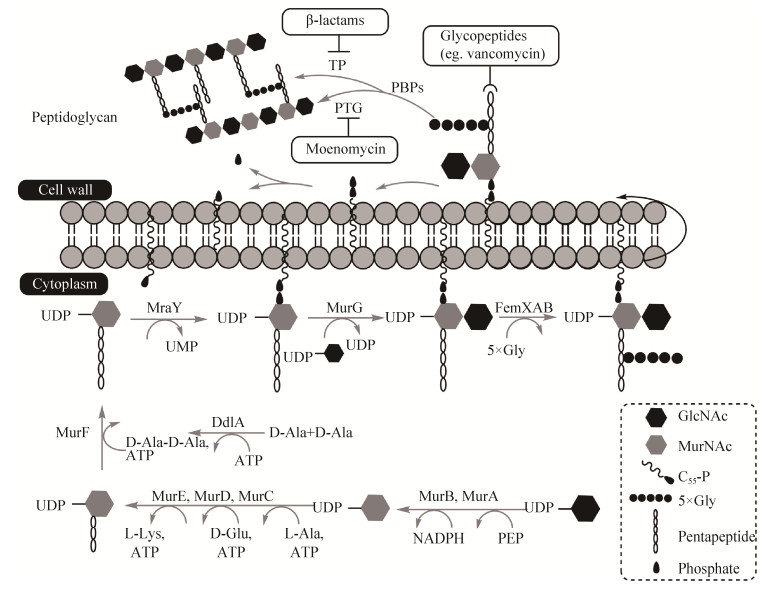
1.3 默诺霉素家族化合物的结构与活性的关系默诺霉素类抗生素B-C-E-F四个糖基环的不同修饰,对各自化合物的生物活性具有重要影响。一般认为磷酸甘油酸长脂肪链H、N-乙酰氨基半乳糖单位C环、N-乙酰氨基葡萄糖单位E环和烯脲酰胺F环结构的完整性是保持默诺霉素A抗菌活性的最小结构单位(图 3-A)。小鼠体内实验表明C环的缺失会导致默诺霉素在体内抗菌活性的丧失,但在体外仍对金黄色葡萄球菌等革兰氏阳性菌具有显著的活性[19]。
|
| 图 3 PGT-moenomycin A复合物晶体结构[22] (A) 和默诺霉素A的活性基团(B) Figure 3 The composite crystal structure of PGT-moenomycin A[22] (A) and the pharmacophore of moenomycin A (B). A: superimposition of two structures of S. aureus MtgA, PDB 3VMR (yellow) and PDB 3HZS(red). Moenomycin (green) bound to the donor site and a lipid Ⅱ analog (cyan) bound to the acceptor site are represented in stick. The mobile region between the two sites is highlighted by a circle. B: the moenomycin pharmacophore is represented in green, the other parts of molecule that do not contribute significantly to protein binding are shown in black. Red arrows mark the functional groups that form crucial hydrogen bonds to conserved active sites of amino acid residues of peptidoglycan transferase, and the key amino acid residues are represented by magenta ovals. |
在默诺霉素的作用靶点PGT家族的蛋白中存在5个保守的氨基酸基序EDxxFxxHxG、GxSTxTQQ、RKxxE、KxxIxxxYxN和RxxxL,具有相同的双侧螺旋状褶皱,活性位点位于双侧螺旋之间,5个PGTs特征氨基酸基序帮助形成活性中心[20]。金黄色葡萄球菌单功能肽聚糖糖基转移酶(monofunctional glycosyltransferase,Mtg)和默诺霉素A复合物的共结晶结构显示,默诺霉素A的戊多糖部分沿PGT活性口袋延伸(图 3-A)。默诺霉素A磷酸甘油酸部分的两个负电荷组和E-F双糖基环的氨基部分,在Mtg前3个高度保守的氨基酸基序EDxxFxxHxG、GxSTxTQQ和RKxxE中,与活性中心的100位谷氨酸、137位谷氨酰胺、132位丝氨酸和153位赖氨酸4个高度保守的关键氨基酸残基形成直接或水介导的氢键(图 3-B)[21-23]。2-氨基环戊烷-1, 3-二酮(C5N)发色团单位A环和B-D双糖基环与靶点上其他非活性关键位点氨基酸残基发生少量非特异性结合相互作用,这些相互作用有助于增强默诺霉素与靶点的结合力。从三磷酸甘油的位置和默诺霉素异戊二烯长脂肪链H前15个碳的电子密度可以推断,脂链从活性位点裂口的疏水沟向膜方向排列,使其沿着面向细胞膜的疏水性沟槽结合。异戊二烯长脂肪链H在默诺霉素发挥杀菌功能方面扮演着非常重要的角色,它的缺失会使默诺霉素同时丧失体内和体外活性[24]。
此外,默诺霉素与靶点结合的特异性在很大程度上也取决于E-F二糖和三磷酸甘油酸与活性位点侧链的相互作用;结合于五糖远端部分的其他相互作用以及25C长脂肪链H和Mtg之间的疏水相互作用。对默诺霉素家族化合物结构与生物活性关系的分析,有助于进一步了解革兰氏阳性耐药细菌对默诺霉素产生抗性的机制。
2 细菌的默诺霉素耐药机制的发掘默诺霉素类抗生素的作用靶点为PGTs,一些病原耐药细菌对默诺霉素产生抗性的原因是菌株在自然进化与选择过程中,靶蛋白基因(pbps)的突变所造成的。根据耐药菌株不同的抗性机制,将可耐受莫诺霉素的靶蛋百分为点突变后的aPBPs和bPBPs两种类型;(1) 肽聚糖糖基转移酶结构域的突变所产生的aPBPs降低了与默诺霉素的结合力;(2) bPBPs与同源SEDS蛋白家族(The shape, elongation,division and sporulation proteins)耦合形成复合蛋白,代替aPBPs行使肽聚糖链合成与交联功能。
2.1 细菌肽聚糖糖基转移酶结构域点突变类型aPBPs的默诺霉素耐药机制默诺霉素A靶点突变型耐药突变株最早发现于金黄色葡萄球菌中,研究者通过在菌株的培养基中,按照1−100 μg/mL的浓度范围梯度添加默诺霉素抗生素,得到了在默诺霉素浓度为10 μg/mL的压力条件下,依然可以生长的金黄色葡萄球菌[16]。这些菌株对默诺霉素A的最高耐受浓度从0.5 μg/mL提升到了12.5 μg/mL,对默诺霉素A的耐受性提高了25倍。对产生耐药的金黄色葡萄球菌精确的基因测序分析发现,所有的耐药突变均为肽聚糖糖基转移酶PBP2第196位的酪氨酸被天冬氨酸(PBP2Y196D)取代,或者234位的脯氨酸被谷氨酰胺(PBP2P234Q)取代,作用靶点PBP2的抗性突变频率为10–10到10–9。
金黄色葡萄球菌肽聚糖糖基转移酶的第196和234位氨基酸残基位于默诺霉素A结合位点空隙区,但不接触包括E-F双糖和三磷酸甘油酸组成的默诺霉素A药效基团的活性位点区[21]。第196和234位氨基酸残基更接近于默诺霉素A的A-B糖基环,它们发生非特异性结合的几率较低,然而这两个非活性关键位点的氨基酸残基发生突变能减弱与默诺霉素的结合,从而提高革兰氏阳性菌对默诺霉素的耐受性[23]。另外,这些耐药金黄色葡萄球菌的PBP2蛋白关键氨基酸的变化,也降低了肽聚糖聚合物在PGTs活性位点的结合力,导致菌株肽聚糖链的提前释放,所以金黄色葡萄球菌耐药突变体在细胞壁合成过程中具有肽聚糖链缩短的特征[16](图 4-B)。

|
| 图 4 aPBP点突变型默诺霉素耐药模型(Y196D) Figure 4 Model of aPBP point mutation-type moenomycin resistance (Y196D). A: aPBP-mediated model of peptidoglycan biosynthesis; B: aPBP point mutation-type proteins' biosynthetic characteristics of peptidoglycan monomers. |
2.2 细菌bPBPs和SEDS蛋白家族耦合类型的默诺霉素耐药机制
SEDS (the shape,elongation,division and sporulation proteins)蛋白家族在菌体生长、分裂和产孢过程中起着非常重要的作用,通过与同源的bPBP耦合形成的复合蛋白具有肽聚糖糖基转移酶的功能[25](图 5),这一研究成果为耐默诺霉素型革兰氏阳性细菌的抗性机制提供了新的理论基础。在研究默诺霉素耐受的革兰氏阳性细菌中发现,枯草芽孢杆菌中的RodA就是属于SEDS家族的成员,该蛋白和其同源的bPBP蛋白耦合,形成具有肽聚糖糖基转移酶和转肽酶双结构域的蛋白复合物,使得枯草芽孢杆菌在缺失所有aPBPs (ponA、pbpD、pbpF和pbpG)的情况下仍然具有细胞壁的合成能力[26],使菌株可以在默诺霉素添加的情况下依然可以存活。不同于aPBPs点突变型耐受机制,SEDS和其同源的bPBPs蛋白复合物的肽聚糖糖基转移酶结构域完全不同于aPBPs的糖基转移酶结构域,所以并不是默诺霉素家族化合物的作用靶点。但是除了枯草芽孢杆菌和少数几种嗜热菌外,并不是所有的细菌都可以在完全缺失aPBPs的情况下依靠SEDS-bPBPs复合蛋白系统完成肽聚糖单体聚合交联直至形成完整细胞壁。通过蛋白质相互作用研究和进化耦合分析,bPBPs和SEDS蛋白序列之间存在很强的共变异[27],在细胞中以双结构域复合蛋白的形式存在。SEDS蛋白家族必须要和其同源进化的bPBPs进行耦合形成蛋白复合物才能够执行肽聚糖糖基转移酶的功能[28-31]。bPBPs和SEDS的蛋白复合物包含肽聚糖糖链聚合和糖肽交联的活性位点,具有aPBPs的双重催化活性[32]。
|
| 图 5 bPBP-SEDS蛋白复合物介导的肽聚糖生物合成模型 Figure 5 The peptidoglycan biosynthetic model mediated by bPBP-SEDS protein complex. |
SEDS蛋白家族与bPBPs形成的蛋白耦合物和aPBPs对于肽聚糖的正确组装都起到至关重要的作用,虽然都含有肽聚糖糖基转移酶结构域,但由于是完全不同的蛋白家族,其结构上存在巨大差异,因此前者并不是糖基转移酶抑制剂默诺霉素的作用靶点。对SEDS蛋白家族与bPBPs活性和调控机制的进一步研究将为未来的抗生素开发铺平道路。
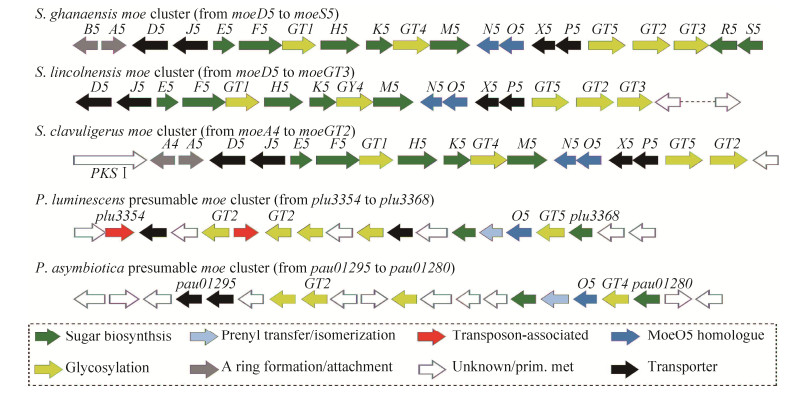
3 默诺霉素的生物合成研究进展默诺霉素A的生物合成基因发现于加纳链霉菌(S. ghanaensis)、斑堡链霉菌(S. bambergiensis)、喷泉链霉菌(S. geysiriensis)和埃德尔链霉菌(S. ederensis) 4种链霉菌的染色体上,并以簇状排列(表 1)。同时在林可链霉菌(S. lincolnensis)、棒状链霉菌(S. clavuligerus)和发光杆菌(Photorhabdus luminescens)中存在部分默诺霉素生物合成基因[9]。在林可链霉菌中包含默诺霉素生物合成基因簇cluster 1,而缺失了moeA4、moeB4和moeC4 3个A环合成基因以及moeR5和moeS5 2个糖基供体修饰基因;同时在默诺霉素产生菌加纳链霉菌中,默诺霉素的基因也排布于基因组的不同位置,其中moeD5到moeS5成簇状排列成cluster 1 (图 6),而moeA4、moeB4和moeC4单独成簇。
| Genes | aa | Proposed functions | Similar proteins (acc. number) | Identical (aa/%) |
| GT3 | 414 | Glycosyltransferase | WP_079165202.1 | 65 |
| GT2 | 286 | Glycosyltransferase | WP_067441233.1 | 84 |
| GT5 | 312 | Glycosyltransferase | ANS68555.1 | 67 |
| P5 | 235 | ABC transporter | WP_067441237.1 | 76 |
| X5 | 262 | ABC transporter | WP_067441240.1 | 80 |
| O5 | 286 | Prenyltransferase | WP_107406718.1 | 69 |
| N5 | 260 | Prenyltransferase | WP_107406888.1 | 70 |
| M5 | 530 | Carbamoyltransferase | WP_067441252.1 | 83 |
| GT4 | 427 | Glycosyltransferase | ANS68562.1 | 70 |
| K5 | 407 | Radical SAM domain-containing protein | WP_067441261.1 | 90 |
| H5 | 490 | Asparagine synthetase | WP_067441262.1 | 73 |
| GT1 | 402 | Glycosyltransferase | WP_067441264.1 | 76 |
| F5 | 645 | Asparagine synthetase | WP_067441267.1 | 74 |
| E5 | 340 | UDP-glucose 4-epimerase | WP_079164866.1 | 69 |
| J5 | 564 | ABC transporter | WP_067441273.1 | 71 |
| D5 | 591 | ABC transporter | WP_067445980.1 | 72 |
| S5 | 282 | Hexose-4-ketoreductase | WP_055690758.1 | 91 |
| R5 | 374 | Hexose-4, 6-dehydratase | WP_079060715.1 | 87 |
| A4 | 516 | Acyl CoA ligase | WP_131121788.1 | 88 |
| B4 | 521 | Amide synthetase | WP_079256894.1 | 83 |
| C4 | 412 | Aminolevulinate synthase | CCF23198.1 | 89 |
|
| 图 6 默诺霉素生物合成基因簇 Figure 6 Moenomycin biosynthesis gene clusters. |
默诺霉素的生物合成过程可以分成起始阶段、中期阶段和末期阶段3个部分(图 7)。在默诺霉素生物合成的起始阶段和中期阶段基因簇中的2个异戊二烯转移酶基因moeO5和moeN5参与了默诺霉素结构中通过顺式烯丙基乙醚连接到磷酸甘油酸酯上的不规则类异戊二烯链的合成[33]。MoeO5负责15C磷酸甘油酸酯的形成,MoeN5催化了10C的异戊二烯转移(图 7)。25C链长的默诺霉素长脂肪链的形成是通过10C异戊二烯转移到一个15C的中间体上形成的[34-35]。实验结果表明默诺霉素长脂肪链是在两个不同的阶段组装的,但是这种奇特的组装顺序的原因还并不清楚。

|
| 图 7 推导的默诺霉素生物合成途径 Figure 7 Proposed pathway for moenomycin biosynthesis. |
通过对生物合成基因簇中5个糖基转移酶基因的研究,验证了默诺霉素A 5个糖基的加载顺序。在默诺霉素合成的起始阶段,MoeGT1负责将第一个糖基加载到磷酸甘油酸酯,而后MoeGT4负责加载第2个E环的糖基,之后生物合成途径进入中期阶段后就分成了2个可能的支路,一条是MoeGT5加载C环的糖基,然后MoeGT3加载D环的糖基,最后由MoeGT2加载B环的糖基;另一条是MoeGT3加载D环的糖基后,MoeGT5加载C环的糖基,最后由MoeGT2加载B环的糖基(图 7)。MoeGT5和MoeGT3两个糖基转移酶对底物的识别可能具有宽泛性,在默诺霉素生物合成的末期阶段MoeGT5的识别底物既可以是chinovosamine (Ch)也可以是N-乙酰葡糖胺(GlcNAc),取决于MoeR5和MoeS5的催化效率[15]。
此外,在默诺霉素生物合成的过程中moeE5编码的蛋白是核苷酸糖差向异构酶[36],在起始阶段负责将UDP-葡萄糖醛酸转变为UDP-半乳糖醛酸,为默诺霉素生物合成提供第一个糖基。moeR5和moeS5编码了4, 6脱水酶/酮还原酶组合,在合成末期阶段负责将UDP-GlcNAc转化为UDP-chinovosamine。
默诺霉素生物合成基因簇中含有大量糖修饰酶基因。其中,MoeA4、MoeB4和MoeC4负责末期阶段A环的合成。moeH5和moeF5都可以编码氨基转移酶,负责默诺霉素家族末端修饰[37]。MoeK5是属于radical SAM依赖的甲基转移酶,对三糖的中间产物进行甲基化修饰[15]。到目前为止,虽然有很多研究人员作了默诺霉素相关基因功能的验证,可是还有很多机制尚未研究清楚,比如糖基加载的具体顺序等,很值得后续的研究人员作更深一步的探索。默诺霉素生物合成机制的完善将有助于通过代谢工程产生新的类似物。
4 默诺霉素产生菌对该类产物的外排在放线菌中,几乎所有已知的抗生素基因簇都含有质子或ATP依赖的转运蛋白的基因,这些转运蛋白负责相应的次级代谢产物的输出以行使防御或其他功能[38]。许多耐药菌通过表达大量的外排泵,将包括抗生素在内的多种分子从细菌细胞中运输出来,从而达到抗生素耐受的目的[39-40]。虽然前人对默诺霉素生物合成机制作了很多研究工作,但是关于默诺霉素外排机制的研究却相对较少。在默诺霉素的生物合成基因簇中,moeX5与moeP5和moeD5与moeJ5编码2对ATP依赖的转运蛋白(ATP-dependent transporters)。根据现有分类标准,MoeD5和MoeJ5是典型的Ⅲ型ABC转运蛋白,它们的跨膜域(TMD)和核苷酸结合域(NBD)融合为一个多肽;MoeX5和MoeP5是Ⅰ型ABC转运蛋白,这两个蛋白是Ⅰ型ABC转运蛋白的两种组分[41-42]。在默诺霉素的生物合成阶段,这两对蛋白的表达也处于活跃期,以促进默诺霉素排出菌体外[43]。
Walker等在默诺霉素产生菌加纳链霉菌中,通过基因敲除得到了3个加纳链霉菌突变体:dDJ5 (△moeD5和moeJ5)、dXP5 (△moeX5和moeP5)和dDJXP(缺失所有默诺霉素基因簇中转运蛋白基因),这些突变体在耐受默诺霉素、生长速度和产孢方面与亲本菌株无明显差异。但与野生型加纳链霉菌相比,所有突变体默诺霉素外分泌能力和产量下降,dXP5突变株尤其明显,其合成默诺霉素的平均水平降低了2倍,dDJ5菌株则降低了1.5倍;而在dDJXP突变菌株中也依然可以检测到默诺霉素的外分泌,这表明在加纳链霉菌中还存在默诺霉素基因簇以外的转运蛋白来执行外排功能[43]。
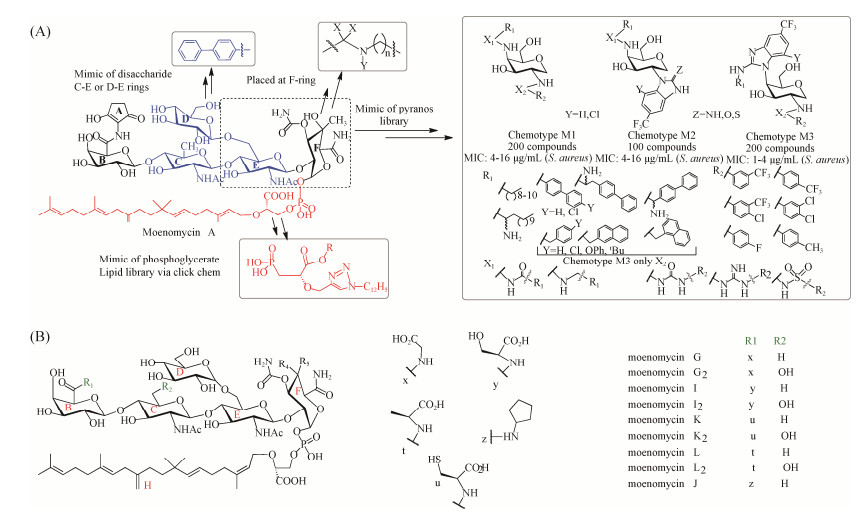
5 默诺霉素的新结构创制随着微生物药物合成研究的不断深入,利用合成生物学智能创制抗生素的研究思路和方法,以默诺霉素基础骨架为蓝本设计新的磷酸糖脂类抗生素,创制药代动力学特征更加优良的新结构或新活性默诺霉素衍生物,成为了新抗生素创制研究的生长点。在默诺霉素的结构中E-F双糖、磷酸甘油酸盐和长脂肪链脂尾是默诺霉素保持肽聚糖糖基转移酶抑制作用和抗细菌活性的关键官能团。基于E-F双糖-磷酸甘油酸脂结构设计了由联苯、胺连接剂和2-烷氧基-3-磷酸基丙酸酯组成的一系列截短模拟物[44-45](图 8-A),在此化合物库中筛选到了两种联苯和2-烷氧基-3-磷酸基丙酸酯连接的化合物对鲍曼不动杆菌(Acinetobacter baumannii)的糖基转移酶表现出适度的抑制活性(IC50≈150 mmol/L),并且对耐甲氧西林金黄色葡萄球菌具有良好的抗菌活性(MIC=6.3 mmol/L)。另外,以默诺霉素E-F双糖基环为模板,合成了基于新型吡喃糖的化合物文库,得到了与默诺霉素作用靶点相同但具有改善的药物代谢动力学性质的新化合物,这些新化合物都对金黄色葡萄球菌表现出不同程度的抑菌活性(图 8-A)[19]。
|
| 图 8 默诺霉素类似物结构的多样性 Figure 8 The structural diversity of moenomycin analogues. |
同时,默诺霉素B环周围的结构变化也是默诺霉素家族化合物生物合成多样性的重要来源,B环周围的化学结构涉及到默诺霉素生物合成过程中的末端修饰。MoeH5 (谷氨酰胺转移酶超家族的同源物,负责默诺霉素糖基B环的末端修饰)的底物识别具有宽泛性[37-46],在氨基酸喂养和moeH5过表达的条件下,MoeH5在体内可以识别结构相似的甘氨酸、丙氨酸、丝氨酸和半胱氨酸(A环的一部分),是一个通过酶法合成默诺霉素新结构的有效工具,对默诺霉素家族化合物末端结构变化提供了多样性(图 8-B)。B糖基环R1基团的侧链修饰对默诺霉素发挥杀菌活性非常重要,在高度耐甲氧西林金黄色葡萄球菌的生物活性测试实验中,默诺霉素A的MIC值达到了0.08 μg/mL,是默诺霉素家族中生物活性最好的化合物,默诺霉素G、默诺霉素I、默诺霉素K、默诺霉素L和默诺霉素J在氨基酸喂养实验的发酵产物中含量低,提纯困难,无法测定其准确的MIC值[37]。由于MoeH5供体底物的特异性,需要对其进行更广泛的研究和蛋白质工程,以充分利用MoeH5的潜力,从生物合成的角度开发出更具潜力的默诺霉素新结构。
6 讨论和展望默诺霉素类抗生素的发掘、全化学合成、生物合成通路的解析及其与作用靶点PGTs的构效关系的研究,为人们对默诺霉素的全面认识和应用开发奠定了基础。由于默诺霉素具有巨大的药用潜力,近几年来基于默诺霉素A的新结构衍生物的创制已成为新的研究关注点。但是,由于默诺霉素A的25C长脂肪链在哺乳动物体内半衰期极长,不利于全身系统性给药[24]。因此,基于默诺霉素和PGTs的药构关系分析,利用E-F双糖基环和磷酸脂链与PGTs的结合常数分析,同时通过A、B、C、D、E和F糖基环的修饰来进一步增强生物活性。定向创制默诺霉素高活性结构类似物,是改善默诺霉素药代动力学以实现其临床应用的重要手段。目前基于默诺霉素结构定向创制的一系列新化合物在体外具有良好的抗菌活性(图 8-A),但不利的理化性质限制了新化合物在体内的应用。然而,这组活性化合物提供了有价值的构效关系信息,基于较小的单糖骨架设计化合物文库,这种策略可以降低化合物的疏水性和化学复杂性,是未来默诺霉素成药性研究的重点方向。
尽管前人针对默诺霉素类抗生素的生物活性机制、生物合成研究、外排机制以及默诺霉素新结构创制等方面作了很多的研究工作,但是其生物合成机制依然没有完全得到解析,药物开发依然较为缓慢,其中一个关键问题就是默诺霉素在其野生型产生菌中的产量较低。为了提高默诺霉素的产量,很多研究人员尝试用白色链霉菌、天蓝链霉菌等模式菌株对默诺霉素生物合成基因簇进行异源表达[47],但收效甚微。根据默诺霉素的高活性特征进行推测,可能是由于宿主链霉菌对默诺霉素耐受性较差,从而无法将默诺霉素的产量提高到较高水平。因此,可以通过构建耐受性更强的底盘宿主链霉菌对基因簇进行异源表达,来提高默诺霉素生物合成产量,进而为创制多样性的默诺霉素类似物奠定研究基础。
此外,默诺霉素类抗生素除了具有重要的临床应用价值以外,还可以调节动物肠道菌群的代谢活性,使肠道壁变薄,有利于营养物质的吸收,现已经被大量运用于动物生长促进剂,在饲料中添加默诺霉素也可以诱导动物机体的抗炎作用,防止壁细胞丢失,在小鼠的动物实验中斑堡霉素对猪幽门螺杆菌的最低抑菌浓度为8 μg/mL[48-49]。因此,针对该类抗生素的全面深入研究,提高活性组分的产量,以及通过生物或者化学方法创制结构更为多样的结构类似物,依然具有重要的理论与应用价值。
| [1] | Horbal L, Ostash B, Luzhetskyy A, Walker S, Kalinowski J, Fedorenko V. A gene cluster for the biosynthesis of moenomycin family antibiotics in the genome of teicoplanin producer Actinoplanes teichomyceticus. Applied Microbiology and Biotechnology, 2016, 100(17): 7629-7638. DOI:10.1007/s00253-016-7685-3 |
| [2] | Zhao QQ, Luo YC, Zhang X, Kang QJ, Zhang D, Zhang LL, Bai LQ, Deng ZX. A severe leakage of intermediates to shunt products in acarbose biosynthesis. Nature Communication, 2020, 11(1): 1468. DOI:10.1038/s41467-020-15234-8 |
| [3] | Juhas M, Widlake E, Teo J, Huseby DL, Tyrrell JM, Polikanov YS, Ercan O, Petersson A, Cao S, Aboklaish AF, Rominski A, Crich D, Böttger EC, Walsh TR, Hughes D, Hobbie SN. In vitro activity of apramycin against multidrug-, carbapenem- and aminoglycoside-resistant Enterobacteriaceae and Acinetobacter baumannii. Journal of Antimicrobial Chemotherapy, 2019, 74(4): 944-952. DOI:10.1093/jac/dky546 |
| [4] | Lv MN, Ji XJ, Zhao JF, Li YZ, Zhang C, Su L, Ding W, Deng ZX, Yu Y, Zhang Q. Characterization of a C3 deoxygenation pathway reveals a key branch point in aminoglycoside biosynthesis. Journal of the American Chemical Society, 2016, 138(20): 6427-6435. DOI:10.1021/jacs.6b02221 |
| [5] | Qu S, Kang QJ, Wu H, Wang L, Bai LQ. Positive and negative regulation of GlnR in validamycin A biosynthesis by binding to different loci in promoter region. Applied Microbiology and Biotechnology, 2015, 99(11): 4771-4783. DOI:10.1007/s00253-015-6437-0 |
| [6] | Wu Q, Zhang LD, Xia H, Yu CJ, Dou K, Li YQ, Chen J. Omics for understanding synergistic action of validamycin A and Trichoderma asperellum GDFS1009 against maize sheath blight pathogen. Science Reports, 2017, 7(1): 40140. DOI:10.1038/srep40140 |
| [7] | Kahne D, Leimkuhler C, Lu W, Walsh C. Glycopeptide and lipoglycopeptide antibiotics. Chemical Reviews, 2005, 105(2): 425-448. DOI:10.1021/cr030103a |
| [8] | Brown ED, Wright GD. Antibacterial drug discovery in the resistance era. Nature, 2016, 529(7586): 336-343. DOI:10.1038/nature17042 |
| [9] | Wallhausser KH, Nesemann G, Prave P, Steigler A. Moenomycin, a new antibiotic. I. Fermentation and isolation. Antimicrobial Agents and Chemotherapy, 1965, 5(1): 734-736. |
| [10] | Welzel P. Syntheses around the transglycosylation step in peptidoglycan biosynthesis. Chemical Reviews, 2005, 105(12): 4610-4660. DOI:10.1021/cr040634e |
| [11] | Uchida R, Iwatsuki M, Kim YP, Omura S, Tomoda H. Nosokomycins, new antibiotics discovered in an in vivo-mimic infection model using silkworm larvae. Ⅱ: structure elucidation. Journal of Antibiotics, 2010, 63(4): 157-163. DOI:10.1038/ja.2010.10 |
| [12] | Packiam M, Weinrick B, Jacobs WR Jr, Maurelli AT. structural characterization of muropeptides from Chlamydia trachomatis peptidoglycan by mass spectrometry resolves "chlamydial anomaly". Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(37): 11660-11665. DOI:10.1073/pnas.1514026112 |
| [13] | Pazos M, Peters K. Peptidoglycan. Sub-Cellular Biochemistry, 2019(92): 127-168. |
| [14] | Schneider T, Sahl HG. An oldie but a goodie-cell wall biosynthesis as antibiotic target pathway. International Journal of Medical Microbiology, 2010, 300(2/3): 161-169. |
| [15] | Ostash B, Doud E, Lin C, Ostash I, Perlstein D, Fuse S, Wolpert M, Kahne D, Walker S. Complete characterization of the seventeen step moenomycin biosynthetic pathway. Biochemistry, 2009, 48(37): 8830-8841. DOI:10.1021/bi901018q |
| [16] | Rebets Y, Lupoli T, Qiao Y, Schirner K, Villet R, Hooper D, Kahne D, Walker S. Moenomycin resistance mutations in Staphylococcus aureus reduce peptidoglycan chain length and cause aberrant cell division. ACS Chemical Biology, 2014, 9(2): 459-467. DOI:10.1021/cb4006744 |
| [17] | Chen L, Walker D, Sun BY, Hu Y, Walker S, Kahne D. Vancomycin analogues active against vanA-resistant strains inhibit bacterial transglycosylase without binding substrate. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(10): 5658-5663. DOI:10.1073/pnas.0931492100 |
| [18] | Zagursky RJ, Pichichero ME. Cross-reactivity in β-lactam allergy. The Journal of Allergy and Clinical Immunology: in Practice, 2018, 6(1): 72-81. DOI:10.1016/j.jaip.2017.08.027 |
| [19] | Zuegg J, Muldoon C, Adamson G, McKeveney D, Le Thanh G, Premraj R, Becker B, Cheng M, Elliott AG, Huang JX, Butler MS, Bajaj M, Seifert J, Singh L, Galley NF, Roper DI, Lloyd AJ, Dowson CG, Cheng TJ, Cheng WC, Demon D, Meyer E, Meutermans W, Cooper MA. Carbohydrate scaffolds as glycosyltransferase inhibitors with in vivo antibacterial activity. Nature Communications, 2015(6): 7719. |
| [20] | Heaslet H, Shaw B, Mistry A, Miller AA. Characterization of the active site of S. aureus monofunctional glycosyltransferase (Mtg) by site-directed mutation and structural analysis of the protein complexed with moenomycin. Journal of Structural Biology, 2009, 167(2): 129-135. DOI:10.1016/j.jsb.2009.04.010 |
| [21] | Gampe CM, Tsukamoto H, Doud EH, Walker S, Kahne D. Tuning the moenomycin pharmacophore to enable discovery of bacterial cell wall synthesis inhibitors. Journal of the American Chemical Society, 2013, 135(10): 3776-3779. DOI:10.1021/ja4000933 |
| [22] | Sauvage E, Terrak M. Glycosyltransferases and transpeptidases/penicillin-binding proteins: valuable targets for new antibacterials. Antibiotics, 2016, 5(1): 12. DOI:10.3390/antibiotics5010012 |
| [23] | Fuse S, Tsukamoto H, Yuan YQ, Wang TSA, Zhang Y, Bolla M, Walker S, Sliz P, Kahne D. Functional and structural analysis of a key region of the cell wall inhibitor moenomycin. ACS Chemical Biology, 2010, 5(7): 701-711. DOI:10.1021/cb100048q |
| [24] | Ostash B, Walker S. Moenomycin family antibiotics: chemical synthesis, biosynthesis, and biological activity. Natural Product Reports, 2010, 27(11): 1594-1617. DOI:10.1039/c001461n |
| [25] | Meeske AJ, Riley EP, Robins WP, Uehara T, Mekalanos JJ, Kahne D, Walker S, Kruse AC, Bernhardt TG, Rudner DZ. SEDS proteins are a widespread family of bacterial cell wall polymerases. Nature, 2016, 537(7622): 634-638. DOI:10.1038/nature19331 |
| [26] | Emami K, Guyet A, Kawai Y, Devi J, Wu LJ, Allenby N, Daniel RA, Errington J. RodA as the missing glycosyltransferase in Bacillus subtilis and antibiotic discovery for the peptidoglycan polymerase pathway. Nature Microbiology, 2017(2): 16253. |
| [27] | Sjodt M, Brock K, Dobihal G, Rohs PDA, Green AG, Hopf TA, Meeske AJ, Srisuknimit V, Kahne D, Walker S, Marks DS, Bernhardt TG, Rudner DZ, Kruse AC. Structure of the peptidoglycan polymerase RodA resolved by evolutionary coupling analysis. Nature, 2018, 556(7699): 118-121. DOI:10.1038/nature25985 |
| [28] | Sjodt M, Rohs PDA, Gilman MSA, Erlandson SC, Zheng SD, Green AG, Brock KP, Taguchi A, Kahne D, Walker S, Marks DS, Rudner DZ, Bernhardt TG, Kruse AC. Structural coordination of polymerization and crosslinking by a SEDS-bPBP peptidoglycan synthase complex. Nature Microbiology, 2020, 5(6): 813-820. |
| [29] | Reichmann NT, Tavares AC, Saraiva BM, Jousselin A, Reed P, Pereira AR, Monteiro JM, Sobral RG, VanNieuwenhze MS, Fernandes F, Pinho MG. SEDS-bPBP pairs direct lateral and septal peptidoglycan synthesis in Staphylococcus aureus. Nature Microbiology, 2019, 4(8): 1368-1377. |
| [30] | Taguchi A, Welsh MA, Marmont LS, Lee W, Sjodt M, Kruse AC, Kahne D, Bernhardt TG, Walker S. FtsW is a peptidoglycan polymerase that is functional only in complex with its cognate penicillin-binding protein. Nature Microbiology, 2019, 4(4): 587-594. DOI:10.1038/s41564-018-0345-x |
| [31] | Cho H, Wivagg CN, Kapoor M, Barry Z, Rohs PDA, Suh H, Marto JA, Garner EC, Bernhardt TG. Bacterial cell wall biogenesis is mediated by SEDS and PBP polymerase families functioning semi-autonomously. Nature Microbiology, 2016, 1(10): 16172. DOI:10.1038/nmicrobiol.2016.172 |
| [32] | Rohs PDA, Buss J, Sim SI, Squyres GR, Srisuknimit V, Smith M, Cho H, Sjodt M, Kruse AC, Garner EC, Walker S, Kahne DE, Bernhardt TG. A central role for PBP2 in the activation of peptidoglycan polymerization by the bacterial cell elongation machinery. PLoS Genetics, 2018, 14(10): e1007726. |
| [33] | Zhang LL, Chen CC, Ko TP, Huang JW, Zheng YY, Liu WD, Wang I, Malwal SR, Feng XX, Wang K, Huang CH, Hsu STD, Wang AHJ, Oldfield E, Guo RT. Moenomycin biosynthesis: structure and mechanism of action of the prenyltransferase MoeN5. Angewandte Chemie International Edition, 2016, 55(15): 4716-4720. |
| [34] | Neundorf I, Köhler C, Hennig L, Findeisen M, Arigoni D, Welzel P. Evidence for the combined participation of a C10 and a C15 precursor in the biosynthesis of moenocinol, the lipid part of the moenomycin antibiotics. ChemBioChem, 2003, 4(11): 1201-1205. |
| [35] | Zhang LL, Ko TP, Malwal SR, Liu WD, Zhou SY, Yu XJ, Oldfield E, Guo RT, Chen CC. Complex structures of MoeN5 with substrate analogues suggest sequential catalytic mechanism. Biochemical and Biophysical Research Communications, 2019, 511(4): 800-805. |
| [36] | Sun H, Ko TP, Liu WT, Liu WD, Zheng YY, Chen CC, Guo RT. Structure of an antibiotic-synthesizing UDP-glucuronate 4-epimerase MoeE5 in complex with substrate. Biochemical and Biophysical Research Communications, 2020, 521(1): 31-36. |
| [37] | Ostash B, Campbell J, Luzhetskyy A, Walker S. MoeH5:a natural glycorandomizer from the moenomycin biosynthetic pathway. Molecular Microbiology, 2013, 90(6): 1324-1338. |
| [38] | Yang ZJ, Wei X, He JQ, Sun CL, Ju JH, Ma JY. Characterization of the noncanonical regulatory and transporter genes in atratumycin biosynthesis and production in a heterologous host. Marine Drugs, 2019, 17(10): 560. |
| [39] | Blair JMA, Richmond GE, Piddock LJV. Multidrug efflux pumps in Gram-negative bacteria and their role in antibiotic resistance. Future Microbiology, 2014, 9(10): 1165-1177. |
| [40] | Kabra R, Chauhan N, Kumar A, Ingale P, Singh S. Efflux pumps and antimicrobial resistance: paradoxical components in systems genomics. Progress in Biophysics and Molecular Biology, 2019(141): 15-24. |
| [41] | Orelle C, Mathieu K, Jault JM. Multidrug ABC transporters in bacteria. Research in Microbiology, 2019, 170(8): 381-391. |
| [42] | Ford RC, Beis K. Learning the ABCs one at a time: structure and mechanism of ABC transporters. Biochemical Society Transactions, 2019, 47(1): 23-36. |
| [43] | Ostash B, Doud E, Walker S. ABC transporter genes from Streptomyces ghanaensis moenomycin biosynthetic gene cluster: roles in antibiotic production and export. Archives Microbiology, 2012, 194(11): 915-922. |
| [44] | Yu JY, Cheng HJ, Wu HR, Wu WS, Lu JW, Cheng TJ, Wu YT, Fang JM. Structure-based design of bacterial transglycosylase inhibitors incorporating biphenyl, amine linker and 2-alkoxy-3-phosphorylpropanoate moieties. European Journal of Medicinal Chemistry, 2018(150): 729-741. |
| [45] | Mesleh MF, Rajaratnam P, Conrad M, Chandrasekaran V, Liu CM, Pandya BA, Hwang YS, Rye PT, Muldoon C, Becker B, Zuegg J, Meutermans W, Moy TI. Targeting bacterial cell wall peptidoglycan synthesis by inhibition of glycosyltransferase activity. Chemical Biology & Drug Design, 2016, 87(2): 190-199. |
| [46] | Kuzhyk Y, Lopatniuk M, Luzhetskyy A, Fedorenko V, Ostash B. Genome engineering approaches to improve nosokomycin a production by Streptomyces ghanaensis B38.3. Indian Journal of Microbiology, 2019, 59(1): 109-111. DOI:10.1007/s12088-018-0761-x |
| [47] | Lopatniuk M, Ostash B, Luzhetskyy A, Walker S, Fedorenko V. Generation and study of the strains of streptomycetes-heterologous hosts for production of moenomycin. Russian Journal of Genetics, 2014, 50(4): 360-365. |
| [48] | Nair S, Farzan A, O'Sullivan TL, Friendship RM. A clinical trial investigating the impact of in-feed flavophospholipol on Salmonella shedding and antimicrobial resistance in pigs. The CanadianVeterinary Journal, 2018, 59(1): 59-65. |
| [49] | De Witte C, Taminiau B, Flahou B, Hautekiet V, Daube G, Ducatelle R, Haesebrouck F. In-feed bambermycin medication induces anti-inflammatory effects and prevents parietal cell loss without influencing Helicobacter suis colonization in the stomach of mice. Veterinary Research, 2018, 49(1): 35. |
 2021, Vol. 61
2021, Vol. 61