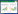中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 韦俊宏, 张集慧, 江玲娟, 谭华荣, 牛国清
- Junhong Wei, Jihui Zhang, Lingjuan Jiang, Huarong Tan, Guoqing Niu
- 谷氏菌素生物合成基因gouC和gouD的功能研究
- Functional characterization of gouC and gouD in gougerotin biosynthesis
- 微生物学报, 2016, 56(3): 406-417
- Acta Microbiologica Sinica, 2016, 56(3): 406-417
-
文章历史
- 收稿日期:2016-01-08
- 修回日期:2016-01-29
- 网络出版日期:2016-02-18
2. 中国科学院微生物研究所, 微生物资源前期开发国家重点实验室, 北京 100101
2. State Key Laboratory of Microbial Resources, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
谷氏菌素属于核苷肽类抗生素,具有抗肿瘤、抗病毒、抗支原体、驱虫与杀螨等生物活性[1, 2, 3, 4],是一种具有开发价值的农用与医用抗生素,其产生菌主要包括谷氏链霉菌(Streptomyces gougerotii) 和禾粟链霉菌(Streptomyces graminearus)[5, 6]。谷氏菌素是蛋白质合成的抑制剂[7],通过抑制核糖体中肽基转移酶的功能来阻碍多肽链延伸[8]。谷氏菌素由核苷和肽基两部分组成,核苷部分为1-(4-脱氧-β-D-吡喃葡萄糖醛酰胺)胞嘧啶,肽基部分为肌氨酸-D-丝氨酸二肽,核苷部分通过与肽基部分D-丝氨酸的羧基部分缩合生成酰胺键相连(图 1-A)。谷氏菌素的生物合成基因簇已经从禾粟链霉菌克隆得到,并通过异源表达和基因突变确定了其边界[6]。该基因簇由15个基因组成,包括1个调控基因、1个转运蛋白编码基因和13个结构基因(图 1-B)。谷氏菌素生物合成基因簇中唯一的调控基因gouR通过调控结构基因转录单元gouL-gouB和转运蛋白编码基因gouM的转录来控制谷氏菌素的生物合成[9]。通过构建基因阻断突变株,分离相应突变株中积累的中间产物及其结构解析,初步揭示了谷氏菌素的生物合成途径[6]。
|
| 图 1. 谷氏菌素的化学结构及其生物合成基因簇 Figure 1. Chemical structure of gougerotin (A) and organization of the gene cluster for gougerotin biosynthesis (B). |
谷氏菌素生物合成的前体主要包括UDP-葡萄糖、胞嘧啶、甘氨酸和三磷酸甘油酸。基因簇中gouF、gouA、gouH和gouB与谷氏菌素核苷部分的生物合成相关,其中gouF、gouA和gouH共同作用催化核苷部分骨架4-氨基-CGA的形成,而gouB则负责糖苷羧基的酰胺化。谷氏菌素的肽基部分由D-丝氨酸与肌氨酸组成,推测gouG、gouI和gouL参与了D-丝氨酸的形成,而甲基转移酶GouN则负责催化甘氨酸N端甲基化形成肌氨酸。D-丝氨酸会先同核苷部分4位氨基缩合,随后丝氨酸的氨基部分再与肌氨酰辅酶A缩合得到终产物谷氏菌素。GouK含有一个乙酰辅酶A结合结构域,负责将丝氨酸或甘氨酸活化形成丝氨酰辅酶A或甘氨酰辅酶A,而N-乙酰基转移酶GouJ催化活化的氨基酸与核苷部分缩合。在谷氏菌素的生物合成途径中,gouC与gouD发挥的功能并不清楚。生物信息学分析表明GouC仅与数据库中少量蛋白具有低的同源性,且其同源蛋白功能未知,而GouD与糖基转移酶有一定的同源性[6]。研究gouC与gouD的功能有助于阐明谷氏菌素的生物合成机制,也有可能发现具有新催化活性的蛋白家族,还可以为谷氏菌素与其他核苷肽类抗生素进行组合生物合成奠定基础[10]。
由于谷氏菌素产生菌禾粟链霉菌遗传操作效率较低,本实验室已成功地将谷氏菌素生物合成基因簇在天蓝色链霉菌M1146中得到了异源表达。因此本研究选择在天蓝色链霉菌M1146中构建了gouC和gouD的缺失突变株及其相应遗传互补株。HPLC分析发现gouC和gouD缺失均导致谷氏菌素不能合成,而导入相应互补质粒能够回复谷氏菌素的产生,证明gouC和gouD是谷氏菌素生物合成的关键结构基因。通过对gouC和gouD缺失突变株中积累化合物C与化合物D的结构解析,发现它们与谷氏菌素的差异在肌氨酸残基部分,由此推断gouC和gouD与谷氏菌素肽基部分的肌氨酸合成相关,而生物活性检测发现肌氨酸残基对谷氏菌素的抗肿瘤活性至关重要。本研究为进一步阐明谷氏菌素生物合成机制以及与其他核苷肽类抗生素的组合生物合成奠定了基础。
1 材料和方法 1.1 材料1.1.1 菌株、质粒及引物:本文所用的菌株、质粒以及基因操作过程中的引物详见表 1-3。
| Strains | Genotype/Description | Reference/Source | |
| Streptomyces | |||
| S. coelicolor M1146 | Δact, Δred, Δcpk, Δcda | [11] | |
| M1146-GOUe | S. coelicolor M1146 containing pGOUe | This work | |
| M1146-DgouC | S. coelicolor M1146 containing pGOUe-ΔC | This work | |
| M1146-DgouD | S. coelicolor M1146 containing pGOUe-ΔD | This work | |
| M1146-gouCc | M1146-DgouC containing pIJ10500-gouC | This work | |
| M1146-gouDc | M1146-DgouD containing pIJ10500-gouD | This work | |
| Escherichia coli | |||
| DH5α | F–, φ80lacZΔM15, Δ(lacZYA-argF) U169, deoR, recA1, endA1, hsdR17 (rK–mK+), phoA, supE44, λ–, thi–1, gyrA96, relA1 | Gibco BRL | |
| ET12567/pUZ8002 | dam– dcm– hsdM–/pUZ8002 | [12] | |
| BW25113/pKD20 | K-12 derivative, ΔaraBAD, ΔrhaBAD/pKD20 | [13] | |
| DH5α/BT340 | DH5a derivative containing pCP20 | [13] | |
| Plasmids | Relevant characteristics | Reference/Source |
| pIJ10500 | int-BT1, attP-BT1, oriT, integrative vector | [14] |
| pSET152 | int-fC31, attp-fC31, oriT, integrative vector | [12] |
| D6-4H | pCC1FOS derivate containing the whole gougerotin biosynthesis gene cluster | [6] |
| pSV152TA | pCC1FOS derivates containing apramycin and thiostrepton resistance gene cassettes flanked by FRT sites | [6] |
| pSET152Erm::hA/MN | pSET152 derivate containing gouN with PhrdB, part of gouM and gouA with PhrdB | [15] |
| D6-4H-Cta | D6-4H derivate containing the whole gougerotin biosynthesis gene cluster, with gouC replaced by apramycin and thiostrepton resistance gene cassettes | This work |
| D6-4H-Dta | D6-4H derivate containing the whole gougerotin biosynthesis gene cluster, with gouD replaced by apramycin and thiostrepton resistance gene cassettes | This work |
| D6-4H-ΔC | D6-4H-Cta derivate with apramycin and thiostrepton resistance gene cassettes excised | This work |
| D6-4H-ΔD | D6-4H-Dta derivate with apramycin and thiostrepton resistance gene cassettes excised | This work |
| pGOUe | pSET152 derivate containing the whole gougerotin biosynthesis gene cluster with engineered promoters for gouA, gouM and gouN | [15] |
| pGOUe-ΔC | pGOUe derivate with gouC deleted | This work |
| pGOUe-ΔD | pGOUe derivate with gouD deleted | This work |
| pIJ10500-gouC | pIJ10500 derivate containing gouC with hrdB promoter | This work |
| pIJ10500-gouD | pIJ10500 derivate containing gouD with hrdB promoter | This work |
| Primers | Sequences (5′→3′) | Purpose |
| gouC-dF | AGGGCCGGGAGCGGCGCGCCGCGCCCCGT GCGACGGCTATTCCGGGGATCCGTCGACC | gouC disruption |
| gouC-dR | ACGTTTTCGGGGGTGGGCGGACGGGCGG ACCCCGCGGGTTGTAGGCTGGAGCTGCTTC | gouC disruption |
| gouD-dF | CGCCGGCCACCGAGCACCGCCCCGTCGAC GTCAGCGTCAATTCCGGGGATCCGTCGACC | gouD disruption |
| gouD-dR | GGGCGCGGTGCTCGAACCGCACGGCGGTG TCCACCCATGTGTAGGCTGGAGCTGCTTC | gouD disruption |
| hrdBp-F | AATTTCTAGACCGCCTTCCGCCGG | PCR of hrdB promoter |
| hrdBp-R | GAACAACCTCTCGGAACGTTG | PCR of hrdB promoter |
| gouC-cF | GTGCGACGGCTCGTGTACCTG | gouC complementation |
| gouC-cR | AATTAGGCCTTCAGGCGTACCGTCCGAGGTC | gouC complementation |
| gouD-cF | ATGACCGAGCACGCCCGGCC | gouD complementation |
| gouD-cR | ATTAGGCCTTCACCCGTGGTGGGCCGGCT | gouD complementation |
| gouA-B-F | GTTCAGCCTCTCCACGTGGC | PCR 757 bp of gouA and gouB |
| gouA-B-R | ACCTGGTGGTGGACGACCCT | PCR 757 bp of gouA and gouB |
| gouG-H-F | AGAAGGCAGAGGCGCTGGCG | PCR 748 bp of gouG and gouH |
| gouG-H-R | GGACAGCCCCATCGCGTACG | PCR 748 bp of gouG and gouH |
| gouM-N-F | GAGTCTCGCCGAGACCTGCC | PCR 859 bp of gouM and gouN |
| gouM-N-R | GCAGGGAGAGCAGGAGGAGC | PCR 859 bp of gouM and gouN |
1.1.2 培养基和培养条件:LB培养基用于大肠杆菌的培养,培养温度为30 °C或37 °C;MS培养基和YEME培养基用于链霉菌培养,GP为谷氏菌素的发酵培养基[6],培养温度为28 °C。
1.1.3 主要试剂:限制性内切酶购自NEB公司,高保真KOD-plus DNA聚合酶购自TOYOBO公司,T4 DNA连接酶购自Thermo公司,Diaion HP-20购自日本三菱化学公司,阳离子交换树脂Dowex®50wx2购自SIGMA-Aldrich公司,葡聚糖凝胶Sephadex LH-20购自GE Healthcare公司。
1.2 DNA基本操作与分析常规的分子生物学操作参见分子克隆指南[13],链霉菌基因组的提取参见链霉菌操作手册[12],PCR-targeting的操作按照文献方法进行[16]。
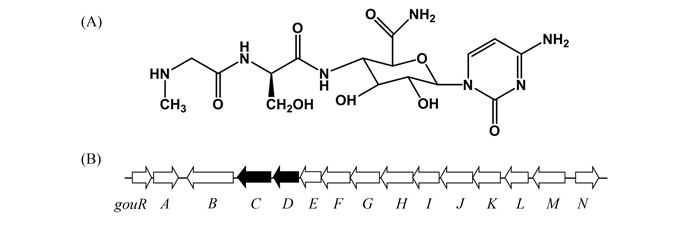
1.3 重组质粒的构建用于gouC基因敲除的重组质粒pGOUe-ΔC的构建策略如图所示(图 2):以质粒pSV152TA[6]为模板,用引物对gouC-dF/gouC-dR进行PCR扩增得到一个2435 bp的DNA片段,该片段包含了安普霉素与硫链丝菌素的抗性基因,而两侧则与gouC部分序列同源。通过电击转化将该片段导入含有D6-4H的大肠杆菌 BW25113/pKD20中,经过大肠杆菌内重组酶介导的同源重组后,gouC基因被安普霉素与硫链丝菌素的抗性基因所替换,得到了D6-4H-Cta。将D6-4H-Cta导入大肠杆菌DH5α/BT340中,通过FLP系统识别抗性基因两端的FRT位点将抗性基因环出得到D6-4H-ΔC。将pSET152Erm::hA/MN经EcoRV酶切,线性片段切胶回收后,通过电击转化导入包含D6-4H-ΔC的大肠杆菌BW25113/pKD20中,通过重组酶介导的同源重组得到重组质粒pGOUe-ΔC。pGOUe-ΔC包含替换了各结构单元启动子的谷氏菌素生物合成基因簇,但是gouC部分则发生了缺失突变。采用相同的方法,构建了重组质粒pGOUe-ΔD。
|
| 图 2. 重组质粒pGOUe-ΔC的构建 Figure 2. Schematic diagram of pGOUe-ΔC construction. PhrdB: hrdB promoter; TA: apramycin and thiostrepton resistance genes; pSET152Erm::hA/MN EcoR V: EcoR V linearized pSET152Erm::hA/MN. |
为了在天蓝色链霉菌M1146-GOUe-ΔC中互补gouC基因,以pGOUe为模板,使用引物对gouC-cF/gouC-cR扩增得到一个包含完整gouC基因编码区的1302 bp DNA片段(gouC-cds)。同时利用T4激酶将引物hrdBp-R的5′端磷酸化后,使用引物hrdBp-F/hrdBp-R扩增得到一个包含天蓝色链霉菌hrdB基因启动子区PhrdB 的525 bp DNA片段。以Stu I酶切gouC-cds,Xba I酶切PhrdB,将两个酶切后的DNA片段同时与Spe I和Stu I酶切过的pIJ10500 连接,得到gouC回补载体pIJ10500-gouC。采用相同的方法,构建了用于gouD基因互补的重组质粒pIJ10500-gouD。
1.4 天蓝色链霉菌的接合转移重组质粒转化ET12567/pUZ8002,通过接合转移[12]的方法导入天蓝色链霉菌M1146中,得到的转化子通过PCR验证导入的基因簇是否完整。
1.5 谷氏菌素的发酵与HPLC分析谷氏菌素的发酵与HPLC分析按照文献方法[6] 进行,抗肿瘤测试方法参见文献[17]。
1.6 化合物C与化合物D的分离纯化与结构鉴定发酵液通过大孔树脂Diaion HP-20,收集流出液;通过阳离子交换树脂Dowex®50wx2,用 0.2 mol/L氨水洗脱;将洗脱液通过葡聚糖凝胶Sephadex LH-20,分段收集并进行HPLC分析确证目标化合物所在区段;将前述粗提物通过Agilent 1100 高效液相色谱仪以及ZORBAX SB-C18柱 (4.6 mm×250 mm,5 μm)进一步纯化;纯化后的产物通过质谱与核磁共振进行结构分析。
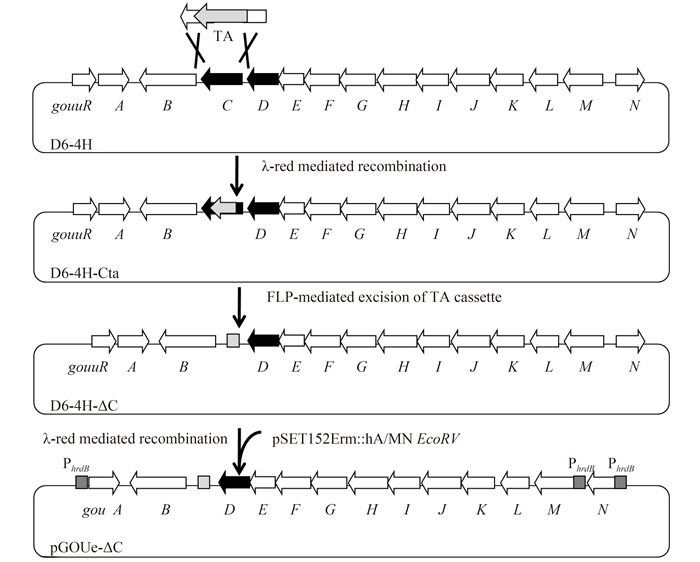
2 结果和分析 2.1 gouC、gouD基因阻断突变株和互补菌株的构建为了研究gouC和gouD在谷氏菌素生物合成中的功能,分别构建了重组质粒pGOUe-ΔC和pGOUe-ΔD (图 2),通过接合转移分别将这两个重组质粒导入天蓝色链霉菌M1146中,获得M1146-GOUe-ΔC和M1146-GOUe-ΔD。同时将含有完整谷氏菌素生物合成基因簇的pGOUe导入天蓝色链霉菌M1146中,获得异源表达菌株M1146-GOUe。 随机挑选3个转化子提取基因组DNA,选用3对引物(gouA-B-F/gouA-B-R,gouG-H-F/gouG-H-R,gouM-N-F/gouM-N-R)进行PCR验证,得到的扩增产物片段与预期大小一致,证明3个重组质粒成功地转入天蓝色链霉菌M1146中(图 3-A)。此外,构建了用于遗传互补的重组质粒pIJ10500-gouC和pIJ10500-gouD (具体步骤详见材料和方法部分)。将pIJ10500-gouC和pIJ10500-gouD分别导入相应突变株M1146-GOUe-ΔC和M1146-GOUe-ΔD中,筛选出具有潮霉素抗性的接合转化子,即得到互补菌株M1146-gouCc和M1146-gouDc。提取M1146-gouCc和M1146-gouDc的基因组DNA,分别用引物对gouC-cF/gouC-cR和gouD-cF/gouD-cR进行PCR验证。所有PCR扩增产物片段大小与预期一致,表明回补质粒已成功地整合到M1146-GOUe-ΔC和M1146-GOUe-ΔD中(图 3-B)。
|
| 图 3. 突变株(A)与互补株(B)的PCR验证 Figure 3. Confirmation of deletion mutants and complementary strains by PCR. A: Confirmation of M1146-GOUe-ΔC, M1146-GOUe-ΔD and M1146-GOUe by PCR. M: DNA ladder; lane 1: genomic DNAs from S. coelicolor M1146 as templates; lane 2–3: genomic DNAs from different M1146-GOUe as templates; lane 4–6: genomic DNAs from different M1146-GOUe-ΔD as templates; lane 7–9: genomic DNAs from different M1146-GOUe-ΔC as templates. B: Confirmation of M1146-gouCc (left) and M1146-gouDc (right) by PCR. M: DNA ladder; lane 1: genomic DNAs from S. coelicolor M1146 as templates; lane 2: genomic DNAs from M1146-GOUe as templates; lane 3–6: genomic DNAs from different M1146-gouCc or M1146-gouDc as templates. |
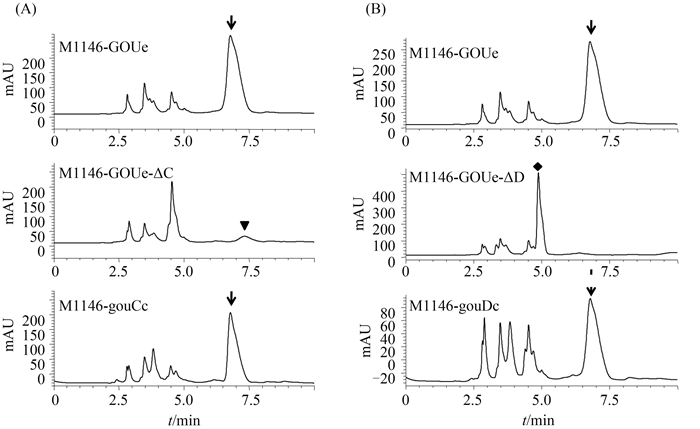
将得到的重组菌株(M1146-GOUe-ΔC、M1146-GOUe-ΔD、M1146-GOUe、M1146-gouCc和M1146-gouDc)分别接种于GP发酵培养基,发酵5天后收集发酵液,通过HPLC检测谷氏菌素的产生情况。结果表明:与M1146-GOUe不同,M1146-GOUe-ΔC和M1146-GOUe-ΔD均丧失了产生谷氏菌素的能力,而相应的遗传互补菌株M1146-gouCc和M1146-gouCc则能够回复谷氏菌素的产生(图 4)。进一步分析发现:M1146-GOUe-ΔC和M1146-GOUe-ΔD分别在保留时间7.4 min和4.5 min的位置有新的吸收峰出现,对应的化合物命名为化合物C和化合物D。以上结果证明gouC和gouD与谷氏菌素的生物合成有直接的关系(图 4)。
|
| 图 4. gouC (A)和gouD (B)突变株与互补株发酵液的HPLC分析 Figure 4. HPLC analysis of compound C (triangle, figure A), compound D (rhombus, figure B) and gougerotin (arrow, figure A and B). |
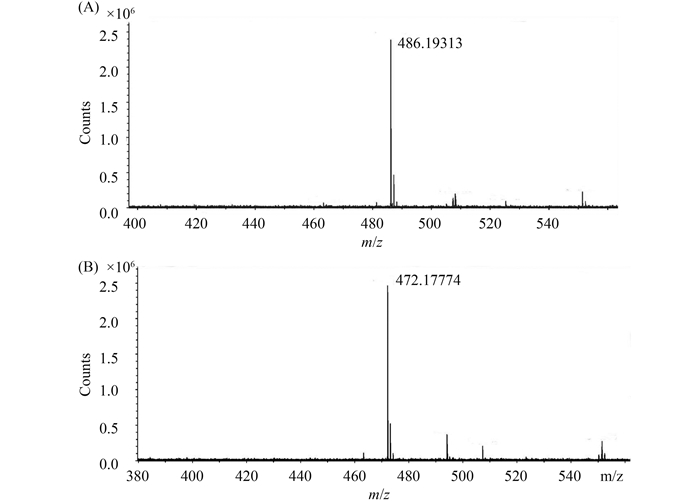
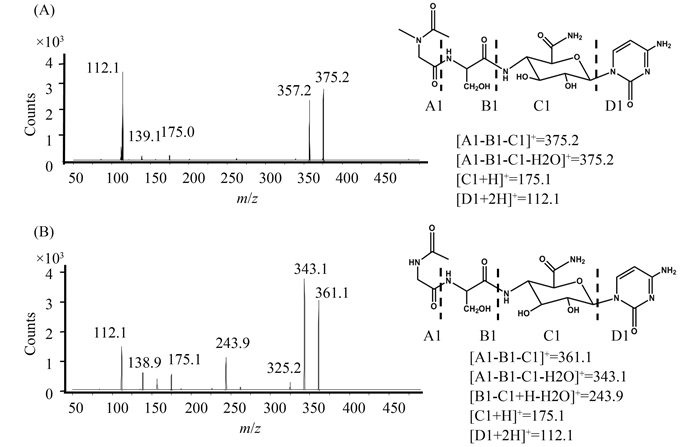
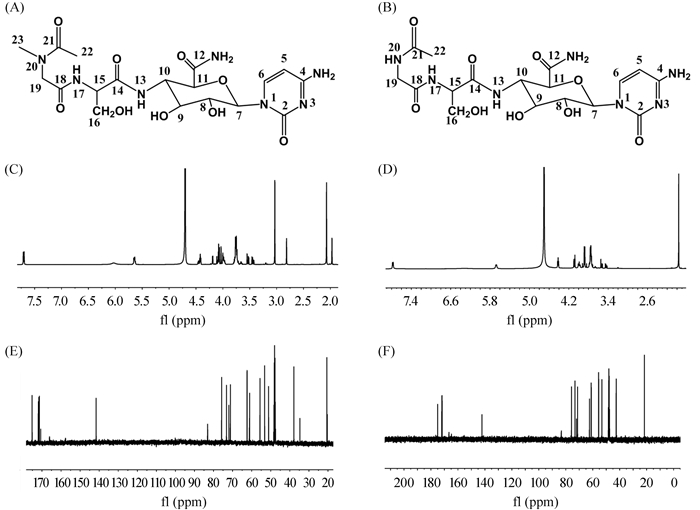
为了揭示gouC和gouD在谷氏菌素生物合成途径中的作用,需要对突变株中积累的中间产物进行结构鉴定。为此,对M1146-GOUe-ΔC和M1146-GOUe-ΔD进行了大量发酵培养,发酵液经大孔树脂Diaion HP-20、阳离子交换色谱、葡聚糖凝胶Sephadex LH-20和HPLC等一系列方法分离纯化后获得了化合物C和D的纯品,并将这些样品用于质谱和核磁共振分析。高分辨率质谱数据显示:化合物C的相对分子量[M+H]+为486.19312,推测其分子式为C18H27N7O9 (图 5-A);化合物D的相对分子量[M+H]+为472.17774,推测其分子式为C17H25N7O9 (图 5-B)。串联质谱分析表明化合物C与化合物D的核苷部分与谷氏菌素相同,而肽基部分则发生了明显的变化(图 6),分别比谷氏菌素的肽基部分多了42 和28个质量数。结合1H (图 7-C、D)、13C (图 7-E、F)、1H-13C HMBC、1H-13C HSQC等核磁共振波谱数据分析,最终化合物C被 鉴定为1-(4-N-乙酰肌氨酰胺-D-丝氨酰胺-4-脱氧-β-D-吡喃葡萄糖醛酰胺)胞嘧啶(图 7-A),化合物D被鉴定为1-(4-N-乙酰甘氨酰胺-D-丝氨酰胺-4-脱氧-β-D-吡喃葡萄糖醛酰胺)胞嘧啶(图 7-B),相关核磁共振波谱数据分析请见表 4。化合物C和化合物D与谷氏菌素的差别仅在肌氨酸残基,化合物C对应位置为N-乙酰肌氨酸残基,而化合物D对应位置则为N-乙酰甘氨酸残基。
|
| 图 5. 化合物C和D的高分辨质谱分析 Figure 5. High resolution mass spectrum of compound C (A) and compound D (B). |
|
| 图 6. 化合物C和D串联质谱分析 Figure 6. Tandem mass spectrum of compound C (A) and compound D (B). |
|
| 图 7. 化合物C和D的结构鉴定 Figure 7. Structural determination of compound C and compound D. Structure of compound C (figure A) and compound D (figure B); 1H-NMR spectrum of compound C (figure C) and compound D (figure D); 13C-NMR spectrum of compound C (figure E) and compound D (figure F). |
| Position | Compound C | Compound D | ||
| 1H(δ) | 13C(δ) | 1H(δ) | 13C(δ) | |
| 2 | 155.0 | |||
| 4 | 166.0 | 164.0 | ||
| 5 | 6.03 | |||
| 6 | 7.70 | 142.0 | 7.78 | 142.0 |
| 7 | 5.64 | 83.0 | 5.67 | 83.0 |
| 8 | 3.75 | 71.0 | 3.75 | 71.0 |
| 9 | 3.75 | 73.0 | 3.76 | 73.0 |
| 10 | 3.99 | 52.9 | 3.99 | 53.0 |
| 11 | 4.09 | 75.5 | 4.09 | 75.7 |
| 12 | 171.7 | 171.6 | ||
| 14 | 171.9 | 171.9 | ||
| 15 | 4.41 | 55.8 | 4.41 | 55.0 |
| 16 | 3.75 | 61.0 | 3.74 | 61.0 |
| 18 | 171.7 | 171.8 | ||
| 19 | 4.06 | 51.3 | 3.88 | 42.6 |
| 21 | 175.0 | 175.0 | ||
| 22 | 2.07 | 20.5 | 1.95 | 21.6 |
| 23 | 3.03 | 38.2 | ||
次级代谢产物基团上的变化一般会引起其生物活性发生改变[18, 19]。与谷氏菌素相比,化合物C在肌氨酸残基处发生了N-乙酰化,而化合物D则在N-乙酰化的基础上缺失了N-甲基,为了研究这些变化对其生物活性的影响,需要比较分析这些化合物的生物活性。鉴于谷氏菌素具有抗肿瘤活性[20],分别测定了化合物C、化合物D和谷氏菌素对人肺腺癌细胞A549生长的抑制效应。结果发现:谷氏菌素对人肺腺癌细胞A549增殖表现出良好的抑制作用,而化合物C和D均丧失了抑制人肺腺癌细胞A549增殖的能力(图 8)。这些结果表明谷氏菌素肽基部分的肌氨酸残基对其生物活性至关重要。
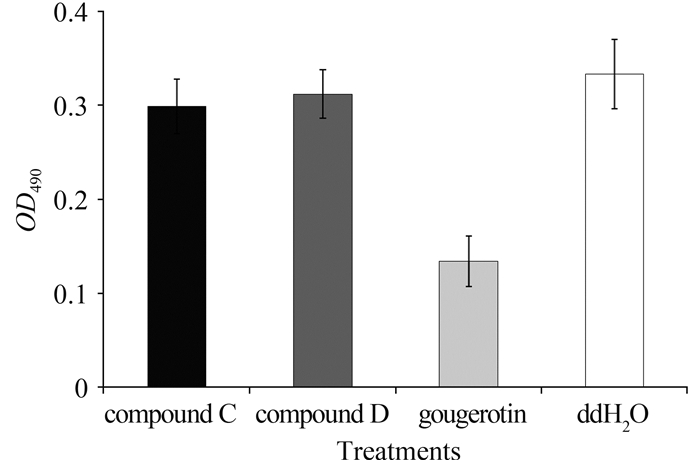
|
| 图 8. 化合物C、化合物D及谷氏菌素的抗肿瘤活性分析 Figure 8. Antitumor assay of compound C, compound D and gougerotin. Sterilized ddH2O was added as negative control. |
在次级代谢产物生物合成途径的研究中,对生物合成基因簇中各结构基因进行阻断与互补有助于表征其功能。某些次级代谢产物的产生菌缺乏有效的遗传操作系统,进行结构基因的阻断与回补难度很大,而将其生物合成基因簇导入遗传操作体系成熟的菌株中进行异源表达,则可以很好的解决这些问题[21]。本实验室前期工作已经在天蓝色链霉菌M1146中成功表达了谷氏菌素生物合成基因簇,并通过替换基因簇内各转录单元启动子的方法,将谷氏菌素的产量提高到原产生菌(禾粟链霉菌)的水平[22]。由于禾粟链霉菌的遗传操作难度较大,在该菌中尝试敲除gouC与gouD均未成功,而本研究成功在异源表达菌株天蓝色链霉菌M1146中对gouC和gouD基因进行了遗传操作(阻断和互补),取得了较好的效果,为谷氏菌素生物合成基因簇中其他结构基因的功能研究提供了参考。
阻断次级代谢产物生物合成基因簇中特定结构基因,是产生新结构次级代谢产物的有效手段之一[23, 24]。本研究通过阻断结构基因gouC与gouD获得了谷氏菌素的新衍生物,而该方法同样可能适用于谷氏菌素的结构类似物。谷氏菌素的结构类似物包括宁南霉素和云南霉素:前者为谷氏菌素的手性异构体[25],被广泛运用于植物病害防治;后者是一种有效的抗肿瘤化合物[26],与谷氏菌素唯一差别在于核苷部分,云南霉素核苷部分的糖基为葡萄糖醛酸而谷氏菌素核苷部分的糖基为葡萄糖醛酰胺。这3种抗生素肽基部分的N末端残基均为肌氨酸,因此本研究对宁南霉素与云南霉素的理性改造和构效关系研究也具有指导意义。而且突变株发酵液中也有可能积累了其他新衍生物,例如在谷氏菌素发酵液中存在少量的云南霉素[6],后续工作可尝试从突变株发酵液中分离肌氨酸残基发生改变的云南霉素衍生物并分析其生物活性。
之前的研究已经初步阐释了谷氏菌素的生物合成途径[6],谷氏菌素肌氨酸残基的合成过程主要包括:甘氨酸首先通过GouK催化形成甘氨酰辅酶A,随后通过甲基转移酶GouN在甘氨酸氨基端引入甲基形成肌氨酰辅酶A,最后再连接到D-丝氨酸残基上形成终产物谷氏菌素[6]。但是从gouC与gouD突变株中分离得到了化合物C和D,其对应肌氨酸残基的位置分别为N-乙酰肌氨酸和N-乙酰甘氨酸。这表明在肽基部分的生物合成过程中,肌氨酸残基可能不是通过GouN直接甲基化生成的,而是需要多个酶进行甘氨酰辅酶A的乙酰化、N-乙酰甘氨酰辅酶A与D-丝氨酸残基的缩合、N-乙酰甘氨酸残基的甲基化和N-乙酰肌氨酸残基的去乙酰化等复杂的步骤。基于已有的结果,可以推测GouC参与了化合物C的去乙酰化,而GouD可能参与了将甲基转移到N-乙酰甘氨酸残基上的过程。本工作对谷氏菌素肌氨酸残基的形成提出了新的见解,但是GouC与GouD具体的催化机制还有待进一步体外生化实验的验证。综上所述,本工作为阐明谷氏菌素生物合成机制、通过遗传手段改造谷氏菌素获得新衍生物以及将谷氏菌素与其他核苷肽类抗生素进行组合生物合成奠定了基础。
致谢感谢中国科学院微生物研究所杜德尧和郭正彦博士在化合物分离纯化和结构解析方面给予的建议和帮助。
| [1] | Haneishi T, Arai M, Kitano N, Yamamoto S. Aspiculamycin, a new cytosine nucleoside antibiotic. 3. Biological activities, in vitro and in vivo. The Journal of Antibiotics, 1974, 27(5): 339-342. |
| [2] | Kondo F, Kitano N, Domon H, Arai M, Haneishi T. Aspiculamycin, a new cytosine nucleoside antibiotic. IV. Antimycoplasma activity of aspiculamycin in vitro and in vivo. The Journal of Antibiotics, 1974, 27(7): 529-534. |
| [3] | Lacal J, Vazquez D, Fernandez-Sousa J, Carrasco L. Antibiotics that specifically block translation in virus-infected cells. The Journal of Antibiotics, 1980, 33(4): 441-446. |
| [4] | Thiry L. The action of sparsomycin and gougerotin on virus growth. Journal of General Virology, 1968, 2(1): 143-153. |
| [5] | Kanzaki T, Toshihiko E, Yamamoto H, Shibata M, Nakazawa K, Iwasaki H, Takewaka T, Miyake A. Gougerotin, a new antibacterial antibiotic. The Journal of Antibiotics Ser. A, 1962, 15(2): 93-97. |
| [6] | Niu G, Li L, Wei J, Tan H. Cloning, heterologous expression, and characterization of the gene cluster required for gougerotin biosynthesis. Chemistry & Biology, 2013, 20(1): 34-44. |
| [7] | Clark J, Gunther J. Gougerotin, a specific inhibitor of protein synthesis. Biochimica et Biophysica Acta, 1963, 76(4): 636-638. |
| [8] | Cerna J, Lichtenthaler F, Rychlik I. The effect of gougerotin analogues on ribosomal peptidyl transferase. FEBS Letters, 1971, 14(1): 45-48. |
| [9] | Wei J, Tian Y, Niu G, Tan H. GouR, a TetR family transcriptional regulator, coordinates the biosynthesis and export of gougerotin in Streptomyces graminearus. Applied and Environmental Microbiology, 2014, 80(2): 714-722. |
| [10] | Niu G, Tan H. Nucleoside antibiotics: biosynthesis, regulation, and biotechnology. Trends in Microbiology, 2015, 23(2): 110-119. |
| [11] | Gomez-Escribano J, Bibb M. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology, 2011, 4(2): 207-215. |
| [12] | Kieser T, Bibb M, Buttner M, Chater K, Hopwood D. Practical Streptomyces Genetics. John Innes Foundation, Norwich, 2000. |
| [13] | Sambrook J, Russell D. Molecular Cloning: A Laboratory Manual. Cold Spring Harbor Laboratory Press: Cold Spring Harbor, New York, 2001. |
| [14] | Gregory M, Till R, Smith M. Integration site for Streptomyces phage fBT1 and development of site-specific integrating vectors. Journal of Bacteriology, 2003, 185(17): 5320-5323. |
| [15] | Jiang L, Wei J, Li L, Niu G, Tan H. Combined gene cluster engineering and precursor feeding to improve gougerotin production in Streptomyces graminearus. Applied Microbiology and Biotechnology, 2013, 97(24): 10469-10477. |
| [16] | Gust B, Kieser T, Chater K. REDIRECT technology: PCR-targeting system in Streptomyces coelicolor A3(2). John Innes Foundation: Norwich, 2002. |
| [17] | Wang J, Ma S, Chen X. Anti-tumor effects of AL3810 on A549 human lung adenocarcinoma. World Clinical Drugs, 2013, 34(4): 206-211. |
| [18] | Adamski R, Heymann H, Geftic S, Barkulis S. Preparation and antibacterial activity of some spiramycin derivatives. Journal of Medicinal Chemistry, 1966, 9(6): 932-934. |
| [19] | Benveniste R, Davies J. Mechanisms of antibiotic resistance in bacteria. Annual Review Biochemistry, 1973, 42: 471-506. |
| [20] | Xue Y, Wu S, Li Y, Zhang S, Zhen Y. Antitumor activity of yungumycin. Acta Pharmaceutica Sinica, 1996, 31(3): 171-175. |
| [21] | Hu Y, Phelan V, Farnet C, Zazopoulos E, Bachmann B. Reassembly of anthramycin biosynthetic gene cluster by using recombinogenic cassettes. Chembiochem, 2008, 9(10): 1603-1608. |
| [22] | Du D, Zhu Y, Wei J, Tian Y, Niu G, Tan H. Improvement of gougerotin and nikkomycin production by engineering their biosynthetic gene clusters. Applied Microbiology and Biotechnology, 2013, 97(14): 6383-6396. |
| [23] | Bormann C, Lauer B, Kalmanczhelyi A, Sussmuth R, Jung G. Novel nikkomycins Lx and Lz produced by genetically engineered Streptomyces tendae Tu901. The Journal of Antibiotics, 1999, 52(6): 582-585. |
| [24] | Mendes M, Recio E, Fouces R, Luiten R, Martin J, Aparicio J. Engineered biosynthesis of novel polyenes: a pimaricin derivative produced by targeted gene disruption in Streptomyces natalensis. Chemistry & Biology, 2001, 8(7): 635-644. |
| [25] | Xiang G, Hu H, Chen J, Chen W, Wu L. A new agricultural antibiotic-ningnanmycin. Acta Microbiologica Sinica, 1995, 35(5): 368-374. |
| [26] | Chen W, Cheng C, Pan J. Yunnanmycin, a novel antitumor antibiotic. II. Extraction, purification, physico-chemical properties and identification. Chinese Journal of Antibiotics, 1998, 23(3): 170-174. |
 2016, Vol. 56
2016, Vol. 56